乳酸菌中移動基因元件的預測與分析
(東北農(nóng)業(yè)大學,乳品科學教育部重點實驗室,哈爾濱150030)
乳酸菌中移動基因元件的預測與分析
李婉,于上富,丁秀云,杜金城,徐敏,霍貴成
(東北農(nóng)業(yè)大學,乳品科學教育部重點實驗室,哈爾濱150030)
CR ISPR-Cas系統(tǒng)作為獲得性免疫系統(tǒng)能夠保護乳酸菌免受噬菌體等外源基因元件的入侵,旨在對乳酸菌中CR ISPR-Cas系統(tǒng)的活性與近期發(fā)生水平基因轉(zhuǎn)移的程度是否呈負相關(guān)進行檢驗。預測了乳酸菌中的CR ISPR-Cas系統(tǒng)、前噬菌體及基因島,并分別分析乳酸菌中CR ISPR與前噬菌體及與基因島在數(shù)量上的相關(guān)性。研究發(fā)現(xiàn)CRISPR序列的長度(能反映該免疫系統(tǒng)的活性)與預測的近期發(fā)生的水平基因轉(zhuǎn)移事件的數(shù)目之間沒有明顯的相關(guān)性。結(jié)果表明乳酸菌中的CRISPR-Cas系統(tǒng)并不抑制水平基因轉(zhuǎn)移事件的發(fā)生。
乳酸菌,移動基因元件,CRISPR-Cas系統(tǒng),水平基因轉(zhuǎn)移,間隔序列
0 引言
CR ISPR(clustered regu larly interspaced short pal?indro mic repeats)-Cas(CR ISPR-associated genes)系統(tǒng)能夠為細菌和古細菌提供可獲得、可遺傳的免疫,使其免受病毒[1,2]和質(zhì)粒[3-4]的侵染。CR ISPR-Cas系統(tǒng)在絕大多數(shù)的古細菌及大多數(shù)細菌中普遍存在[5,6],這使得CR ISPR-Cas系統(tǒng)成為近年來學者研究的焦點。CR ISPR-Cas基因座包含一系列短的、部分回文的重復序列,這些重復序列又被間隔序列穿插[7]。
水平基因轉(zhuǎn)移(ho rizontal gene transfer,HGT),是指在差異生物個體之間,或單個細胞內(nèi)部細胞器之間所進行的遺傳物質(zhì)的交流。水平基因轉(zhuǎn)移能使原核生物獲得新的而分子功能,并提供選擇性的進化優(yōu)勢。有研究指出CR ISPR-Cas系統(tǒng)活性可能會造成水平基因轉(zhuǎn)移縮減,阻止宿主從細菌中獲得新基因[8-9]。因此,驗證乳酸菌中CR ISPR-Cas系統(tǒng)是否抑制水平基因轉(zhuǎn)移對于后期CR ISPR-Cas系統(tǒng)的應用具有重要意義。
1 實驗
1.1 材料
本研究所包含的菌株有NCBI上已測序的9株嗜熱鏈球菌、4株徳氏乳桿菌保加利亞亞種、7株瑞士乳桿菌、5株乳酸乳球菌、7株植物乳桿菌及3株嗜酸乳桿菌,共35株乳酸菌。
1.2 CR ISPR-Cas系統(tǒng)活性的預測
在NCBI上查找并下載已測序的植物乳桿菌、嗜酸乳桿菌及乳酸乳球菌的全基因組序列,利用CR IS?PR Finder[10](http://crispr.u-psud.fr/Server/)對CR IS?PR序列進行查找,統(tǒng)計各CR ISPR所含間隔序列的數(shù)目。
1.3 前噬菌體預測
利用PHAST(http://phast.w ishartlab.com/)網(wǎng)站在線預測乳酸菌基因組中所含前噬菌體,統(tǒng)計各乳酸菌中所含前噬菌體的數(shù)目及噬菌體蛋白的數(shù)目。
1.4 基因島預測
利用IslandView er 3[11](http://w ww.pathogenom?ics.sfu.ca/islandviewer/)對實驗菌株的基因島進行預測,推定的基因島需大于8個基因或者大于8 kb。統(tǒng)計基因島的數(shù)目并計算基因島中所含基因占基因組的百分比。

表1 課題使用乳酸菌信息
1.5 相關(guān)性分析
利用O rigin 9軟件制作條形圖,分別分析乳酸菌中CR ISPR與前噬菌體及與基因島在數(shù)量上的相關(guān)性。
2 結(jié)果和分析
2.1 乳酸菌中CR ISPR序列概況
利用CR ISPR Finder預測得到乳酸菌中所含CR ISPR序列的情況如表2所示。表2中,僅確定的CR ISPR序列被列出,可疑的CR ISPR序列未列出,不含CR ISPR序列的菌株未列出。9株嗜熱鏈球菌及4株德氏乳桿菌均含CR ISPR序列,5株乳酸乳球菌均不含CR ISPR序列。
2.2 乳酸菌中前噬菌體概況
經(jīng)PHAST預測得到乳酸菌中前噬菌體情況如表3所示。表3中,不含前噬菌體的菌株未列出。其中,徳氏乳桿菌僅ND 02含有1個前噬菌體,其余3株均不含前噬菌體,瑞士乳桿菌除H 9和DPC 4571外均含不同數(shù)目的前噬菌體。
35株乳酸菌中,13株無間隔序列的乳酸菌所含前噬菌體蛋白的平均值為156,1株含6個間隔序列的乳酸菌所含前噬菌體蛋白數(shù)目為182,2株間隔序列數(shù)目在7到21之間的乳酸菌其平均前噬菌體蛋白數(shù)目為19個,有12株乳酸菌的間隔序列數(shù)目在22個到42個之間,它們的前噬菌體蛋白數(shù)目平均為45個,有7株乳酸菌的間隔序列數(shù)目在43個到76個之間,它們的前噬菌體蛋白數(shù)目平均為20個。由此可見,基因組中前噬菌體蛋白數(shù)目與間隔序列數(shù)目并不呈負相關(guān)。
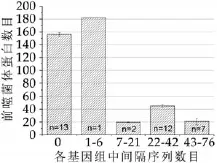
圖1 前噬菌體蛋白與間隔序列之間的數(shù)量關(guān)系
2.3 基因島預測
利用IslandViewer 3預測得到乳酸菌中所含的基因島,其中大于8個基因或大于8 kb的基因島被統(tǒng)計,每個基因組中所含基因島的大小占基因組的比例如表4所示。
基因組中基因島所占比例與間隔序列數(shù)目之間的關(guān)系如圖2,13株不含間隔序列的乳酸菌其基因組中基因島所占比例的平均值為1.03%,間隔序列數(shù)目在1到6個之間的乳酸菌其基因島所占比例為0.33%,間隔序列數(shù)目在7到21個之間的乳酸菌其基因島所占比例為1.51%,間隔序列數(shù)目在22到42個之間的乳酸菌其基因島所占比例為1.19%,而間隔序列數(shù)目在43到76個之間的乳酸菌其基因島所占比例為1.76%。基因組中基因島占基因組的比例與間隔序列數(shù)目亦不呈負相關(guān)。
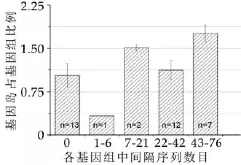
圖2 基因島占基因組的比例與間隔序列數(shù)目之間的關(guān)系
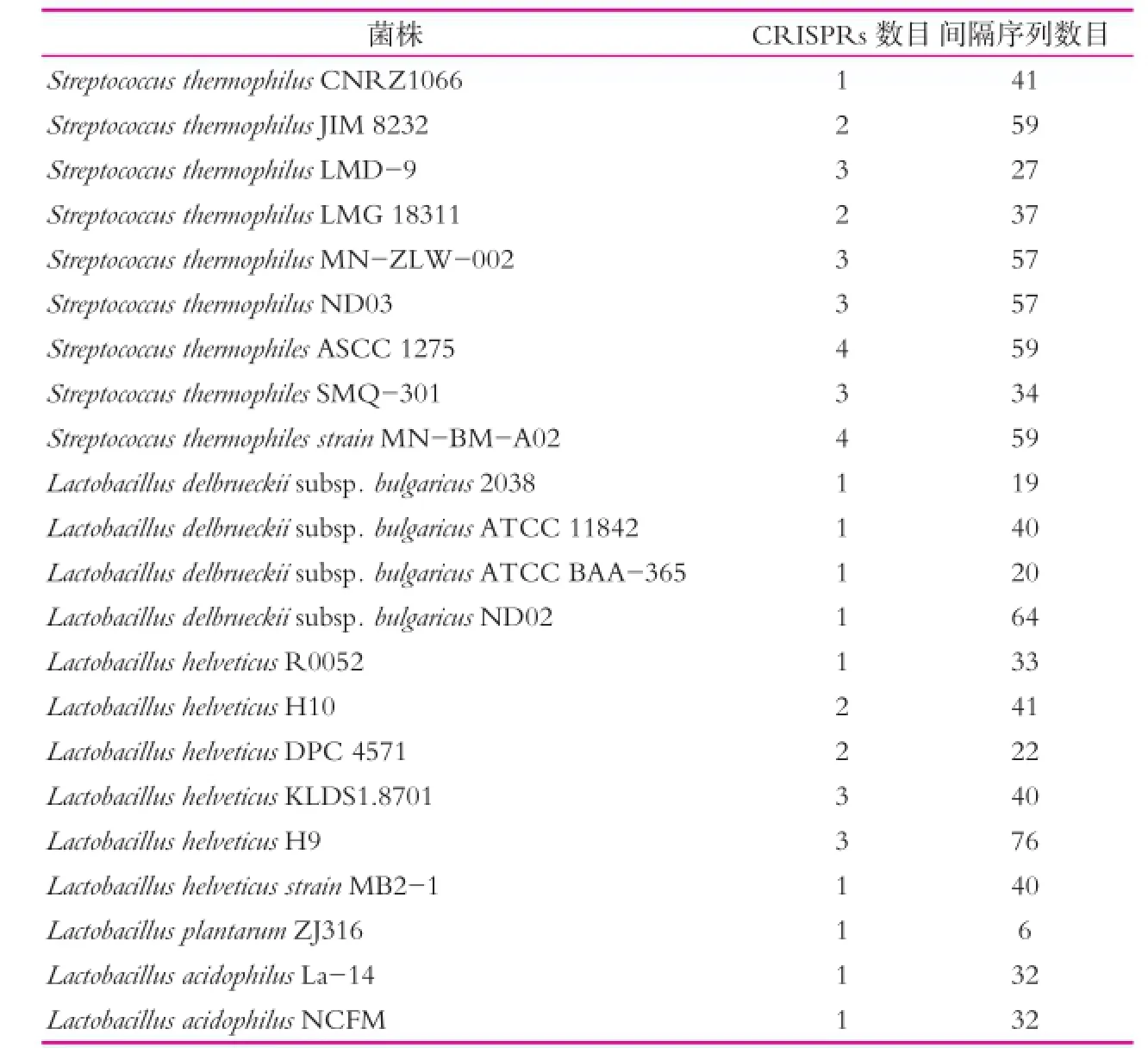
表2 乳酸菌中CR ISPR序列概況

表3 乳酸菌中前噬菌體概況
3 討 論
移動基因元件如前噬菌體、基因島等能通過水平基因轉(zhuǎn)移機制被細菌基因組獲得或失去。比較基因組學研究表明移動基因元件的水平基因轉(zhuǎn)移是一個主要的進化步驟,能夠增加基因和表型的多樣性,使得細菌能夠適應多變的環(huán)境。然而,有研究表明在葡萄球菌中CR ISPR干擾限制了水平基因轉(zhuǎn)移,并因此限制了抗生素抗性在致病菌中的傳播[3]。的確,臨床分離的多藥耐藥性腸球菌與敏感菌相比缺乏CR IS?PR-Cas系統(tǒng),這可能是由于敏感菌所含的CR ISPR-Cas系統(tǒng)阻礙了抗性質(zhì)粒的獲得[12]。因此看來,在活躍的獲得性免疫與通過水平基因轉(zhuǎn)移獲得新基因或者新功能之間有一個權(quán)衡。
目前,利用間隔序列數(shù)目來反映CR ISPR系統(tǒng)活性被認為是最佳的計算方法。現(xiàn)已證明一個有活性的CR ISPR-Cas系統(tǒng)能夠連續(xù)獲得間隔序列,使得CR IS?PR序列不斷增長[19]。相反,在缺乏選擇壓力的情況下,為了維持CR ISPR-Cas系統(tǒng)的活性,間隔序列則會被快速刪除[7,13,14]。因此,無活性的CR ISPR序列應趨向于縮短,而有活性的CR ISPR序列則將擴大或者至少保持一個相對穩(wěn)定的大小。此外,盡管一個CR IS?PR-Cas系統(tǒng)近期在某個基因組中沒有活性,但該基因組仍攜帶一個大的CR ISPR序列,這似乎更適合觀察CR ISPR-Cas系統(tǒng)影響其近期的進化歷史。
由于CR ISPR-Cas系統(tǒng)能夠阻止質(zhì)粒接合、前噬菌體整合以及裸DNA的轉(zhuǎn)化,因此該免疫系統(tǒng)被認為是阻礙水平基因轉(zhuǎn)移的柵欄[15]。觀察人類的病原菌[12]并結(jié)合實驗和模型[16],證實了當宿主在選擇壓力下獲得基因(當暴露在抗生素環(huán)境下時,獲得抗性便是存活或復制的關(guān)鍵)時,CR IS?PR-Cas系統(tǒng)往往是失活的或者丟失的。然而,在有效地抵抗外源基因元件與無法獲得新基因之間存在一個權(quán)衡。本研究檢驗了水平基因轉(zhuǎn)移與CR ISPR-Cas系統(tǒng)活性之間的關(guān)系,結(jié)果并不支持權(quán)衡假說。事實上,細菌中CR ISPR-Cas系統(tǒng)的活性與水平轉(zhuǎn)移基因的比例并不呈負相關(guān)。
本研究中CR ISPR-Cas系統(tǒng)的活性與通過水平轉(zhuǎn)移基因獲得的基因之間不存在負相關(guān),這歸因于一些不相互排斥的進化機制。最簡單的解釋是CR ISPR-Cas系統(tǒng)經(jīng)常自發(fā)地移動,因此它們在任何基因組中的存在與否并不能表明其仍具有長期的影響。其次,在某些環(huán)境下,當微生物面臨較高的移動基因元件入侵的風險時,CR ISPR-Cas系統(tǒng)的作用是微不足道的[17]。因此,大的CR ISPR序列表明基因組積極地整合新的間隔序列,并且由于其遭受一連串的外源DNA元件的入侵,CR ISPR序列需要選擇性地維持自身活性。此外,近期研究表明一些CR ISPR-Cas系統(tǒng)需要轉(zhuǎn)錄外源DNA序列用于干擾,以允許溶源作用,防止烈性感染[18]。第三,細菌和古細菌中間隔序列的獲得并非隨機的,而是優(yōu)先從含有匹配已經(jīng)存在的間隔序列的基因組中獲得。

表4 乳酸菌中基因島概況
本研究檢驗了CR ISPR-Cas系統(tǒng)活性與水平基因轉(zhuǎn)移事件之間的關(guān)系,結(jié)果乳酸菌中CR ISPR-Cas系統(tǒng)的活性與所含前噬菌體蛋白數(shù)目及基因島所占基因組的比例均不呈負相關(guān),證明了CR ISPR-Cas系統(tǒng)并不抑制前噬菌體、基因島等移動基因元件的水平轉(zhuǎn)移,表明在進化的時間尺度上CR ISPR-Cas系統(tǒng)對水平基因轉(zhuǎn)移事件沒有抑制作用。
[1]BARRANGOU R,FREMAUX C,DEVEAU H,et al.CR ISPR provides acquired resistance against viruses in prokaryotes[J].Science, 2007,315(5819):1709-1712.
[2]MAN ICA A,ZEBEC Z,TEICHMANN D,et al.In vivo activity of CRISPR-mediated virus defence in a hyperthermophilic archaeon[J]. Molecular Microbiology,2011,80(2):481-491.
[3]MARRAFFIN I L,SONTHEIMER E.CR ISPR interference li mits horizontal gene transfer in staphylococci by targeting DNA[J].Sci?ence,2008,322(5909):1843-1845.
[4]SOLEY G,LING D,ZHENGJUN C,et al.Dynamic properties of the Sulfolobus CR ISPR/Casand CRISPR/C mr systemswhen chal?lenged w ith vector-borne viraland plas mid genesand protospacers[J]. Molecular Microbiology,2011,79(1):35-49.
[5]ANDERSON R,BRAZELTON W,BAROSS J.Using CR ISPRsas ametageno mic tool to identifymicrobialhostsof a diffuse flow hydro?thermalvent viralassemblage[J].FemsMicrobiology Ecology,2011,77 (1):120-133.
[6]W EINBERGER A,WOLF Y,LOBKOVSKY A,et al.Viral diversity threshold for adaptive immunity in prokaryotes[J].Mbio,2012,3(6): 17.
[7]HORVATH P,ROMERO D,CO?T?-MONVO ISIN A,etal.Di?versity,activity,and evolution of CR ISPR loci in streptococcus ther?mophilus[J].Journalof bacteriology,2008,190(4):1401-1412.
[8]MARRAFFIN I L.CR ISPR-Cas immunity against phages:its effects on the evolution and survivalof bacterial pathogens[J].Plos Pathogens, 2013,9(12):1831-1836.
[9]HATOUM-ASLAN A,MARRAFFIN IL.I mpact of CRISPR im?munity on the emergence and virulence of bacterial pathogens[J]. CurrentO pinion in Microbiology,2014,17(3):82-90.
[10]GR ISSA I,VERGNAUD G,POURCEL C.CR ISPRFinder:aweb tool to identify clustered regularly interspaced short palindromic re?peats[J].Nucleic acids research,2007,35(suppl2):W 52-W 57.
[11]DH ILLON B,LAIRD M,SHAY J,etal.IslandViewer3:more flexi?ble,interactive genomic island discovery,visualization and analysis[J]. Nucleic AcidsR esearch,2015,43(W 1):104-108.
[12]PALMER K,GILMORE M.Multidrug-resistant enterococci lack CRISPR-cas[J].Mbio,2010,1(4):516-524.
[13]TYSON G,BANFIELD J.Rapidly evolving CRISPRsimplicated in acquired resistance of microorganisms to viruses[J].Environmentalmi?crobiology,2008,10(1):200-207.
[14]DEVEAU H,BARRANGOU R,GARNEAU J,et al.Phage re?sponse to CRISPR-encoded resistance in streptococcus thermophilus [J].Journalofbacteriology,2008,190(4):1390-1400.
[15]ROTEME,UDIQ.The Escherichia coli CRISPR syste mprotects fromλ lysogenization,lysogens,and prophage induction[J].Journal of Bacteriology,2010,192(23):6291-6294.
[16]WENYAN J,INBALM,FAWAZ A,et al.Dealingw ith the evolu?tionary downside of CR ISPR immunity:bacteria and beneficial plas?mids[J].PlosGenetics,2013,9(9):119-129.
[17]GOPHNA U,KRISTENSEN D,WOLF Y,et al.No evidence of inhibition ofhorizontalgene transfer by CR ISPR-Cason evolution?ary ti mescales[J].Isme Journal,2015,9(9).
[18]GOLDBERG G,WENYAN J,DAVID B,et al.Conditional toler?ance of temperate phages via transcription-dependent CR ISPR-Cas targeting[J].Nature,2014,514(7524):633-637.
Prediction and analysis of mobile genetic ele ments in Lacticacid bacteria
LIW an,YU Shangfu,DING Xiuyun,DU Jincheng,XU Min,HUO Guicheng
(Key Laborato ry o f Dairy Science,Ministry o f Education,No rtheast Agricu ltu ral University,H arbin 150030,China)
W e aimed to test thatwhether the activity of CRISPR–Cas in microbes isnegatively correlated w ith the extent of recent HGT or not.W e predict the CRISPR–Cassystems,prophagesand geno mic islands to analysis the correlations between CRISPR spacer count and HGT measures.We found no significant dependence between the length of CRISPR arrays,which reflects the activity of the immune system,and the estimated number of recent HGT events.No evidence of inhibition of horizontal gene transfer by CRISPR–Cason evolu?tionary timescales.
Lacticacid bacteria;mobilegenetic elements;CRISPR-Cassystem;horizontalgene transfer;spacers
Q 933
A
1001-2230(2017)01-0004-04
2016-06-12
國家自然科學基金(31171717);國家“863計劃”(2012AA022 108)。
李婉(1990–),女,碩士研究生,研究方向為乳酸菌生物技術(shù)。
霍貴成

