動態-靜態混合的時序蛋白質網絡構建方法
代啟國, 郭茂祖, 劉曉燕, 王春宇
(1.哈爾濱工業大學 計算機科學與技術學院, 哈爾濱 150001; 2.大連民族大學 計算機科學與工程學院, 遼寧 大連 116600;3.大連市民族文化數字化重點實驗室(大連民族大學), 遼寧 大連116600)
?
動態-靜態混合的時序蛋白質網絡構建方法
代啟國1,2,3, 郭茂祖1, 劉曉燕1, 王春宇1
(1.哈爾濱工業大學 計算機科學與技術學院, 哈爾濱 150001; 2.大連民族大學 計算機科學與工程學院, 遼寧 大連 116600;3.大連市民族文化數字化重點實驗室(大連民族大學), 遼寧 大連116600)
目前已公開的蛋白質網絡多為靜態網絡,不能有效描述細胞中蛋白質的動態活動特點. 通過融合基因表達數據,研究人員可以構建出描述蛋白質動態性的時序蛋白質網絡. 現有方法假設所有蛋白質都是動態變化的,而事實上除動態蛋白質外細胞中還包含相對穩定的靜態蛋白質. 為此,提出了一種基于動態-靜態蛋白質混合的時序網絡構建新方法. 該方法根據基因表達變化情況將蛋白質分為動態和靜態兩類,并在構建各時刻網絡時考慮動態與靜態蛋白質之間的相互作用關系. 實驗結果表明,利用本文方法構建的時序蛋白質網絡可以提高蛋白質復合體識別的準確性,從而驗證了本文方法的可行性.
時序蛋白質網絡;蛋白質相互作用;基因表達;生物網絡;蛋白質復合體識別
蛋白質相互作用網絡(protein-protein interaction network,簡稱蛋白質網絡)是描述細胞中蛋白質活動的一種生物網絡[1-2]. 由于蛋白質在生命活動中具有十分重要的作用,因而基于蛋白質網絡的分析和挖掘方法越來越受到關注. 蛋白質復合體識別[3-5]、關鍵蛋白質識別[6]等很多生物信息研究都是以蛋白質網絡為基礎的.
現有公開蛋白質網絡通常可表示為一個圖,其中節點表示蛋白質,邊表示蛋白質之間的相互作用關系. 這種網絡一般假設蛋白質的豐度都是靜態不變的,所描述的蛋白質之間相互作用也是持續穩定的,因而稱為“靜態蛋白質網絡”. 然而,真實細胞系統中蛋白質及其相互作用是隨時變化的,具有一定的動態性. Lichtenberg等人研究發現,大部分復合體都是由動態和靜態兩類蛋白質構成[7]. 然而,靜態蛋白質網絡的簡單圖結構無法描述蛋白質動態特性[8-10],因此有必要構建能夠描述蛋白質動態特性的時序蛋白質網絡模型.
在構建時序蛋白質網絡的過程中,一個關鍵問題是如何確定蛋白質在何時處于激活狀態. 采用單一固定閾值方法確定蛋白質的活動狀態與非活動狀態雖然簡單[8],但存在一定局限. 例如,一些蛋白質的含量在整個細胞周期都較低. 如果采用單一的固定閾值,那么這些蛋白質可能會被認為是非激活狀態而被忽略掉. Hegde等人提出了利用不同基因表達數據中表達值的均值確定蛋白質的活動狀態[10]. 王建新等人[11]針對蛋白質不同的表達模式特性,提出了3-σ閾值計算方法. 上述方法假設所有蛋白質豐度都是動態變化. 然而,生物學研究表明,細胞中蛋白質并非全部都是動態的,有些蛋白質的豐度受細胞周期、環境等影響較小,表現出一定的靜態特性[12]. 因此,將所有蛋白質均視為動態的方法,不能準確地刻畫細胞中真實的蛋白質活動規律.
為此,本文提出一種動態-靜態蛋白質混合的時序蛋白質網絡構建方法(A hybrid temporal protein network,HTPIN). 該方法首先將蛋白質劃分為動態和靜態兩類,然后同時利用動態蛋白質與靜態蛋白質之間相互作用關系構建時序網絡. 通過相關實驗測試與分析,利用本文方法所構建的時序蛋白質網絡具有很好的可靠性,可以提高蛋白質復合體識別的準確性.
1 動態-靜態混合的時序網絡構建方法
給出一種基于動態-靜態蛋白質混合的時序蛋白質網絡構建方法(HTPIN). 首先,以基因表達數據為基礎,利用蛋白質編碼基因在不同時刻表達值變化的波動性,將蛋白質劃分為動態和靜態兩類;然后,采用3-σ閾值[11]判斷動態蛋白質激活時刻;最后,構建各時刻蛋白質網絡. 該方法的基本流程如圖1所示.
1.1 動態與靜態蛋白質
以基因表達量作為判定蛋白質動、靜態屬性的依據. 假設給定蛋白質網絡中有N個蛋白質,基因表達數據中記錄了T個時間點的表達量. 對于蛋白質i(1≤i≤N),其對應編碼基因在所有時刻的表達值Expi可表示為
其中,eit表示蛋白質i的編碼基因在t時刻的表達量. 則蛋白質i表達量的方差[12]為
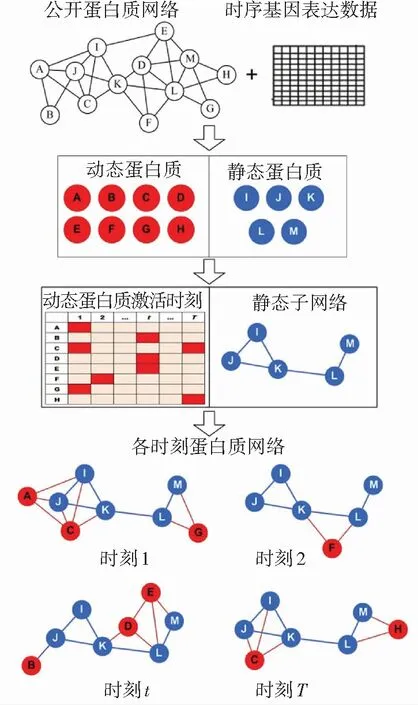
圖1 HTPIN方法構建時序蛋白質網絡過程示意
Fig.1 Illustration of construction process of temporal protein networks with HTPIN
利用表達量波動值EVi,表示蛋白質i在所有時刻表達量的方差值. 具體來講,表達量方差值高于蛋白質i表達量方差的蛋白質數目記為ni,則蛋白質i編碼基因表達量波動值EVi=ni/N. 該值介于0和1之間,越接近于0表明蛋白質表達量越穩定;越接近于1則表示蛋白質表達量波動性越大.
引入波動性閾值thDyn(0≤thDyn≤1)確定每個蛋白質的動、靜態屬性. 蛋白質i的波動值EVi大于或等于閾值thDyn,則認為該蛋白質是動態蛋白質,否則為靜態蛋白質,具體可形式化為
其中isDyn(i) 值為1表示蛋白質i是動態蛋白質,為0表示蛋白質是靜態蛋白質. 動態閾值thDyn為HTPIN算法參數.
1.2 確定蛋白質激活時刻
1.2.1 動態蛋白質的激活狀態
動態變化的蛋白質只有在其處于激活狀態時,才與其他蛋白質發生相互作用. 因此,需要確定動態蛋白質處于激活狀態的時間點. 動態蛋白質在不同時間點對應表達量具有很大的波動性. 對于某一時刻,如果一個動態蛋白質的表達量相對較高,則認為其處于活動狀態;而表達量相對較低,則認為是非活動狀態. 為此,引入激活閾值thiAct來判斷動態蛋白質在各時間點所處的狀態. 對于動態蛋白質i(isDyn(i)=1),如果其在時間點t對應基因的表達值eit高于該激活閾值thiAct,則認為其在t時刻為激活狀態,否則為未激活狀態,即
其中,isActive(i, t)值為1表示激活,0表示未激活. 采用3-σ方法[11]確定激活閾值thiAct,其具體方法為
1.2.2 靜態蛋白質的激活狀態
靜態蛋白質對應的基因表達量相對穩定,因而通常認為它們在整個生物過程中活動狀態是持續穩定的[7, 12]. 因此,靜態蛋白質在所有時刻均處于激活狀態.
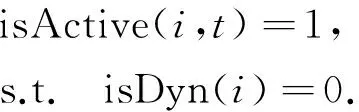
1.3 構建時序蛋白質網絡
基于動態-靜態蛋白質混合的時序網絡構建方法主要分為兩個步驟:首先,抽取靜態蛋白質之間相互作用網絡,即靜態子網絡;然后,以其為基礎在每個時間點增加在該時刻處于激活狀態的動態蛋白質及其相互作用關系.
1.3.1 輸入數據描述
輸入數據包括公開蛋白質網絡G和某個生物過程中的時序基因表達數據Exp. 其中,網絡G可描述為G=(V, E),V表示其中蛋白質集合,E表示蛋白質相互作用關系集合. 網絡中的蛋白質數目記為N(N=|V|). 時序基因表達數據Exp中收錄了所有N個蛋白質在T個時刻下對應基因的表達量,即

1.3.2 抽取靜態子網絡
抽取靜態子網絡是指獲取輸入蛋白質網絡G中所有靜態蛋白質構成的子網絡. 通過預先抽取靜態子網絡可以避免在各時刻重復構建,提高構建時序網絡的效率. 所抽取的靜態子網絡可表示為GStat=(VStat, EStat),其中包含所有靜態蛋白質VStat和它們在G中的相互作用關系EStat.
1.3.3 構建各時刻動態-靜態混合網絡
時序蛋白質網絡GTC可表示為
(8)
其中,Gt為t時刻蛋白質網絡,表示為Gt=(Vt,Et),Vt和Et分別構成該時刻網絡的蛋白質和相互作用關系集合. 由于已經預提取了每個時刻靜態子網絡,因此構建各時刻網絡可以轉化為在靜態子網絡基礎上,增加在該時刻處于激活狀態的動態蛋白質以及與它們之間的相互作用關系;此外,還增加它們與靜態蛋白質之間的作用關系.
1.3.4 動態-靜態混合時序蛋白質網絡構建算法
以上述主要過程為基礎,基于基因表達數據的動態-靜態混合時序蛋白質網絡的構建算法(HTPIN)的描述如下.
輸入:蛋白質相互作用網絡G=(V,E);基因表達數據Exp={Exp1,…,Exp|V|};動態閾值thDyn.
輸出:混合型時序蛋白質網絡GTC.
初始化靜態子網絡GStat=(VStat,EStat),VStat=?,EStat=?;
初始化動態蛋白質集合VDyn=?;
for each p in V/* 將蛋白質分為動態和靜態兩類蛋白質 */
if p為動態蛋白質isDyn(p,thDyn)
將p加入到動態蛋白質集合VDyn=VDyn∪{p};
else
將p加入到靜態蛋白質集合VStat=VStat∪{p};
end if
end for
for each epqin E
if both p and q in Estat
將epq加入到EStat;
end if
end for
初始化時序網絡GTC={G1,…,GT}
for t=1 to T/* 獲取各時刻蛋白質網絡 */
初始化t時刻網絡Gt=(Vt,Et),Vt=?,Et=?;
for each p in VDyn/* 獲取t時處于激活狀態的動態蛋白質 */
if 蛋白質p在t時刻處于激活狀態isActive(p,t)
Vt=Vt∪{p};
end if
end for
for each p in Vt/* 獲取激活動態蛋白質之間的相互作用 */
for each q≠p in Vt
if 輸入網絡G中存在p和q的相互作用,即epqin E
將epq加入到Et;
end if
end for
end for
for each p in Vt/* 激活動態蛋白質與靜態蛋白質相互作用*/
for each q in VStat
if 輸入網絡G中存在p和q的相互作用,即epqin E
將epq加入到Et;
end if
end for
end for
Vt=Vt∪VStat;
end for
輸出時序蛋白質網絡GTC
2 實驗結果
為了驗證本文HTPIN方法的合理性,對其進行多方面的測試. 基于蛋白質網絡識別復合體是針對蛋白質網絡進行信息挖掘的典型問題. 蛋白質網絡是計算識別復合體的重要數據依據. 蛋白質復合體是由多個蛋白質通過物理相互作用結合形成,這些相互作用緊密的蛋白質一般在蛋白質網絡中對應稠密的子圖結構[1-2,13]. 因而,可以通過挖掘網絡中稠密子圖識別蛋白質復合體. 也就是說,從蛋白質網絡中識別復合體是一個圖聚類問題. 因此,本文通過分析所構建的時序蛋白質網絡是否可以提高復合體識別準確性來評價HTPIN方法的有效性.
2.1 實驗設計及實驗數據
為了達到通過識別復合體驗證時序網絡的目的,在實驗中采用了如下方案:對于給定的一種復合體識別算法和構建的時序網絡GTC={G1,G2,…,Gt,…,GT},首先將識別算法依次在每個時刻的蛋白質網絡Gt上運行,獲得在t時刻的識別結果Ct;然后將在所有時刻網絡上的識別結果集合在一起,去除重復復合體,形成復合體集合C,即為該識別算法在時序網絡GTC上最終識別結果. 通過分析識別結果C與已知復合體集合(CYC2008)[14]匹配的組合評分,以及功能一致性兩方面評價時序網絡. 組合評分具體為F-Measure、ACC和MMR三項子指標之和[15]. 為了更加客觀地進行評價,避免個別算法對時序網絡的偏好性,在實驗中測試了CPL[16]、ClusterONE[17]和PLSMC[15]3種不同類型算法.
在基因表達數據方面,采用了GEO數據庫中與酵母代謝周期相關的GSE3431系列數據[18]. 該數據采集背景是限制營養條件下連續培養酵母細胞的3個代謝周期,每個周期采樣12個時間點,間隔為25 min.
在靜態蛋白質網絡方面,采用了酵母的BioGRID蛋白質網絡[19]. 在兩種類型數據之間,采用ORF命名類型來作為關鍵字以建立兩種數據中的基因與蛋白質之間的對應關系. 濾掉兩種數據中未建立映射關系的基因或蛋白質.
2.2 波動性閾值的影響
HTPIN時序網絡構建方法的核心是動態-靜態蛋白質混合的思想. 為了驗證這種方案的合理性,本文研究了控制靜態與動態蛋白質混合比例的波動性閾值thDyn對所構建網絡的影響. 測試了該閾值thDyn={0.0,0.1,…,0.9,10}的不同取值情況. 對于每個取值,統計分析不同算法在時序網絡上的復合體識別結果.
圖2描述了CPL、ClusterONE和PLSMC三種算法在采用不同波動性閾值而構建的時序網絡上識別復合體結果的對比情況. 當thDyn=0時表明所有蛋白質均被視為動態蛋白質,并認為這些蛋白質只在部分時刻處于活動狀態;而當thDyn=1時則表示所有蛋白質都被視為靜態蛋白質,在所有時刻均出現.
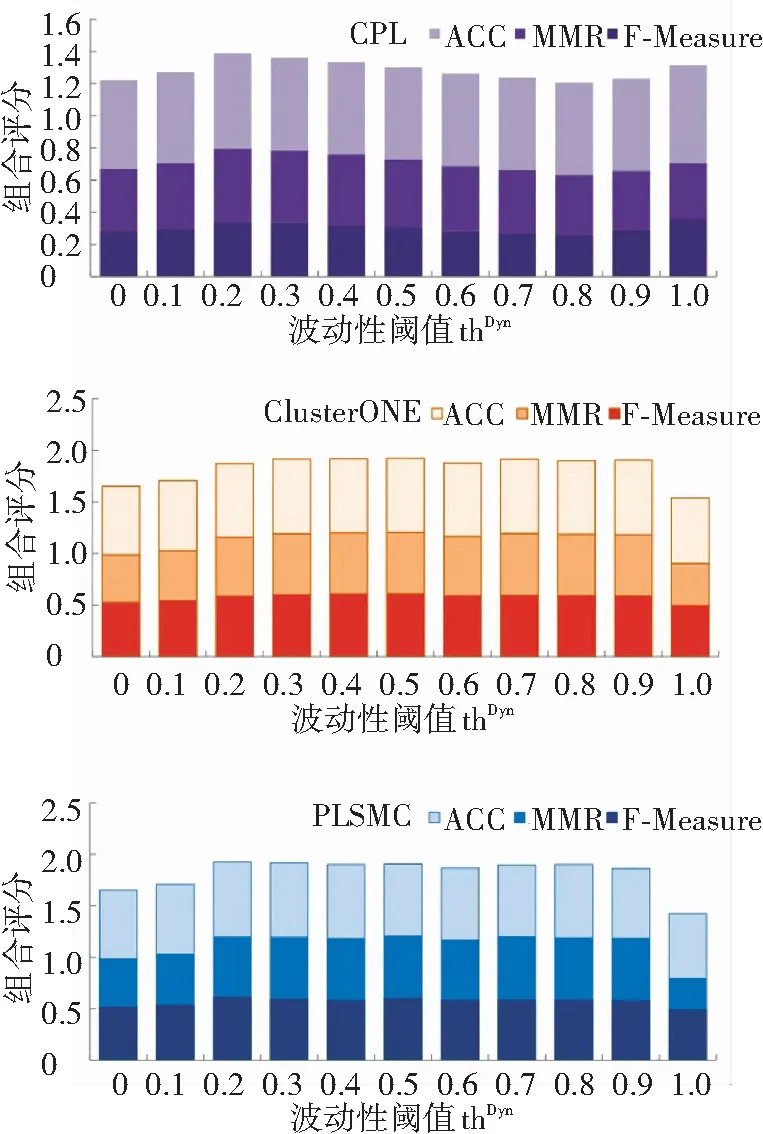
圖2 采用不同波動性閾值thDyn構建時序網絡對復合體識別性能的影響
Fig.2 The performance comparison of complex detection on time-course protein network constructed by HTPIN with different volatility threshold thDyn
從圖2可見,不同算法識別結果的組合評分均隨波動性閾值thDyn的改變而較大發生變化. 但所有參與測試的算法識別結果的組合評分峰值均出現在當thDyn=0.2至0.4之間,即此時復合體識別結果要好于thDyn=0和thDyn=1兩種極端情況. 由此表明,在構建時序蛋白質網絡時,將動態蛋白質與靜態蛋白質控制在合理的比例可以有效提高復合體識別的總體性能. 上述實驗結果表明,本文提出的基于靜態-動態蛋白質混合思想構建時序蛋白質網絡的方法具有一定的合理性.
圖3描述了在波動性閾值thdyn=0.2條件下,YKL101w、YNL286w和YLR011w3個靜態蛋白質及其相鄰接的部分動態蛋白質,在時刻1、4、7、10等4個時間點所構成的蛋白質網絡. 從4個示例網絡中可見,在每個時刻,與指定的靜態蛋白質鄰接的動態蛋白質有所差別,既體現了靜態蛋白質的穩定性,同時也刻畫了動態蛋白質的變化特點.
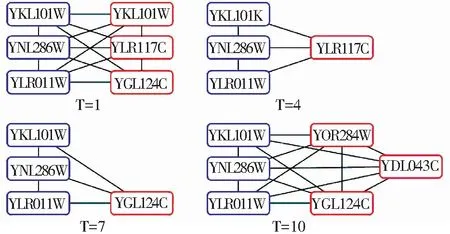
圖3 在thDyn=0.2條件下,第1、4、7和10時刻點蛋白質子網絡示意圖
Fig.3 Sub-networks on time 1, 4, 7 and 10 under the condition of thDyn=0.2
2.3 不同類型蛋白質網絡的對比
下面將HTPIN方法構建的時序網絡、同類方法DPIN方法[11]所構建的時序網絡進行比較. DPIN與HTPIN均是用來構建時序蛋白質網絡的方法. 二者主要區別在于,DPIN方法將所有蛋白質均視為動態蛋白質,而HTPIN方法則根據表達量波動情況將蛋白質劃分為動態、靜態兩類,并在此基礎上構建混合蛋白質網絡. 通過比較兩類方法可以證明本文提出的基于“混合”思想構建時序網絡方法的有效性. 此外,還與原始蛋白質網絡進行了對比分析. 實驗中,HTPIN方法采用了2.2節中獲得最優組合評分的波動性閾值.
2.3.1 識別復合體與已知復合體匹配評分
表1列出了CPL、ClusterONE以及PLSMC三種算法在不同類型蛋白質網絡上識別復合體的評價指標,其中組合評分為F-Measure、ACC和MMR三項之和[15]. 在HTPIN和DPIN構建的網絡上,ClusterONE和PLSMC算法識別結果的組合評分要好于在原始蛋白質網絡評分值,表明通過融合基因表達數據構建時序蛋白質網絡可以提高蛋白質復合體識別的準確性的. 值得注意的是,所有參與測試的復合體識別算法在HTPIN方法構建的網絡上均取得了最優的組合得分,表明本文方法較DPIN方法更有利于復合體的識別.
2.3.2 識別復合體的功能一致性分析
下面對復合體識別算法在不同類型網絡上識別復合體的GO功能一致性進行了分析. 利用GOTermFinder工具[20]分析所識別復合體的功能一致性. 實驗中計算了在p-value≤1.0E-10時功能一致性的復合體占所有識別復合體的比例. 該比例越高,表明所識別復合體功能一致性越高,具有更強的生物意義.
表1 不同蛋白質網絡識別的復合體與已知復合體匹配
Tab.1 Comparison of matching between known complexes and the complexes detected by using different protein networks
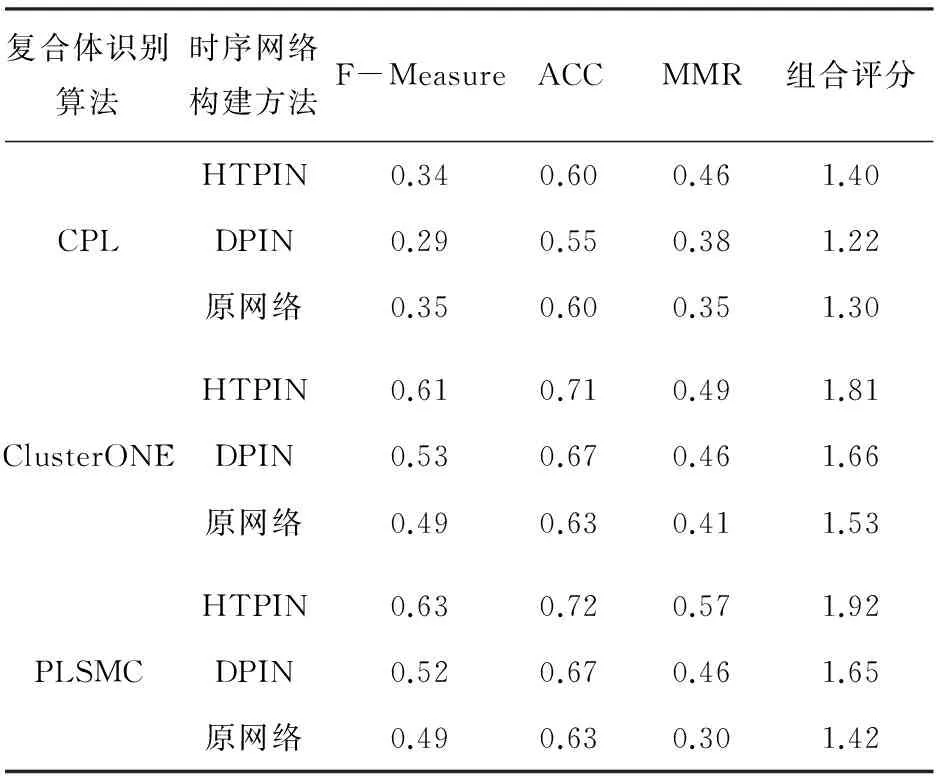
復合體識別算法時序網絡構建方法F-MeasureACCMMR組合評分HTPIN0.340.600.461.40CPLDPIN0.290.550.381.22原網絡0.350.600.351.30HTPIN0.610.710.491.81ClusterONEDPIN0.530.670.461.66原網絡0.490.630.411.53HTPIN0.630.720.571.92PLSMCDPIN0.520.670.461.65原網絡0.490.630.301.42
表2 不同蛋白質網絡下識別復合體的功能一致性
Tab.2 Comparison of function homogeneity of the complexes detected by using different protein networks
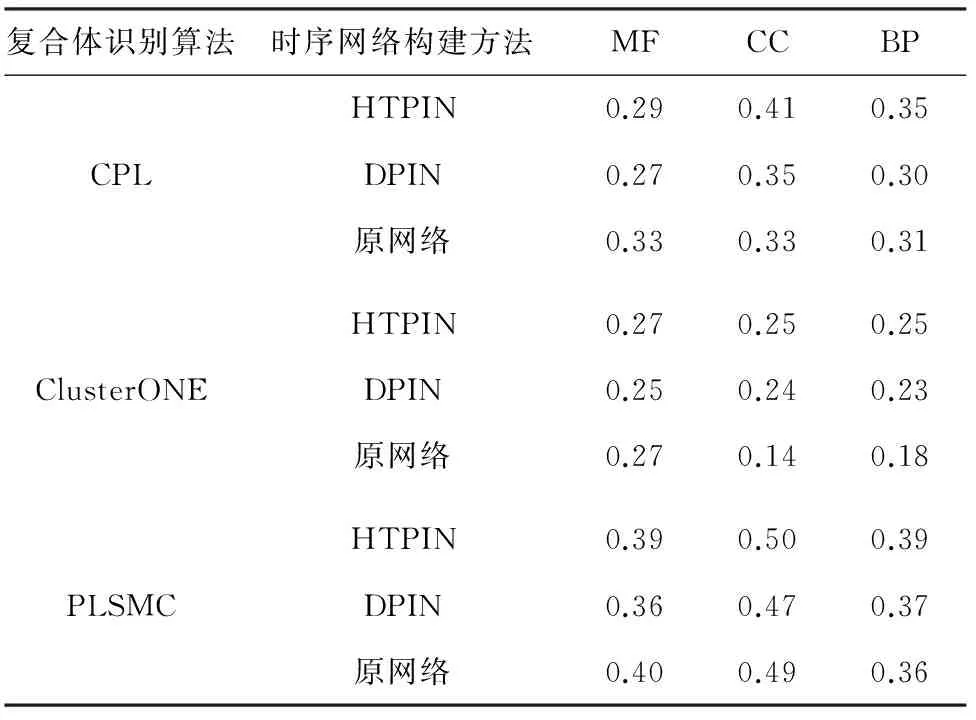
復合體識別算法時序網絡構建方法MFCCBPHTPIN0.290.410.35CPLDPIN0.270.350.30原網絡0.330.330.31HTPIN0.270.250.25ClusterONEDPIN0.250.240.23原網絡0.270.140.18HTPIN0.390.500.39PLSMCDPIN0.360.470.37原網絡0.400.490.36
表2列出了CPL、ClusterONE和PLSMC三種算法在不同類型網絡上識別復合體的功能一致性比例. 表2列出了關于GO所包含的三個方面功能注釋的分析結果,分別為分子功能(MF)、細胞組件(CC)和生物過程(BP). 從表2可以看出,所有參與測試識別算法在HTPIN與DPIN兩種時序蛋白質網絡上所識別的復合體,在細胞組件和生物過程兩個方面的功能一致性均要優于在原始網絡上的識別結果. 并且,基于HTPIN網絡的識別結果要優于基于DPIN的識別結果. 上述實驗結果表明,利用HTPIN方法構建的時序網絡可以提高識別復合體的功能一致性.
3 結論與展望
動態-靜態蛋白質混合的時序蛋白質網絡構建方法的主要特點是,在構建各時刻網絡過程中,同時考慮動態蛋白質與靜態蛋白質的相互作用關系. 實驗結果表明,這種具有混合特征的時序網絡能夠提高復合體識別的準確性,從而驗證了基于“動-靜混合”思想構建時序蛋白質網絡的方法是合理的.
值得注意的是,本文方法中的波動性閾值本質上是用于控制網絡中靜態與動態蛋白質比例的一種參數. 文中雖然考察了該參數的不同取值對網絡構建的影響,但并未關注該參數的具體計算方法. 作為下一步的工作,將繼續研究用于確定波動性閾值的自動計算方法,以增強本文方法的實際應用能力.
[1] TONG A H, DREES B, NARDELLI G, et al. A combined experimental and computational strategy to define protein interaction networks for peptide recognition modules[J]. Science, 2002, 295(5553): 321-324.
[2] SPIRIN V, MIRNY L A. Protein complexes and functional modules in molecular networks[J]. Proceedings of the National Academy of Sciences, 2003, 100(21):12123-12128.
[3]冀俊忠, 劉志軍, 劉紅欣, 等. 蛋白質相互作用網絡功能模塊檢測的研究綜述[J]. 自動化學報, 2014, 40(4):577-593.
JI Junzhong, LIU Zhijun, LIU Hongxin, et al. An overview of research on functional module detection for protein-protein interaction networks[J]. Acta Automatica Sinica, 2014, 40(4): 577-593.
[4]魚亮, 高琳, 孫鵬崗. 蛋白質網絡中復合體和功能模塊預測算法研究[J]. 計算機學報, 2011, 34(7):1239-1251.
YU Liang, GAO Lin, SUN Penggang. Research on algorithms for complexes and functional modules prediction in protein-protein interaction networks[J]. Chinese Journal of Computers, 2011,34(7): 1239-1251.
[5]湯希瑋, 王建新, 胡秋玲. 蛋白質復合物預測方法分析與比較[J]. 計算機應用研究, 2011, 28(10): 3611-3614.
TANG Xiwei, WANG Jianxin, HU Qiuling. Analysis and compare of methods predicting protein complex[J]. Application Research of Computers[J]. 2011, 28(10): 3611-3614.
[6]黃海濱, 楊路明, 王建新, 等. 基于復合參數的蛋白質網絡關鍵節點識別技術[J]. 自動化學報, 2008, 34(11):1388-1395.
HUANG Haibin, YANG Luming, WANG Jianxin, et al. Identification technique of essential nodes in protein networks based on combined parameters[J]. Acta Automatica Sinica, 2008, 34(11): 1388-1395.
[7] LICHTENBERG U, JENSEN L J, BRUNAK S, et al. Dynamic complex formation during the yeast cell cycle[J]. Science, 2005, 307(5710):724-727.
[8] TANG Xiwei, WANG Jianxin, LIU Binbin, et al. A comparison of the functional modules identified from time course and static PPI network data[J]. BMC Bioinformatics, 2011, 12(1):1-15.
[9] WANG Jianxin, PENG Xiaoqing, PENG Wei, et al. Dynamic protein interaction network construction and applications[J]. Proteomics, 2014, 14(4-5): 338-352.
[10]HEGDE S R, MANIMARAN P, MANDE S C. Dynamic changes in protein functional linkage networks revealed by integration with gene expression data[J]. PLoS Computational Biology, 2008, 4(11):e1000237.
[11]WANG Jianxin, PENG Xiaoqing, LI Min, et al. Construction and application of dynamic protein interaction network based on time course gene expression data[J]. Proteomics, 2013, 13(2):301-312.
[12]KOMUROV K, WHITE M. Revealing static and dynamic modular architecture of the eukaryotic protein interaction network[J]. Molecular Systems Biology, 2007, 3(1):110.
[13]JANJIC V, SHARAN R, PRZULJ N. Modelling the Yeast Interactome[J]. Scientific Reports, 2014, 4:4273.
[14]PU S, WONG J, TURNER B, et al. Up-to-date catalogues of yeast protein complexes[J]. Nucleic Acids Research, 2009, 37(3):825-831.
[15]DAI Qiguo, GUO Maozu, GUO Yingjie, et al. A least square method based model for identifying protein complexes in protein-protein interaction network[J]. Biomed Research International, 2013,2014:720960-720960.
[16]DAI Qiguo, GUO Maozu, LIU Xiaoyan, et al. CPL: Detecting protein complexes by propagating labels on protein-protein interaction network[J]. Journal of Computer Science and Technology, 2014, 29(6): 1083-1093.
[17]NEPUSZ T, YU H, PACCANARO A. Detecting overlapping protein complexes in protein-protein interaction networks[J]. Nature Methods, 2012, 9(5): 471-472.
[18]TU B P, KUDLICKI A, ROWICKA M, et al. Logic of the yeast metabolic cycle: temporal compart-mentalization of cellular processes[J]. Science, 2005, 310(5751):1152-1158.
[19]STARK C, BREITKREUTZ B J, REGULY T, et al. BioGRID: a general repository for interaction datasets[J]. Nucleic Acids Research, 2006, 34(suppl 1):D535-D539.
[20]BOYLE E I, WENG S, GOLLUB J, et al. GO: TermFinder—open source software for accessing Gene Ontology information and finding significantly enriched Gene Ontology terms associated with a list of genes[J]. Bioinformatics, 2004, 20(18):3710-3715.
(編輯 王小唯 苗秀芝)
A method of constructing temporal protein networks by hybridizing dynamic and static proteins
DAI Qiguo1,2,3, GUO Maozu1, LIU Xiaoyan1, WANG Chunyu1
(1.School of Computer Science and Technology, Harbin Institute of Technology, Harbin 150001, China;2. School of Computer Science and Engineering, Dalian Minzu University, Dalian 116600, Liaoning, China;3.Dalian Key Lab of Digital Technology for National Culture(Dalian Minzu University), Dalian 116600, Liaoning, China)
Public available protein networks at present are static, which could not be used to describe the dynamic characteristics of proteins in a cell effectively. It is necessary to construct temporal protein network by integrating other biological data, which reflects the dynamic activities of proteins. Most of previous methods assume that all proteins are dynamic. However, in addition to dynamic protein, there are many static proteins in the cell. To this end, this paper proposes a new method to construct a temporal protein network both with dynamic and static proteins. In the method, proteins are classified into two types of dynamic and static, and then a protein network is constructed on each time point by both considering the interactions of dynamic and static proteins. Experimental test results show that the temporal protein network constructed by using the proposed method can improve the accuracy of the identification of protein complexes, which verified the reliability of the proposed method.
temporal protein network; protein interaction; gene expression; biological network; protein complex identification
10.11918/j.issn.0367-6234.2016.11.007
2015-05-08
國家自然科學基金(91335112,61571163,61532014,61402132,61271346);中央高校基本科研業務費專項資金(DC201501030)
代啟國(1985—),男,講師; 郭茂祖(1966—),男,教授,博士生導師
郭茂祖,maozuguo@hit.edu.cn
TP391
A
0367-6234(2016)11-0041-06

