海島棉基于雙色FISH的核型分析
肖水平,王坤波,柯興盛,楊 磊,劉新穩,孫亮慶,楊紹群,陳 宜*
(1.江西省棉花研究所/國家棉花產業技術體系鄱陽湖綜合試驗站,江西 九江 332105;2.中國農業科學院 棉花研究所/棉花生物學國家重點實驗室,河南 安陽 455000)
?
海島棉基于雙色FISH的核型分析
肖水平1,2,王坤波2,柯興盛1,楊 磊1,劉新穩1,孫亮慶1,楊紹群1,陳 宜1*
(1.江西省棉花研究所/國家棉花產業技術體系鄱陽湖綜合試驗站,江西 九江 332105;2.中國農業科學院 棉花研究所/棉花生物學國家重點實驗室,河南 安陽 455000)
摘要:以45S rDNA作探針對海島棉體細胞中期染色體進行熒光原位雜交(Fluorescence in situ Hybridization,FISH),檢測出6個強的熒光信號(4大2小),結合DAPI圖像觀察發現信號分布在各自染色體隨體位置,稱為NOR(Nucleolar Organizer Region)信號。以二倍體A基因組棉種亞洲棉基因組DNA(gDNA)為探針對海島棉進行FISH,結果發現52條染色體中有無雜交信號的各占一半,熒光信號分布在較長的A亞組染色體上,直接區分開海島棉A、D亞組染色體,直觀證實了海島棉的異源雙二倍體起源。以45S rDNA和A組棉種亞洲棉gDNA作探針對海島棉進行雙色FISH時,結果發現海島棉染色體上既檢測到了6個強的NOR信號又檢測出了gDNA雜交信號,且清楚觀察到2個NOR信號分布在A亞組上,另4個在D亞組上。基于該雙色FISH圖像的核型分析表明,海島棉的核型公式為:2n=4x=52=40m(2 SAT)+12sm(4 SAT),屬于2A類型,且有3對隨體,其中1對定位于A9號染色體,屬于中部著絲點(m)類型;另2對分別定位于D8和D12號染色體上,屬于近中部著絲點(sm)類型;且發現海島棉A亞組染色體的相對長度的并非全部大于D亞組,兩亞組染色體間在長度上存在交叉,說明基于FISH的核型分析比傳統方法更為準確。
關鍵詞:海島棉;45S rDNA;gDNA;FISH;核型分析
目前,棉屬(GossypiumL.)已經定名的種(亞種)共51個[1],其中二倍體棉種(2n=2x=26)46個,包括A~G及K共8個基因組;另外四倍體(2n=4x=52)棉種5個,包括2個栽培棉種,3個野生棉種,其中海島棉是目前世界上大面積種植的主要栽培種之一。研究表明,海島棉染色體由異源雙二倍體組成[2-3],用(AD)2表示。二倍體A和D基因組則分別是四倍體棉種A和D亞組的供體種[4-5]。
Beasley[6]和Phillips[7]在關于棉種的細胞學研究中,發現A組棉種的染色體大于D組棉種。Kadir等[8]通過棉種的基因組DNA(genomic DNA,簡稱gDNA)含量比較,也證實了A組棉種gDNA明顯大于D組棉種。這為后來關于四倍體棉種核型分析中對確定A、D亞組染色體順序編號提供了依據[9-10]。但王坤波等[11]在對陸地棉的核型分析中,發現僅僅依據核型圖像中染色體的長短順序確定四倍體棉種的A、D亞組未免有失偏頗,并提出了原位雜交(in situ hybridization)技術對四倍體棉A、D亞組的確定可能更為理想。
熒光原位雜交技術(Fluorescence in situ hybridization,簡稱FISH)于20世紀80年代中期開始應用于植物的研究當中,至今已經發展了近30年,在植物染色體識別、細胞遺傳圖譜構建、重復序列定位、物種起源與進化及基因組間親緣關系等方面應用廣泛[12-17]。
FISH技術在棉花上的應用遠落后于其他作物,主要原因在于棉花在染色體制片過程中,其細胞質濃厚,細胞壁較堅硬,酶解細胞比較困難,再加上棉花染色體數目較多、且形態小。
本研究以45S rDNA和二倍體A基因組gDNA為探針對靶材料海島棉根尖體細胞有絲分裂中期染色體進行FISH分析,旨在利用FISH技術對海島棉染色體進行核型分析,為海島棉遺傳物理圖譜的構建及亞組染色體的鑒定與識別提供更為準確的數據。
1材料與方法
1.1材料
用作探針的DNA為二倍體A基因組棉種亞洲棉“石系亞1號”,取自于中國農業科學院棉花研究所海南三亞野生棉種質圃;用作探針的45S rDNA是一個來源于擬南芥的克隆JHD2-15A(8.2 kb,BgⅠ+XhoⅠ片段,含擬南芥幾乎全45S rDNA重復序列),由北京林業大學戴絲蘭教授提供。用作原位雜交的靶材料為四倍體海島棉品種“新海7號”根尖體細胞染色體。另外為了書寫方便,本文圖中所涉及到的原位雜交組合均按照“靶染色體-探針-封阻DNA”格式書寫。
1.2探針的制備與標記
棉花gDNA的提取與純化參照宋國立等[18]的方法,純化后的棉花基因組總DNA經高壓滅菌鍋滅菌打斷10 min,0.8%瓊脂糖凝膠電泳檢查片段大小,其大小要求在300~600 bp范圍為宜。探針的標記:其中gDNA探針所用的標記物為DIG-11-dTUP(顯示紅色熒光),采用德國Roche公司的DIG-High-Prime標記系統對其進行標記,按其提供的標準流程操作;45S rDNA探針則采用該公司的biotion-Nick-Translation MIX(顯示綠色熒光)標記系統對其進行標記,按其提供的標準流程操作。
1.3靶染色體的制備
染色體制片按王春英等[19]的方法稍加改進。取生長旺盛的根尖,用25×10-6的放線菌酮于20 ℃處理2 h,新鮮卡諾固定液于4 ℃下固定2~24 h。用4%的纖維素酶(Sigma)和0.5%的果膠酶(Sigma)在37 ℃下酶解1 h,水洗后,60%乙酸壓片,液氮冷凍揭蓋片,-20 ℃保存備用。
1.4原位雜交流程
熒光原位雜交流程參照王春英等[20]的操作方法進行。
1.5觀察與圖像處理
在熒光顯微鏡(Ziess Axioskop 2 plus)下觀察各雜交信號,DAPI(4’,6-diamidino-2-phenylindole)、Avidin-Fluorescein和PI(propidium iodide)分別使用濾鏡filter set 49、10和15觀察。同時用ISIS(in situimaging system)軟件拍攝并調節圖片對比度和亮度。測量有絲分裂中期染色體的長度和臂比,染色體形態描述、核型分析參照李懋學等[21]的方法進行。同源染色體的序號確定,則以具有A基因組gDNA探針雜交信號的染色體為A亞組,無雜交信號的染色體為D亞組,兩組均依據染色體長度從長到短進行編號。
2結果與分析
2.1海島棉45S rDNA-FISH
以45S rDNA作探針,用鮭魚精DNA(簡稱ssDNA)作封阻,對海島棉根尖體細胞有絲分裂中期染色體進行FISH(圖1-A),觀察到6個強的綠色熒光信號(圖中白色箭頭所示),4個較大、2個稍小,全部位于染色體短臂端部,與DAPI(是一種與DNA特異結合的熒光染料,染色體被襯染成藍色)圖像進一步對比,發現所有信號均分布在各染色體的隨體位置,稱之為NOR信號(Nucleolar Organizer Region,即核仁組織區)[20],結果表明海島棉有3對隨體。
2.2海島棉gDNA-FISH
以二倍體A組亞洲棉“石系亞1號”gDNA作探針,用ssDNA作封阻,對海島棉體細胞中期染色體進行FISH(圖1-B),結果發現海島棉的52條染色體中,可以觀察到強的紅色雜交信號主要分布在較長的26條A亞組染色體上,而顯示藍色熒光信號(DAPI襯染)的則在較短的26條D亞染色體,直接將海島棉A、D兩亞組染色體區分開,直觀證實了海島棉的異源雙二倍體起源[22],為后續的染色體核型分析奠定了基礎。
2.3海島棉基于雙色FISH的核型分析
為了更準確地對海島棉進行核型分析,筆者同時利用二倍體A組亞洲棉“石系亞1號”gDNA與45S rDNA作探針,ssDNA作封組,對海島棉進行雙色FISH(圖1-C),結果發現不僅二倍體A組的gDNA探針雜交信號可以明顯區分開海島棉A、D亞組染色體,而且45S rDNA探針產生的6個NOR信號則清晰地定位在了3對染色體上,可以看到其中1對在A亞組上(圖中綠色箭頭),2對在D亞組上(圖中白色箭頭)。
基于該雙色FISH結果,進一步獲得了海島棉的核型圖(圖1-D)和核型基本參數(表1),由圖1和表1可知,海島棉的染色體多為對稱類型,在52條染色體中,中部著絲點類型(m)的有40條,其中A亞組占18條,D亞組占22條;近中部著絲點類型(sm)的有12條,其中A亞組占8條,D亞組占4條,無其他著絲點類型、兩著絲點類型占總數的百分率依次為77%和23%,說明海島棉染色體整體的對稱程度很高,是一個比較原始的棉種。在其全部基因組中,染色體長度比(最長染色體與最短染色體的比值)為1.85,平均染色體相對長度為3.85。另外,在海島棉全部基因組中,其臂比值最大的為2.11(A11號染色體),最小的為1.08(D9號染色體),平均臂比值為1.46。將A、D亞組分開分析,A亞組平均臂比值為1.58,D亞組的為1.32,前者大于后者。海島棉的核型公式為:2n=4x=52=40m(2SAT)+12sm(4SAT),屬于2A類型,海島棉有3對隨體,其中1對位于A亞組的第9號染色體,屬于中部著絲點(m)類型;另2對分別位于D亞組的第8和12號染色體,屬于近中部著絲點(sm)類型,且3對隨體都分布在染色體短臂端部。基于雙色FISH的核型參數分析,發現海島棉D亞組核型的對稱程度比A亞組的高,即D亞組中部著絲點染色體數多于A亞組,說明在進化程度上A亞組強于D亞組。
另外從核型參數表中發現,海島棉A、D兩亞組染色體在相對長度上存在一定程度的交叉現象,即較短染色體組的D亞組第1號(D1)染色體的相對長度(為3.70)大于A亞組染色體的最后2對(A12、A13號分別為3.69、3.62),前人的研究也發現過這種情況[23-24],這說明基于原位雜交的核型分析方法較傳統的只按長度大小排序的方法更為準確可靠。
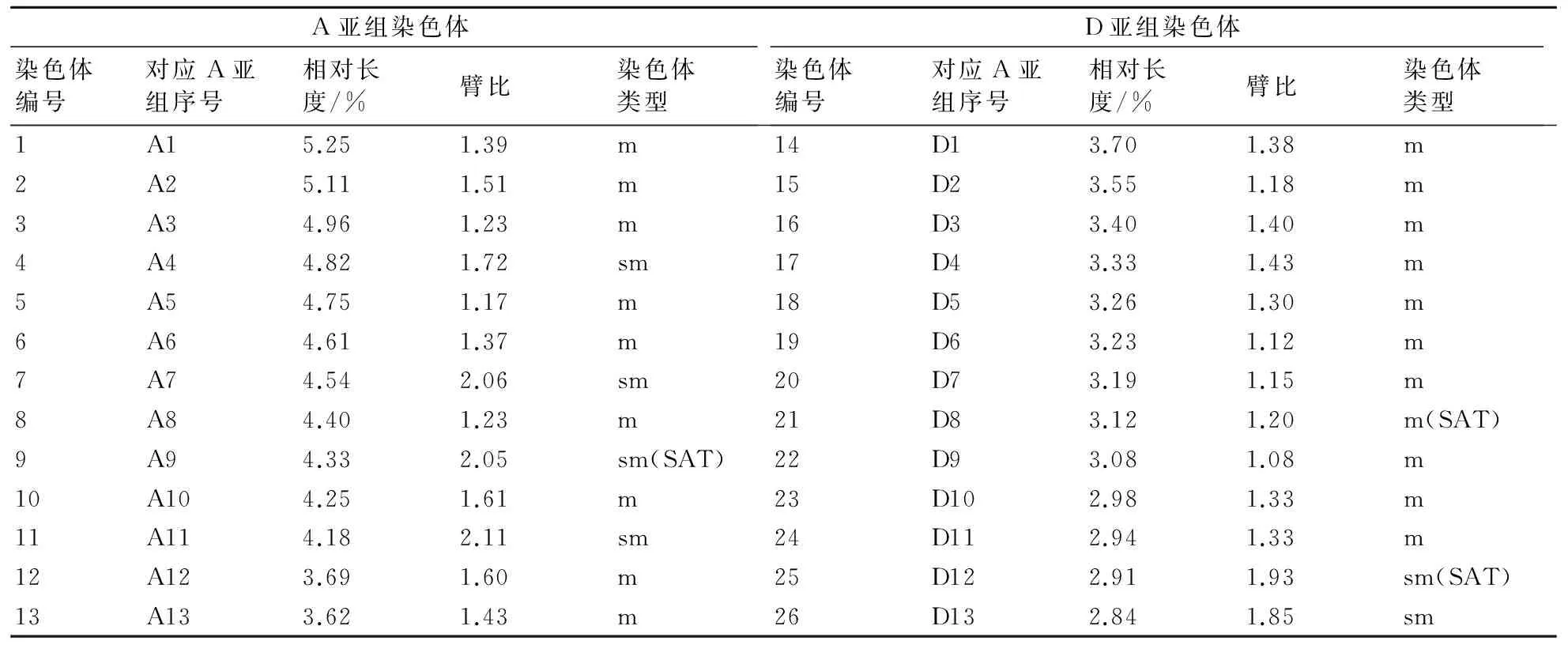
表1 海島棉基于雙色FISH核型參數
注:隨體的長度不參與染色體全長的計算,m:中部著絲粒染色體,臂比范圍1.01~1.7;sm:近中著絲粒染色體,臂比范圍1.71~3.00;SAT:代表隨體。
3結論與討論
將本核型分析結果與聶汝芝等[22]應用傳統細胞學方法對海島棉的核型分析結果相比較,其提出的中部著絲點類型(m)的染色體數為38條,本實驗的m類型染色體數為40條,相差2條;近中部著絲點類型(sm)的染色體數為12條,本實驗結果與其一致,也為12條;隨體數目前者為4個,本實驗的隨體數為6個(4大2小),比其多檢測出2個,但前者還多出2條近端部著絲點類型染色體,本實驗通過45S rDNA-FISH發現,其應為2個較小的隨體所在的染色體,本實驗結果將其歸為中部著絲點類型;染色體平均相對長度前者為2.08,本實驗的為3.85,兩者相差較大;平均臂比值前者為1.55,本實驗的為1.46,存在一定差異。
另外,本實驗核型分析結果以海島棉雙色FISH圖像為依據,由于A基因組gDNA探針的雜交信號,明顯地區分開了A、D兩亞組染色體,然后再依據各亞組染色體長短確定各自順序編號,這種方法對于核型分析更為客觀。雖然細胞學和生物化學研究表明,棉屬A染色體組棉種的染色體(DNA)大于D染色體組棉種[8,25],但并非說明A亞組棉種13對染色體均大于D亞組棉種的所有染色體,本文基于雙色FISH圖像的核型分析結果證實了海島棉2個亞組染色體的相對長度存在交叉現象,這也正是本文核型分析結果與聶汝芝等結果存在差別的原因。所以,核型分析中,雙二倍體起源的四倍體棉種僅僅依據染色體相對長短鑒別2個亞組的染色體是不夠完善的,反之,基于FISH技術來鑒別各染色體組成及確定其物理序號則更為理想,這為多倍體物種特別是異源多倍體物種的核型分析研究提供了更為準確的技術支持。
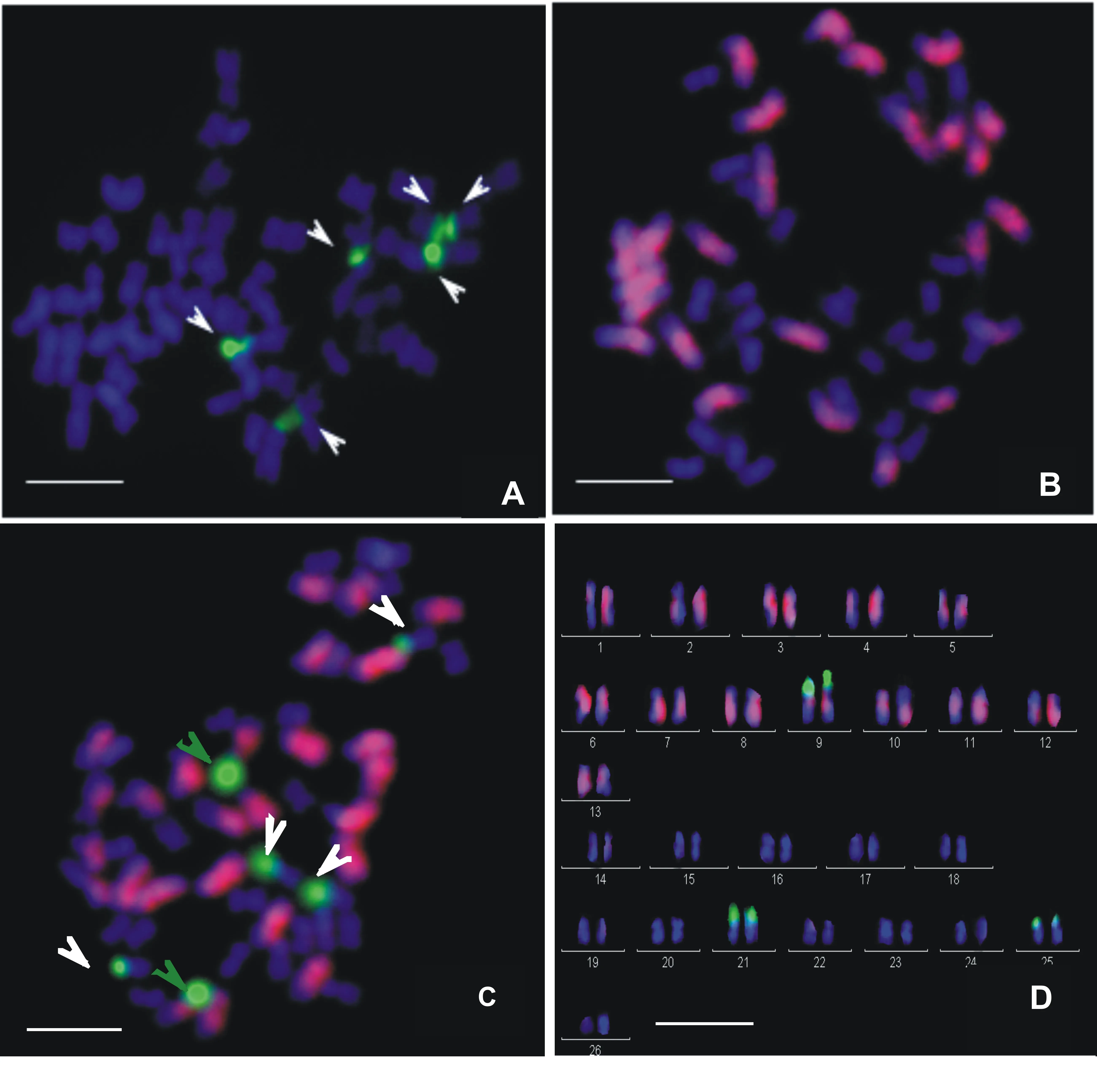
A、B分別是以45S rDNA和A組亞洲棉石系亞1號gDNA作探針,鮭魚精DNA作封阻對海島棉的FISH結果;C為同時以45S rDNA和A組亞洲棉石系亞1號gDNA作探針,鮭魚精DNA作封阻的雙色FISH結果;D為基于C的核型分析圖,其中1~13號為A亞組染色體,14~26號為D亞組染色體;Bar=10m
圖1海島棉FISH圖
參考文獻:
[1] Fryxell P A. A revised taxonomic interpretation ofGossypiumL (Malvaceae)[J]. Rheedea, 1992, 2(2): 108-165.
[2] Phillips L L. The cytology and phylogenetics of the diploid species ofGossypium[J]. American Journal of Botany, 1966, 53(4): 328-335.
[3] Endrizzi J E, Turcotte E L, Kohel R J. Genetics, cytology, and evolution ofGossypium[J]. Adv Genet, 1985, 23: 271-375.
[4] Wendel J F, Albert V A. Phylogenetics of the cotton genus (GossypiumL.): Character-state weighted parsimony analysis of chloroplast DNA restriction site data and its systematic and biogeographic implications[J]. Systematic Botany, 1992, 17(1): 115-143.
[5] Wendel J F, Cronn R C. Polyploidy and the evolutionary history of cotton[J]. Advances in Agronomy, 2002, 78(2): 139-186.
[6] Beasley J O. Meiotic chromosome behavior in species, species hybrids, haploids and induced polyploids ofGossypium[J]. Genetics, 1942, 27(1): 25-54.
[7] Phillips L L. The cytogenetics ofGossypiumand the origin of New World cottons[J]. Evolution, 1963, 17(4): 460-496.
[8] Kadir Z B A. DNA evolution in the genusGossypium[J]. Chromosome, 1976, 56(1): 85-94.
[9] 聶汝芝,李愁學.三種野生和四個栽培棉種核型研究[J].植物學報,1985,27(2):113-121.
[10] 張志端,趙吉勇.陸地棉和海島棉染色體組型比較[J].植物學通報,1988,5(3):173-175.
[11] 王坤波,宋國立,黎紹惠,等.陸地棉的核型模式[J].遺傳,1995,17(6):1-3.
[12] Jiang J M, Gill B S. Current status and the future of fluorescence in situ hybridization (FISH) in plant genome research[J]. Genome, 2006, 49(9): 1057-1068.
[13] Wang K, Guan B, Guo W Z, et al. Completely distinguishing individual A-genome chromosomes and their karyotyping analysis by multiple bacterial artificial chromosome-fluorescence in situ hybridization[J]. Genetics, 2008, 178(2): 1117-1122.
[14] Nemeth C, Yang C Y, Kasprzak P, et al. Generation of amphidiploids from hybrids of wheat and related species from the generaAegilops,Secale,Thinopyrum, andTriticumas a source of genetic variation for wheat improvement[J]. Genome, 2015, 58(2): 71-79.
[15] Melo C A, Silva G S, Souza M M. Establishment of the genomic in situ hybridization (GISH) technique for analysis in interspecific hybrids ofPassiflora[J]. Genet Mol Res, 2015, 14(1): 2176-2188.
[16] Li H, Guo X, Wang C, et al. Spontaneous and divergent hexaploid triticales derived from common Wheat×Rye by complete elimination of D-Genome chromosomes[J]. PLoS One, 2015, 10(3): e0120421.
[17] Mavrodiev E V, Chester M, Suárez-Santiago V N, et al. Multiple origins and chromosomal novelty in the allotetraploidTragopogoncastellanus(Asteraceae) [J]. The New phytologist, 2015, 206(3): 1172-1183.
[18] 宋國立,崔榮霞,郭立平,等.改良CTAB法快速提取棉花DNA[J].棉花學報,1998,10(5):273-275.
[19] 王春英,王坤波,宋國立,等.棉花體細胞染色體rDNA-FISH技術[J].棉花學報,2001,13(2):75-77.
[20] 王春英,王坤波,王文奎,等.棉花gDNA體細胞染色體FISH技術[J].棉花學報,1999,11(2):79-83.
[21] 李懋學,陳瑞陽.關于植物核型分析的標準化問題[J].武漢植物學研究,1985,3(4):297-302.
[22] 聶汝芝,李懋學.棉屬植物核型研究[M].北京:科學出版社,1993.
[23] 王坤波,王文奎,王春英,等.海島棉原位雜交及核型比較[J].遺傳學報,2001,28(1):69-75.
[24] 吳瓊,宋國立,劉三宏,等.黃褐棉45S rDNA的FISH定位及核型分析[J].植物遺傳資源學報,2008,9(4):439-442.
[25] Beasley J O. Meiotic chromosome behavior in species, species hybrids, haploids and induced polyploids ofGossypium[J]. Genetics, 1942, 27(1): 25-54.
(責任編輯:曾小軍)
收稿日期:2015-12-24
基金項目:經濟作物種質資源發掘與創新利用(2013BAD01B03);轉基因生物新品種培育科技重大專項(2011ZX08005002-009);江西省科技支撐計劃項目(20141BBF60011);國家棉花產業技術體系項目(CARS-18-36)。
作者簡介:肖水平(1984─),男,江西吉安人,助理研究員,碩士,從事棉花種質資源與遺傳育種研究。* 通訊作者:陳宜。
中圖分類號:S562
文獻標志碼:A
文章編號:1001-8581(2016)07-0006-05
Karyotype Analysis ofGossypiumbarbadenseBased on Dualcolor-FISH
XIAO Shui-ping1,2, WANG Kun-bo2, KE Xing-sheng1, YANG Lei1,LIU Xin-wen1, SUN Liang-qing1, YANG Shao-qun1, CHEN Yi1*
(1. Cotton Research Institute of Jiangxi Province/ Poyang Lake Station of Cotton Comprehensive Experiment, National Cotton Industrial Technique System, Jiujiang 332105, China; 2. Institute of Cotton Science, Chinese Academy of Agricultural Sciences/ State Key Laboratory of Cotton Biology, Anyang 455000, China)
Abstract:Through the fluorescence in situ hybridization (FISH) of somatic metaphase chromosomes of Gossypium barbadense with the probe of 45S rDNA, six strong NOR (nucleolar organizer region) fluorescent signals were detected, of which 4 were big and 2 were small, and these signals were distributed at the satellite site of corresponding chromosome by combining with DAPI image observation. The FISH of G. barbadense with the probe of genomic DNA (gDNA) of diploid A-genome asiatic cotton found that: there were hybrid signals in the half of 52 chromosomes; the fluorescent signals were distributed in longer A-subgenomic chromosomes, and directly distinguished the A-subgenomic and D-subgenomic chromosomes of G. barbadense, which directly proved that G. barbadense was originated from two different diploid species. Using the gDNA of A-genome asiatic cotton and 45S rDNA as probes, we conducted dualcolor-FISH of G. barbadense, and detected not only 6 strong NOR signals but also gDNA hybrid signals in the chromosomes of G. barbadense, and clearly found 2 NOR signals in sub-genome A and other 4 NOR signals in sub-genome D. The karyotype analysis of G. barbadense based on dualcolor-FISH images indicated that: the karyotypic formula of this cotton species was 2n=4x=52=40m (2 SAT)+12sm (4 SAT), belonging to 2A type; there were three pairs of satellite chromosomes, of which one was located in the number 9 of A sub-genome (m type), and the others were located in the number 8 and number 12 of D sub-genome (sm type). It was also found that not all A sub-genomic chromosomes of G. barbadense had a bigger relative length than D sub-genomic chromosomes, suggesting that the FISH-based karyotype analysis method was more accurate than traditional methods.
Key words:G. barbadense; 45S rDNA; gDNA; Fluorescence in situ hybridization (FISH); Karyotype analysis

