傳統奶酪樣品中乳酸菌的分離鑒定
楊彥榮,任艷,德亮亮,陳紅霞,張冬蕾,劉文俊,張和平
(內蒙古農業大學 乳品生物技術與工程教育部重點實驗室,呼和浩特 010018)
0 引言
傳統發酵奶制品的制作與食用已有數千年的歷史,生產和消費群體遍布世界各地[1]。其中奶酪又是乳中的精品,被譽為“乳業皇冠上的珍珠”。
大量的研究表明,以牛奶為原料通過傳統發酵方法生產的新鮮奶酪制品中包含著豐富的微生物群落,微生物的組成是影響奶酪質量的關鍵因素[2,3]。然而,有關俄羅斯地區傳統發酵乳制品中乳酸菌的研究還相對較少,對其中微生物特別是乳酸菌的研究還不夠深入。
本研究以實驗室純培養的方法,和16S rRNA基因序列分析方法對俄羅斯卡爾梅克地區奶酪的乳酸菌進行分離鑒定,以期為傳統乳制品的品質改良和優良發酵劑菌種的篩選提供基礎知識和珍貴的乳酸菌資源。
1 引言
1.1 材料
1.1.1 樣品來源
本研究的7份傳統發酵奶酪樣品是由內蒙古農業大學乳品生物技術與工程教育部重點實驗室的研究人員于2012年8月~9月于俄羅斯卡爾梅克共和國地區采集的。
1.1.2 培養基與試劑
培養基:MRS固體培養基BD(DifocTMLactobacilli MRS Agar),MRS液體培養基(OXOID),M17液體培養基(OXOID),M17液體培養基加1.5%瓊脂為M17固體培養基,M17培養基中添加2%葡萄糖和0.5%乳糖。
PBS:0.8%氯化鈉,0.02%磷酸二氫鉀,0.115%磷酸氫二鈉。
脫脂乳保護劑:10 g脫脂乳粉,0.1 g谷氨酸鈉,蒸餾水 90 mL(121℃,7 min滅菌,98℃急冷)。
提取DNA所用試劑:0.5 M EDTA,10%SDS,10 mol/L CTAB,TE緩沖液(pH8.0),酚/氯仿/異戊醇(25∶24∶1,體積比),氯仿/異戊醇(24∶1,體積比),濃度為5 mol/L的NaCl,3 mol/L的NaAc,異丙醇,乙醇,蛋白酶K(Proteinase K)、核糖核酸酶A(RnaseA)。
PCR擴增和電泳檢測所用試劑:Easy Taq DNA聚合酶、10×Easy Taq PCR緩沖液(Mg2+)、高純度dNTPs(每種2.5 mmol/L)、5×TBE電泳緩沖液貯液、0.8%~1.2%的瓊脂糖凝膠、核酸染料GELVIEW(北京百泰克)、λ DNA/Hind III Marker和QDL2,000 Quantitative DNA Marker。以上配制試劑所用的化學藥品均為天津市北方天醫化學試劑公司的產品,為分析純。上述酶類和Marker均購自北京全式金生物技術有限公司和大連寶生物技術有限公司(TaKaRa)。由內蒙古農業大學乳品生物技術與工程教育部重點實驗室提供。
1.1.3 設備
JT102N電子天平,HIRAYAMA HA-300M全自動高壓蒸汽滅菌器,ADVANTEC SP-650全自動高壓干熱滅菌器,ZHJH-C1214C雙人雙面超凈工作臺,OLYMPUS BX50光學顯微鏡及OLYMPUS PM-2攝像系統,LABC0NC0LL-6SFPY真空冷凍干燥機,EYELA WFO-400濃縮干燥系統,MIR-1620型電熱恒溫培養箱,ZHWY-200D型水平搖床,HHS 1-Ni電熱恒溫水浴鍋,Eppendorf TGL-168高速臺式離心機,Eppendorf 5810高速冷凍離心機,MJ RESEARCH PTC-200梯度基因擴增儀,DYY-12電泳儀,UVPGDS-8000凝膠成像儀,ND-1000型微量紫外分光光度計。
1.2 方法
1.2.1 樣品采集
本研究在采集樣品時直接用無菌勺取5.0 g左右樣品放入裝有0.5 g滅菌中和劑(淀粉/CaCO3,50∶1,質量比)的無菌螺口凍存管中,混勻后用封口膜封口后標記樣品號。將采集的所有樣品放入4℃便攜式冰箱內保持低溫度狀態,帶回實驗室后,盡快進行微生物組成分析和乳酸菌的分離實驗。
1.2.2 乳酸菌的組成分析、分離純化與保存
乳酸菌計數:采用傾注法。將樣品用漩渦振蕩器混勻,用質量分數為0.85%的無菌生理鹽水以10倍稀釋法對樣品進行梯度稀釋,分別選取稀釋度為10-5,10-6,10-7的稀釋液1 mL于無菌培養皿中,與MRS和M17平板計數培養基混勻,置于30℃恒溫培養箱中倒置厭氧培養48 h。然后計數即為每毫升樣品的菌落數[4]。
將上述梯度為10-4,10-5,10-6的稀釋液分別涂布于含有0.01%(體積份數)放線菌酮的無菌MRS和M17固體培養基上,30厭氧培養48 h,挑取形態學特征不同的單個菌落于相應的液體培養基中,30℃培養24 h。選取過氧化氫陰性、革蘭氏陽性的純培養物,加入含質量分數為0.1%谷氨酸鈉的脫脂乳保護劑,分裝于無菌安瓿管中和凍存管中于-80℃保存,備用[5]。
1.2.3 乳酸菌鑒定
(1)基因組DNA提取與純度檢測。純化好的菌株用CTAB法和凍融法相結合提取基因組DNA[6-7]。使用ND-1000型微量紫外分光光度計檢測基因組DNA的濃度和純度。
(2)16S rRNA基因的PCR擴增。PCR擴增16S rRNA引物采用通用引物,正向引物為FA-27F(GCAGAGTTCTCGGAGTCA CGAAGAGTTTGAT CCTGGCTCAG);反向引物為 RA-1495R(AGCGGATCACTTC ACACAGGACTACGGCTACCTTGT TACGA)[8]。由上海桑尼生物科技有限公司合成。PCR擴增片段約為1 450 bp。取2 μL產物用質量濃度為0.1 g/dL瓊脂糖凝膠電泳檢測。
16S rRNA基因的PCR擴增體系:5 μ L 10×Easy Taq Buffer(+Mg2+);4 μ L High Pure dNTPs(濃 度 2.5 mmol/L);1.5 μL引物FA-27F(濃度10 mmol/L);1.5 μ L引物 RA-1495R(濃度 10 mmol/L);0.5 μLEasyTaq DNA polymerase(5U/μL);2 μLDNA模板(質量濃度100 ng/μL);35.5 μL ddH2O。
16S rRNA基因的PCR擴增循環參數為:94℃預變性5 min;94℃變性1 min,58℃退火1 min,72℃延伸2 min,30個循環;72 ℃末端延伸10 min[9]。
擴增反應完畢后,取約2 μL的PCR擴增產物用1.0%的瓊脂糖凝膠電泳檢測,若在1 500 bp處有清晰的擴增條帶且無拖尾、彌散現象,則PCR擴增成功。
(3)16S rRNA序列測定和構建系統發育樹。將檢測后片段長度約為1500 bp的陽性產物直接送上海桑尼生物技術有限公司進行序列測定。采用DNAStar 5.01軟件中的SepMan模塊,對所測得菌株的兩條16S rRNA基因序列進行整理、拼接、校準,得到1 450 bp左右的有效序列,于NCBI數據庫中利用BLAST(http://blast.ncbi.nlm.nih.gov/Blast.cgi)工具與GenBank數據庫中已知菌株的16S rRNA基因序列進行比對鑒定[10],尋找同源性最高的已知分類學地位的菌種。
利用軟件Mega5中的Neighbor-joining法對37株乳酸菌作系統進化樹,比較了它們之間的進化距離[5,11]。
3 結果與討論
3.1 乳酸菌的計數
俄羅斯地區樣品中乳酸菌的計數結果如表1所示。
表1 俄羅斯卡爾梅開共和國傳統發酵奶酪樣品中乳酸菌計數結果(n=3,±SD)

表1 俄羅斯卡爾梅開共和國傳統發酵奶酪樣品中乳酸菌計數結果(n=3,±SD)
樣品編號Ru 4 Ru 5 Ru 6 Ru 24 Ru 26 Ru 30 Ru 39采樣地俄羅斯卡爾梅克共國亞什庫爾鎮亞什庫爾鎮亞什庫爾鎮亞什庫爾鎮嘎西貢亞什庫爾鎮嘎西貢伊科朝納斯沃茲涅謝諾夫卡乳酸菌數(對數值)/mL-1 8.92±0.01 7.50±0.23 8.19±0.03 7.16±0.01 6.64±0.02 9.05±0.03 8.43±0.02
由表1可以看出,奶酪樣品中乳酸菌數為6.64~9.05 mL-1,平均值為(7.98±0.91)mL-1。研究結果和Bao Qiuhua對甘肅牦牛乳曲拉和四川曲拉的研究結果相近。其平均乳酸菌數分別為(7.9±0.89)mL-1[12]和(7.18±1.49)mL-1[4]。曲拉又稱粗奶酪,與奶酪生產工藝相似。
3.2 乳酸菌的鑒定
3.2.1 菌種DNA提取和16S rRNA擴增
DNA濃度和OD260/280的測定:OD260/280值在1.8~2.0間即為純DNA樣品。將其質量濃度稀釋至100 ng/μL后進行16S rRNA基因擴增。
對乳酸菌分離株16S rRNA基因進行擴增。經1.0%瓊脂糖凝膠電泳檢測后,可以觀察到在大約1 500 bp的位置處有一條清晰明亮的條帶,并且無彌散現象,無明顯非特異擴增現象。
這表明菌株的16S rRNA基因擴增產物可以滿足測序的要求。將16S rRNA基因擴增產物送于上海美吉生物科技有限公司進行測序。所有的16S rDNA序列都與GenBank數據庫中的標準菌株有99%及以上的相似度,根據前人的研究,16S rRNA用于鑒定乳酸菌有很高的可靠性。
3.2.2 系統發育樹的構建和乳酸菌鑒定
將測序獲得的16S rRNA基因序列通過NCBI數據庫中BLAST進行同源序列比對分析,以與模式菌株序列同源性大于98%為種的鑒定閾值,將17株分離株鑒定為乳酸菌的3個屬,20個種。分離株與模式株系統發育樹如圖1所示。
由圖1可以看出,菌株IMAU90028,IMAU90029,IMAU90030,IMAU90032,IMAU90034,IMAU90046,IMAU90048,IMAU90065,IMAU90088,IMAU90150,IMAU90209與標準菌株 Lactobacillusplantarum ATCC14917聚為一類,且同源性為100%,故將其鑒定為Lactobacillus plantarum。菌株IMAU90033、IMAU9 0066與標準菌株Lactobacillus brevis ATCC14869聚為一類,且同源性為100%,故將其鑒定為Lactobacillus brevis。菌株IMAU90215與標準菌株Lactobacillus kefiri LMG9480聚為一類,且同源性為100%,故將其鑒定為Lactobacillus kefiri。菌株IMAU90047與標準菌株Lactobacillus graminis DSM20719聚為一類,且同源性為100%,故將其鑒定為Lactobacillus graminis。菌株IMAU90086,IMAU90087,IMAU90091,IMAU90094,IMAU90210,IMAU90214與標準菌株Lactobacillus paracasei ATCC25302聚為一類,且同源性為100%,故將其鑒 定 為 Lactobacillus paracasei。 菌 株 IMAU90176、IMAU90174與標準菌株Enterococcusdurans ATCC19432聚為一類,且同源性為70%,故將其鑒定為Enterococcus durans。菌株IMAU90155、IMAU9015 6、IMAU90157、IMAU90158、IMAU90212、IMAU9021 3與標準菌株Enterococcus faecium ATCC 19434聚為一類,且同源性為99%,故將其鑒定為Enterococcus faecium。 菌 株 IMAU90067,IMAU90090,IMAU90092,IMAU90093,IMAU90151,IMAU90211與標準菌株Lactobacillus helveticus DSM20075聚為一類,且同源性為100%,故將其鑒定為Lactobacillus helveticus。菌株IMAU90177與標準菌株Leuconostoccitreum ATCC49370聚為一類,且同源性為100%,故將其鑒定為Leuconostoc citreum。菌株IMAU90178與標準菌株Leuconostoc pseudomesenteroides NRIC1777聚為一類,且同源性為69%,故將其鑒定為Leuconostoc pseudomesenteroides。
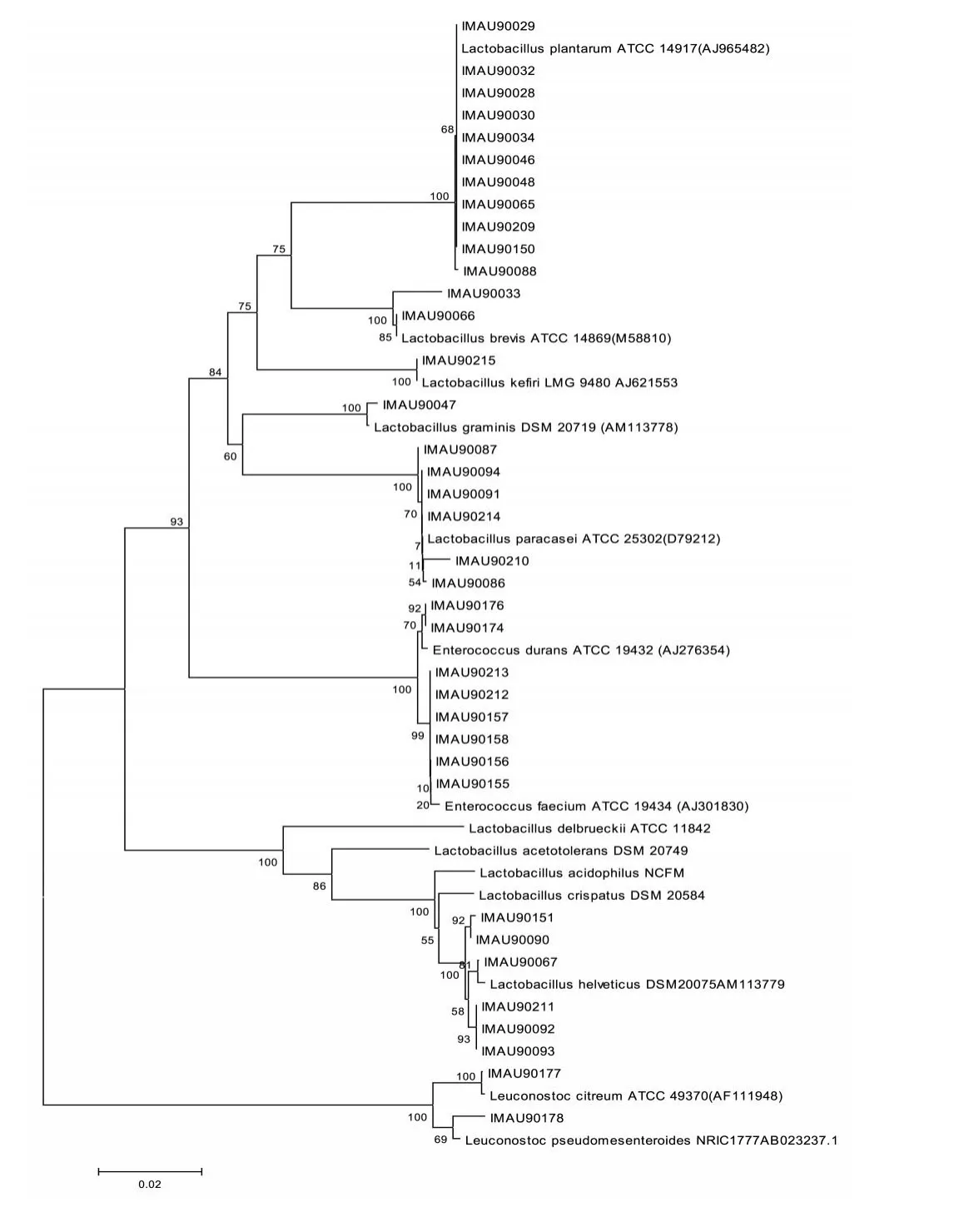
圖1 分離株與其相應模式株16S rRNA系統發育樹
所有分離株鑒定結果和種屬統如表2所示:
本研究的奶酪樣品中乳酸菌的優勢菌群為Lactobacillus plantarum(29.7%),其次分離株數較多的是Lactobacillus paracasei(16.2%)、Lactobacillus helveticus(16.2%)、Enterococcus faecium(16.2%)。不同地區的奶酪樣品中優勢菌群是不同的:Aydemir等所研究的奶酪中優勢菌為Lactobacillus casei和Lactobacillus plantarum[13];Dolci等的研究中Lactobacillus plantarum和Lactobacillus paracasei是Castelmagno PDO奶酪中的優勢菌[14];Coppola R等的研究中Caciocavallo奶酪的優勢菌為Lactobacillus paracasei subsp.Paracasei、Lb.pentosus,Lb.coryneformis subsp.Torquens和 Lb.plantarum.[15];Coppola S等的研究中Mozzarella奶酪的優勢菌為Strep.thermophilus[16];還有的為 Lactobacillus paracasei、Lactobacillus fermentum和Lactobacillus plantarum[17]。不同環境自然發酵的奶酪中優勢菌群是不同的,我們所分離到的Lactobacillus plantarum、Lactobacillus paracasei在奶酪中也是比較常見的。

表2 乳酸菌分離株與模式菌株同源性比對結果
4 結論
7份奶酪樣品中乳酸菌數為6.64~9.05 mL-1(對數值),乳酸菌含量都比較高。7份奶酪樣品中共分出37株菌,分別屬于腸球菌屬、乳桿菌屬、明串株菌屬,共3個屬10個種。乳球菌10株,乳桿菌27株。本研究可以為俄羅斯卡爾梅克地區傳統奶酪中乳酸菌多樣性研究提供原始數據,也可以為以后的乳酸菌篩選與工業生產提供菌株資源。
[1]LIU S,HAN Y,ZHOU Z.Lactic acid bacteria in traditional fermented Chinese foods[J].Food Research International,2011,44(3):643-651.
[2]DE ANGELIS M,CORSETTI A,TOSTI N,et al.Characterization of non-starter lactic acid bacteria from Italian ewe cheeses based on phenotypic,genotypic,and cell wall protein analyses[J].Applied and Environmental Microbiology,2001,67(5):2011-2020.
[3]MARINO M,MAIFRENI M,RONDININI G.Microbiological characterization of artisanal Montasio cheese:analysis of its indigenous lactic acid bacteria[J].FEMS microbiology letters,2003,229(1):133-140.
[4]BAO Q,YU J,LIU W,et al.Predominant lactic acid bacteria in traditional fermented yak milk products in the Sichuan Province of China[J].Dairy Science&Technology,2012,92(3):309-319.
[5]YU J,WANG W H,MENGHE B L G,et al.Diversity of lactic acid bacteria associated with traditional fermented dairy products in Mongolia[J].Journal of dairy science,2011,94(7):3229-3241.
[6]ZHOU J,BRUNS M A,TIEDJE J M.DNA recovery from soils of diverse composition[J].Applied and environmental microbiology,1996,62(2):316-322.
[7]ZHU H,QU F,ZHU L H.Isolation of genomic DNAs from plants,fungi and bacteria using benzyl chloride[J].Nucleic Acids Research,1993,21(22):5279.
[8]SUN Z,LIU W,GAO W,et al.Identification and characterization of the dominant lactic acid bacteria from kurut:The naturally fermented yak milk in Qinghai,China[J].The Journal of general and applied microbiology,2010,56(1):1-10.
[9]YU J,GAO W,QING M,et al.Identification and characterization of lactic acid bacteria isolated from traditional pickles in Sich uan,China[J].The Journal of general and applied microbiology,2012,58(3):163-172.
[10]ALTSCHUL S F,MADDEN T L,SCHAFFER A A,et al.Gapped BLAST and PSI-BLAST:a new generation of protein database search programs[J].Nucleic acids research,1997,25(17):3389-3402.
[11]TAMURA K,DUDLEY J,NEI M,et al.MEGA4:molecular evolutionary genetics analysis(MEGA)software version 4.0[J].Molecular biology and evolution,2007,24(8):1596-1599.
[12]BAO Q H,LIU W J,YU J,et al.Isolation and identification of cultivable lactic acid bacteria in traditional yak milk products of Gansu Province in China[J].The Journal of general and applied microbiology,2012,58(2):95-105.
[13]AYDEMIR O,HARTH H,WECKX S,et al.Microbial communities involved in Kasar cheese ripening[J].Food microbiology,2015,46:587-595.
[14]DOLCIP,ALESSANDRIA V,RANTSIOU K,et al.Microbial dynamics of Castelmagno PDO,a traditional Italian cheese,with a focus on lactic acid bacteria ecology[J].International journal of food microbiology,2008,122(3):302-311.
[15]COPPOLA R,SUCCI M,SORRENTINO E,et al.Survey of lactic acid bacteria during the ripening of Caciocavallo cheese produced in Molise[J].Le Lait,2003,83(3):211-222.
[16]COPPOLA S,BLAIOTTA G,ERCOLINI D,et al.Molecular evaluation of microbial diversity occurring in different types of Mozzarella cheese[J].Journal of Applied Microbiology,2001,90(3):414-420.
[17]PIRAINO P,ZOTTA T,RICCIARDI A,et al.Discrimination of commercial Caciocavallo cheeses on the basis of the diversity of lactic microflora and primary proteolysis[J].Inter national dairy journal,2005,15(11):1138-1149.

