諸氏鯔蝦虎魚轉錄組序列中微衛星標記的初步篩選及特征分析
蔡磊 余露軍 陳小曲 葉惠欣 陳琳 李建軍
(廣東省實驗動物監測所 廣東省實驗動物重點實驗室,廣州 510663)
諸氏鯔蝦虎魚轉錄組序列中微衛星標記的初步篩選及特征分析
蔡磊 余露軍 陳小曲 葉惠欣 陳琳 李建軍
(廣東省實驗動物監測所廣東省實驗動物重點實驗室,廣州510663)
旨在為大規模開發諸氏鯔蝦虎魚微衛星標記,采用高通量測序技術,對諸氏鯔蝦虎魚肝臟轉錄組進行了測序。結果共獲得47 979條Unigenes,利用微衛星查找程序在47 979條Unigenes中共獲得6 225個微衛星位點(12.97%),平均每7.02 kb就出現1個微衛星位點。6 225個微衛星位點由226種重復基序組成,主要分布在三、四和五堿基重復類型中。在數量上,單堿基重復類型微衛星位點最多,占42.49%,二堿基和三堿基重復類型所占比例相似,分別為25.22%和26.27%,四、五、六重復類型較少,合計占6.03%。單堿基重復序列中最多的類型為A/T,二堿基重復序列中以AG/CT重復單元為主,三堿基重復序列中以AGC/TCG為優勢類型。挑選部分二、三和四單元重復類型微衛星序列,共設計76對引物,可穩定擴增出目的條帶的有55對,其中32對具有多態性。結果表明,利用諸氏鯔蝦虎魚轉錄組數據可快速大量開發微衛星標記。
諸氏鯔蝦虎魚;轉錄組序列;微衛星;篩選;分布特征
微衛星DNA,又稱為簡單重復序列(simple sequence repeat,SSR),是一種廣泛分布于原核和真核生物基因組中的遺傳標記,由于其具有共顯性遺傳、多態性豐富、操作簡單和成本低等優點,已在群體遺傳學研究中得到大量應用[1,2]。傳統的微衛星標記開發方法主要是從構建的基因組文庫中富集、回收、克隆微衛星序列,費時費力,且陽性克隆率較低。因此對于基因組背景信息較少的物種,傳統方法在進行微衛星標記的大規模開發方面就受到一定的限制。隨著高通量測序技術的成熟及成本的下降,基于轉錄組數據開發微衛星標記技術的優點正逐漸顯露出來。首先,與基因組測序相比,轉錄組測序費用低、周期短、獲得的信息量足夠豐富可完全滿足標記開發需要。其次,來源于轉錄組的微衛星標記與功能基因緊密連鎖,可為后續基因的定位、性狀關聯分析等提供有用信息。利用轉錄組數據庫開發微衛星標記的方法已在動植物中被大量報道[3,4]。
諸氏鯔蝦虎魚(Mugilogobius chulae)是具有我國特色的一種小型海水魚類,經過近10年比較研究發現諸氏鯔蝦虎魚在各種生物學性狀方面均較具實驗動物的開發潛力[5,6],如個體小、繁殖周期短、繁殖力強、便于實驗室內飼養管理以及對污染物生物毒性敏感等。目前實驗室封閉群已經繁殖到第14代,近交系已培育至第6代,諸氏鯔蝦虎魚的實驗動物化將會是對國際海洋模式魚類空白的填補。目前公共數據庫中關于諸氏鯔蝦虎魚基因組信息資源極其匱乏,且多為線粒體序列,遠不能滿足諸氏鯔蝦虎魚遺傳質量控制和種質資源保護的需求。為更好地了解諸氏鯔蝦虎魚遺傳背景知識,指導諸氏鯔蝦虎魚作為實驗動物在封閉群建群過程中的遺傳質量控制以及近交系近交過程中遺傳純合度檢測,本研究利用Illumina高通量測序平臺對諸氏鯔蝦虎魚肝臟轉錄組進行測序,并對微衛星標記進行初步篩選和分析,以期為諸氏鯔蝦虎魚功能基因及遺傳學研究提供基礎數據。
1 材料與方法
1.1 材料
轉錄組測序以及微衛星引物篩選所用的諸氏鯔蝦虎魚樣本均取自廣東省實驗動物監測所海洋生物保種基地,共20尾,平均體重(0.63±0.15)g,全長范圍在2.6-4.1 cm之間。挑選10尾諸氏鯔蝦虎魚樣本分別取新鮮肝臟組織并混合,液氮冷凍保存,用于總RNA提取。另取10尾樣本固定于無水乙醇中,-20℃保存,用于DNA提取。
1.2 方法
1.2.1 轉錄組測序 參照 Trizol試劑(Invitrogen公司產品)說明書提取肝臟總RNA,利用華大基因科技服務有限公司Illumina HiSeq2000高通量測序平臺進行RNAseq轉錄組測序。用SOAPdenove軟件[7]對轉錄組數據進行從頭組裝獲得大量Unigenes。
1.2.2 微衛星序列的查找及引物設計 使用MISA(MIcroSAtellite identification tool)軟件對組裝的Unigenes進行微衛星序列的查找和篩選,篩選標準為:單堿基重復次數在12次或12次以上,兩堿基在6次或者6次以上,三和四堿基在5次或者5次以上,五和六堿基在4次或者4次以上。用Primer 3.0批量設計程序設計微衛星引物,設計好的引物送上海生工生物工程有限公司合成。
1.2.3 微衛星引物的篩選 微衛星引物篩選所用諸氏鯔蝦虎魚樣本共10尾,按照廣州欣研生物科技有限公司生產的組織DNA提取試劑盒進行樣品基因組DNA的提取,并檢測純度和濃度。PCR擴增總體系為25 μL,包括Premix TaqTMVersion 2.0(TaKaRa)12.5 μL,上下游引物(10 pmol/μL)各1 μL,DNA模板200 ng,無菌去離子水補齊至25 μL。PCR反應程序為:94℃預變性4 min;94℃ 40 s,退火(溫度依據引物而定)30 s,72℃ 30 s,30個循環;72℃延伸10 min。PCR產物在濃度為10%的非變性聚丙烯酰胺凝膠中分離,硝酸銀染色。用NR、Swiss-Port、KEGG和COG蛋白質數據庫對含有多態性的序列進行基因注釋。
2 結果
2.1 諸氏鯔蝦虎魚轉錄組序列中微衛星的數量和分布特點
利用SOAPdenove軟件對轉錄組數據進行組裝,共獲得47 979條Unigenes,總長度為43.7 Mbp,平均長度911 bp,N50達到1 658 bp。對獲得的Unigenes進行微衛星標記的查找,共得到6 225個微衛星位點(12.97%),分布在5 868個Unigenes上,其中有297條Unigenes含有兩個及以上微衛星位點(5.1%),諸氏鯔蝦虎魚轉錄組序列中平均每7.02 kb就出現1個微衛星位點。最短的微衛星序列為12bp,最長的為393 bp。重復單元類型中1-6堿基重復類型均有出現,且分布數量不均。
2.2 諸氏鯔蝦虎魚轉錄組序列中微衛星的重復類型及特征
諸氏鯔蝦虎魚轉錄組序列中微衛星重復基序共226種,1-6堿基重復類型數量分別為3、11、59、61、53和39個。其中單堿基重復類型最多,有2 645個,占42.49%,其次為三堿基和二堿基重復,分別為1 635和1 570個,占26.27%和25.22%,四、五和六堿基重復類型數量較少,總共占6.03%。單堿基重復序列中除1個為C/G重復單元外,其余全為A/T重復單元,二堿基重復序列中以AG/CT和AC/ GT重復單元為主,分別占59.8%和28.5%,三堿基重復序列中以AGC/TCG、AAG/CTT和AGG/CCT為優勢類型,分別占32.4%、14.6%和13.5%,其余重復單元各種重復類型分布較均勻,具體分布見圖1。在重復次數上,單堿基微衛星的核心重復次數主要集中在12-17次,二、三堿基核心序列重復次數主要集中在5-8次,四堿基以5和6次重復為主,五和六堿基以4次重復為主,具體數據見表1。

圖1 諸氏鯔蝦虎魚轉錄組中不同微衛星重復單元類型分布
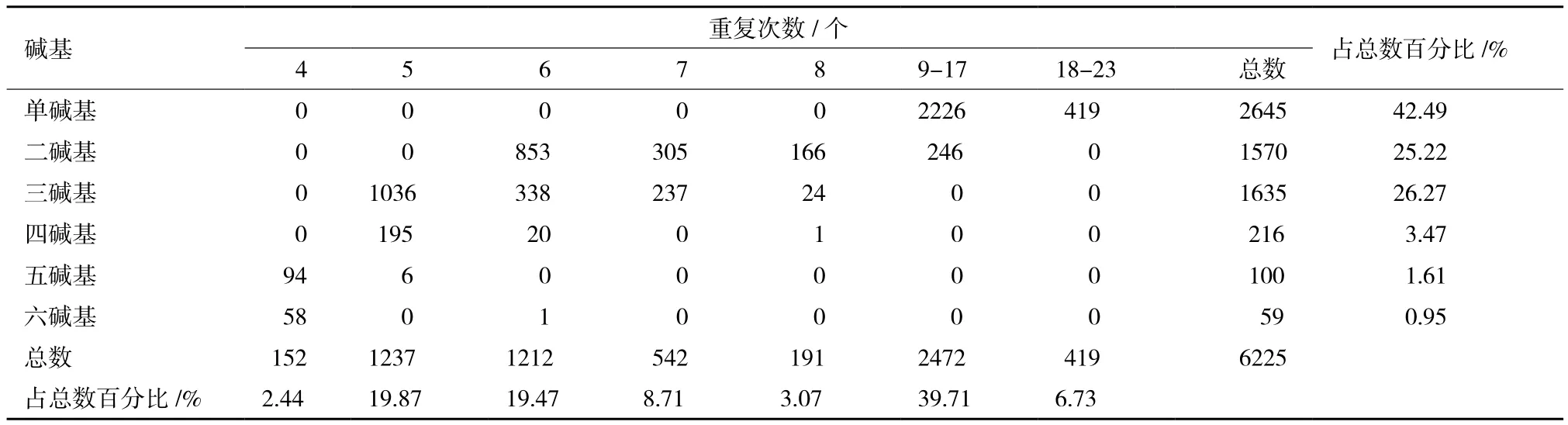
表1 諸氏鯔蝦虎魚轉錄組中微衛星重復單元的數量和類型
2.3 微衛星引物的設計及多態性引物的初步篩選
利用Primer3對獲得的微衛星序列進行引物設計,共設計引物2 671對,挑選部分二、三和四單元重復類型微衛星引物共76對送出合成,其中二堿基重復次數≥9次,三堿基重復次數≥7次,四堿基重復次數≥6次。以10個諸氏鯔蝦虎魚野生群樣本基因組DNA為模板,對76對引物進行了PCR擴增。可穩定擴增出目的條帶的有55對,其中有32對具有多態性,23對為單態,部分擴增結果見圖2。初步篩選獲得的具有多態性的32個微衛星位點及引物序列,見表2,相關序列已上傳至美國國家生物技術信息中心(National Center for BiotechnologyInformation,NCBI)。
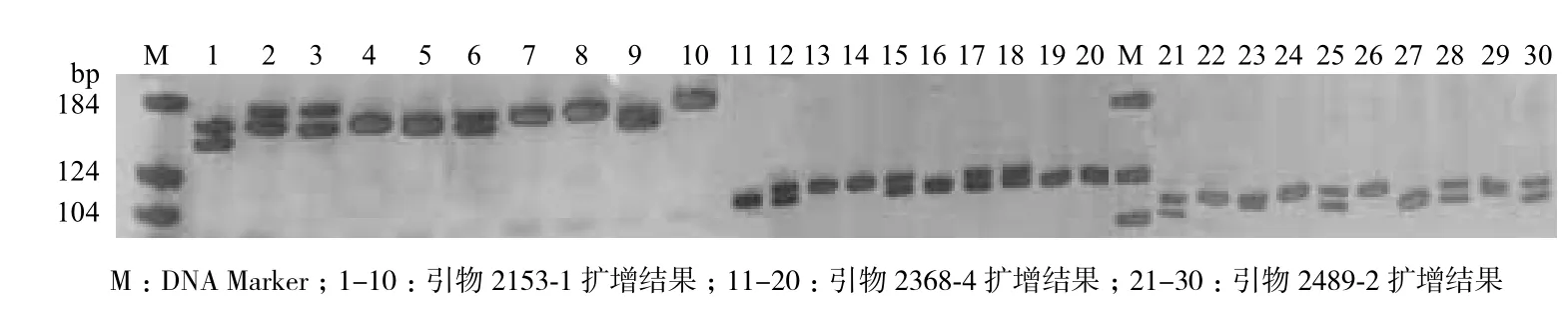
圖2 引物2153-1、2368-4和2489-2在10尾諸氏鯔蝦虎魚野生群體中的擴增結果
2.4 多態性微衛星標記的功能注釋
利用NR、Swiss-Port、KEGG和COG蛋白質數據庫對32對多態性微衛星序列進行基因功能注釋,共有20個微衛星位點獲得注釋,涉及到20個已知的功能基因,包括到轉錄因子、受體、激酶和一些細胞組成成分等重要功能基因(表2)。
3 討論
本研究在47 979條,總長度為43.7 Mbp 的Unignges中共發現微衛星位點6 225個,平均每7.02 kb就出現1個微衛星位點,發生頻率為12.97%。與其他海洋魚類相比,諸氏鯔蝦虎魚轉錄組中大于二堿基重復微衛星標記的出現頻率介于中間狀態(7.46%),比斑點叉尾鮰(Ictalurus punctatus)(11.2%)[8]和紅鰭東方鲀(Fugu rubripes)(11.5%)[9]低,但又高于真鯛(Chrysophrys major)(4%)[10]和大黃魚(Pseudosciaena crocea)(4.24%)[11],與牙鲆(Paralichthys olivaceus)(7.95%)[12]相似。在出現密度上,與其他魚類的差異較小,如鮸魚(Miichthys miiuy) 為1/6.04 kb[13]、 鯽 魚(Carassius auratus)為1/6.39 kb[14], 牙 鲆(Paralichthys olivaceus) 為1/7.9 kb[12]。這種出現頻率和密度上的差異可能與微衛星查找所選用數據量的大小、數據拼裝的質量、組織來源以及物種間差異有關。
一般認為微衛星標記的多態性與重復單元的重復次數呈正相關[15]。對本研究獲得的微衛星序列統計發現,除單堿基重復微衛星外,其余微衛星重復次數范圍在5-8次之間,比來源于基因組的微衛星重復次數低。推測可能是由轉錄組微衛星主要來自編碼區導致,與基因組中非編碼序列相比,編碼區序列受到選擇壓力更大,相對不易發生變異[16]。
在微衛星重復類型上,除單堿基微衛星重復類型外,多數物種中是以二堿基重復類型為主[17],本研究發現諸氏鯔蝦虎魚轉錄組序列微衛星重復類型中以三堿基重復類型為最多,結果與之存在差異。這種現象在大黃魚、鮸魚和縊蟶(Sinonovacula constricta)中也見報道[11,13,18]。由于轉錄組序列主要來源于外顯子,自然選擇機制對轉錄區的三核苷酸基序表現出積極選擇作用,導致在編碼區由于受重大突變壓力的影響而存在豐富的三核苷酸重復序列[19,20]。在重復類型分布上,單堿基、二堿基和三堿基均表現出一定的偏倚性,單堿基重復類型中除發現1個C/G類型外,其余均為A/T類型,在二堿基重復序列中,AG/CT占主要優勢,三堿基重復類型中最多的是AGC/TCG,這種二、三重復單元的重復類別在不同物種間差異較大[13,14,21],這種重復單元數量的偏倚以及類型的差異性可能與物種間差異性有關[22]。另外,在二堿基重復類型中還檢測到18個比較罕見的GC/GC類型。
考慮到標記多態性問題,在進行微衛星引物篩選時,為避免盲目,盡量選取重復單元重復次數較高的序列進行篩選。本研究挑選部分二堿基重復次數≥9次、三堿基重復次數≥7次、四堿基重復次數≥6次的序列,共設計76對微衛星引物,以10個諸氏鯔蝦虎魚野生保種群體樣本基因組DNA為模板,對76對引物進行了初步篩選。其中能穩定擴增出目的片段的引物有55對,剩余21對未擴增或者擴增非目的片段。55對有效擴增引物中有32對具有多態性,多態位點比例達到42.67%。分布上僅見于二和三堿基重復類型,四堿基重復類型序列沒有發現多態位點,可能是選用的位點數量較少導致。通過對獲得的32個多態性微衛星位點進行蛋白庫比對,共有20個位點獲得注釋,這種位于外顯子區域的微衛星位點更適合用于功能基因的定位和表達調控分析。初步開發的32個具有多態性的微衛星位點,為諸氏鯔蝦虎魚種群遺傳結構的分析、封閉群和近交系群體的遺傳質量監測奠定了基礎。
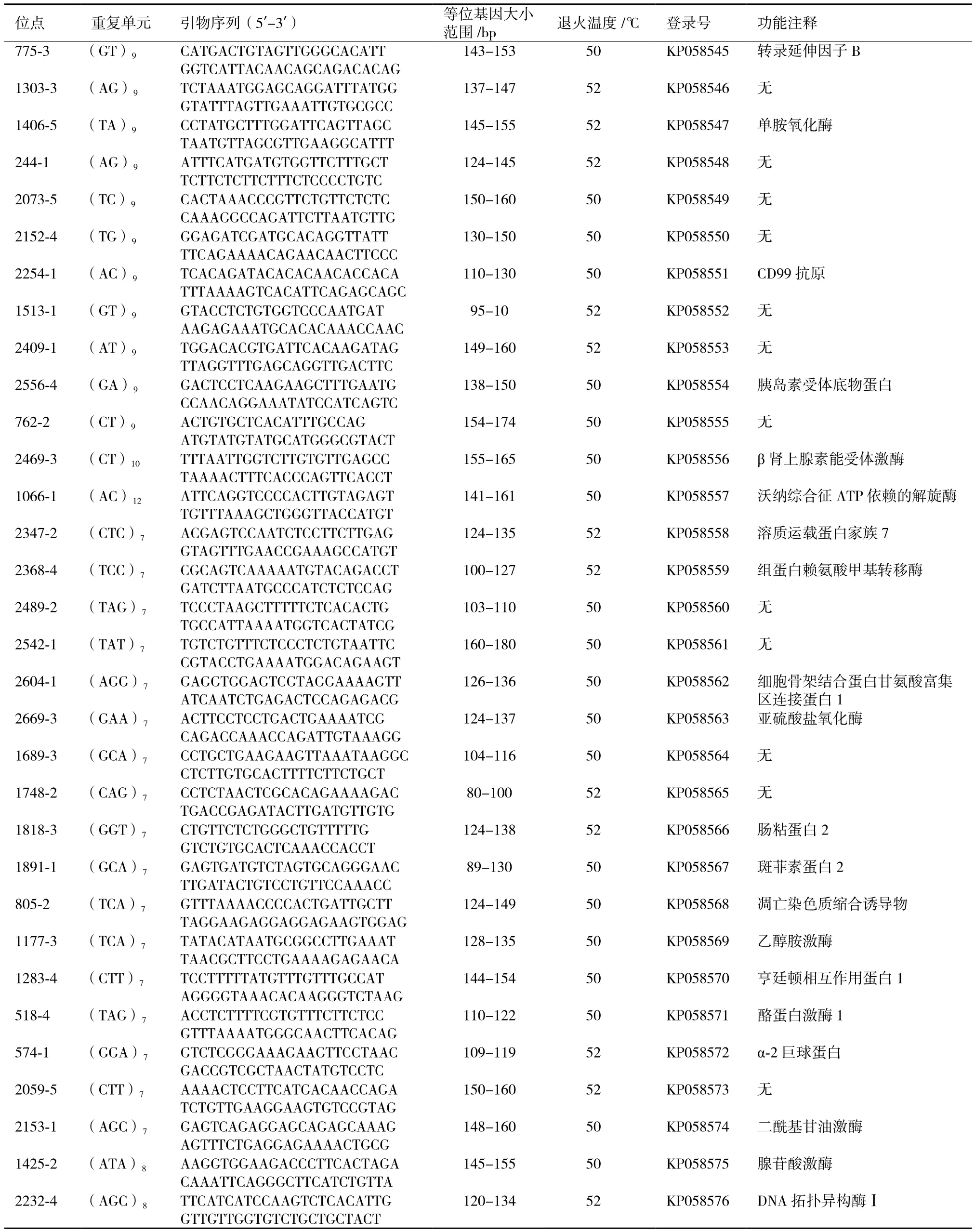
表2 諸氏鯔蝦虎魚32個多態性微衛星標記特征
4 結論
本研究利用高通量測序技術首次對諸氏鯔蝦虎魚肝臟轉錄組進行了測序,共獲得了47 979條Unigenes。在47 979條Unigenes中共查找到6 225個微衛星位點,主要以單堿基、二堿基和三堿基重復類型為主。挑選部分二、三和四單元重復類型微衛星序列,共設計76對引物,可穩定擴增出目的條帶的有55對,其中32對具有多態性。
[1] Dor L, Shirak A, Gorshkov S, et al. Construction of a microsatellitesbased linkage map for the white grouper(Epinephelus aeneus)[J]. Genomes, 2014, 45(5):699-708.
[2] Grattapaglia D, Amaral D, Santos G. Performance of microsatellites for parentage assignment following mass controlled pollination in a clonal seed orchard of loblolly pine(Pinus taeda L.)[J]. Tree Genetics & Genomes, 2014, 10(6):1631-1643.
[3] Norrell AE, Craw ley D, Jones KL, et al. Development and characterization of eighty-four microsatellite markers for the red snapper(Lutjanus campechanus)using Illumina paired-end sequencing[J]. Aquaculture, 2014, 430:128-132.
[4] Vukosavljev M, Esselink GD, Westende WPC, et al. Efficient development of highly polymorphic microsatellite markers based on polymorphic repeats in transcriptome sequences of multiple individuals[J]. Molecular Ecology Resources, 2014, 15(1). DOI:10.1111/1755-0998.1289.
[5] 李建軍, 陳小曲, 林忠婷, 等. 諸氏鯔蝦虎魚的形態與生長特性分析[J]. 實驗動物與比較醫學, 2012, 32(4):334-340.
[6] 李建軍, 吳美慧, 葉慧欣, 等. 不同發育期諸氏鯔蝦虎魚對鉆井液的敏感性比較[J]. 中國比較醫學雜, 2013, 23:48-51.
[7] Luo RB, Liu BH, Xie YL, et al. SOAPdenovo2:an empirically improved memory-efficient short-read de novo assembler[J]. GigaScience, 2012, 1(18):1-6.
[8] Serapion J, Kucuktas H, Feng JN, et al. Bioinformation mining of type I microsatellites from expressed sequence tags of channel catfish(Ictalurus punctatus)[J]. Marine Biotechnology, 2004, 6(4):364-377.
[9] Edwards YJ, Elgar G, Clark MS, et al. The identicication and characteriction of microsatellites in the compact genome of the Japanese puffer fish, Fugu rubripes:Perspectives in functional and comparative genomic analysis[J]. Journal of Molecular Biology,1998, 278:843-854.
[10] Chen SL, Liu YG, Xu MY, et al. Isolation and characterization of polymorphic microsatellite loci from an EST library of red seabream(Chrysophrys major)and cross-species amplification[J]. Molecular Ecology Notes, 2005, 5:215-217.
[11] 謝芳靜, 張子平, 鄒志華, 等. 大黃魚EST微衛星標記初步篩選[J]. 福建水產, 2011, 33(5):9-14.
[12] 陳松波, 龔麗, 劉海金. 牙鲆EST資源的SSR信息分析[J].東北農業大學學報, 2010, 41(10):82-86.
[13] 孫典巧, 孫悅娜, 王日昕, 等. 鮸魚EST序列中微衛星標記的初步篩選及特性分析[J]. 水生生物學報, 2011, 35(5):753-760.
[14] 楊曦. 鯽魚(Carassius auratus)表達序列標簽資源的SSR構成與分布分析[J]. 生物技術通報, 2012(11):139-143.
[15] Weber JL. Informativeness of human(dC-dA)n·(dG-dT)n polymorphisms[J]. Genomics, 1990, 7(4):524-530.
[16] 懂迎輝, 吳國星, 姚韓韓, 等. 泥蚶34個EST-SSR標記的開發及在格粗飾蚶的通用性檢測[J]. 水產學報, 2013, 37(1):70-77.
[17] 郭文久. 微衛星在基因組上的分布與功能及其計算方法初步研究[M]. 西安:陜西科學技術出版社, 2009:5-6.
[18] 劉博, 邵艷卿, 滕爽爽, 等. 縊蟶(Sinonovacula constricta)EST-SSR分布特征及引物開發利用[J]. 海洋與湖沼, 2012,43(1):133-137.
[19] Blanca J, Ca?izares J, Roig C, et al. Transcriptome characterization and high throughput SSRs and SNPs discovery in Cucurbita pepo(Cucurbitaceae)[J]. BMC Genomics, 2011, 12:1-15.
[20] Garg R, Patel RK, Tyagi AK, et al. De novo assembly of chickpea transcriptome using short reads for gene discovery and marker identification[J]. DNA Research, 2011, 18(1):53-63.
[21] Kantety RV, Rota ML, Matthews DE, et al. Data mining for simple sequence repeats in expressed sequence tags from barley, maize,rice, sorghum and wheat[J]. Plant Mol Biol, 2002, 48:501-510.
[22] 許曉軍, 張海琪, 張超, 等. 中華鱉表達序列標簽源中的微衛星信息分析[J]. 經濟動物學報, 2013, 17(1):15-18.
(責任編輯 馬鑫)
A Prelim inary Screening and Characteristic Analysis of M icrosatellite M arkers from Transcriptome Sequences in Mugilogobius chulae
Cai Lei Yu Lujun Chen Xiaoqu Ye Huixin Chen Lin Li Jianjun
(Key Laboratory of Guangdong Laboratory Animals,Guangdong Laboratory Animals Monitoring Institute,Guangzhou510663)
In order to explore microsatellite markers in large-scale, the liver transcriptome of Mugilogobius chulae was sequenced by highthroughput sequencing technology. A total of 6 225(12.97%)microsatellite markers were screened from 47 979 Unigenes by microsatellite software, averagely 1 microsatellite marker in every 7.02 kb. The 6 225 microsatellite markers consisted of 226 repeat motifs, which mainly distributed in tri-nucleotide, quad-nucleotide and penta-nucleotide repeats. The mono-nucleotide repeats were of the most by number, accounting for 42.49% of all the microsatellite markers, the di-nucleotide repeats and tri-nucleotide repeats had the similar proportion(25.22% and 26.27% of all the markers, respectively), the rest were the quad-nucleotide, penta-nucleotide and hexa-nucleotide repeats, together accounting for 6.03%. A/T, AG/CT and AGC/TCG were the most frequent motifs in mono-nucleotide, di-nucleotide and tri-nucleotide repeats, respectively. In this research, 76 pairs of primers were designed and synthesized, by 55 pairs of them the target bands were successfully amplified, and 32 loci were of polymorphism. The results suggested that it was feasible to explore a number of microsatellite markers quickly based on the transcriptome sequences of M. chulae.
Mugilogobius chulae;transcriptome sequence;microsatellites;screening;distribution characteristic
10.13560/j.cnki.biotech.bull.1985.2015.09.020
2014-12-13
廣東省中國科學院全面戰略合作項目(2011B090300099),廣東省科技計劃項目(2012A020602008),國家科技支撐計劃項目(2013BAK11B902)
蔡磊,男,碩士研究生,研究方向:水生實驗動物遺傳質量控制;E-mail:cailei17@163.com
李建軍,男,高級工程師,研究方向:水生實驗動物及生態毒理學;E-mail:lijianjun1125@126.com

