植物中miRNA及其靶基因的檢測與鑒定方法
位明明++李維國

摘 要 miRNAs(microRNA,微RNA)作為植物體內一類重要的內源性非編碼小分子RNA,能夠顯著調控細胞發育的多個生物學過程,對植物生長起重要調控作用。諸多研究表明:在植物生長發育過程中miRNA的表達具有組織特異性和時空特異性。因此,檢測和分析不同組織或樣品中miRNA及其靶基因的種類與表達特征,可為研究miRNA的生物學功能提供重要信息和依據。本文綜述了近年來植物中miRNA及其靶基因的檢測與鑒定方法及其存在的優缺點,為進一步開展miRNA及其靶基因的功能研究提供借鑒和參考。
關鍵詞 miRNA ;靶基因 ;檢測方法
分類號 Q522
Detection and Identification of miRNAs and Their Target Genes in Plants
WEI Mingming LI Weiguo
(State Centre for Rubber Breeding / Ministry of Agriculture Key Laboratory of Biology and Genetic Resources of Rubber Tree / Rubber Research Institute, CATAS,Danzhou,Hainan 571737)
Abstract miRNAs, an important class of endogenous non-coding small RNA molecules in plants, can significantly regulate multiple biological processes of cell development and play an important role in plant growth regulation. Many studies show that the expression of miRNA in the process of plant growth has spatial and temporal specificity. Therefore,detection and analysis the characterization of miRNAs and their target genes in different tissues or samples can provide important information and basis for studying the biological function of miRNA. This paper discusses the detection and identification methods of miRNAs and their target genes in plants, as well as the advantages and disadvantages of different methods in recent years,so to provide the reference and instruction for further studying the function of miRNAs and their target genes.
Keywords miRNA ; target gene ; detection methods
對于小分子RNA的關注,始于2000年6月人類基因組計劃的完成,該計劃的完成向人類揭示了高等哺乳動物基因組中蛋白質編碼序列不足2%,而98%以上的序列都是非蛋白質編碼DNA[1]。此后,隨著多種模式植物基因組測序的完成,科學家發現,非編碼序列占絕大多數的情況同樣存在于植物基因組中。起初許多學者將此類非蛋白編碼基因認為是“垃圾DNA”,但隨后研究發現,生物細胞所表達的RNA中,除了前人熟悉的tRNA、rRNA、mRNA外,還蘊藏著大量不編碼蛋白質的非編碼RNA分子(noncoding RNA,ncRNA),這些在生物體基因組上廣泛分布的非蛋白質編碼區并非“垃圾DNA”,而是包含了大量轉錄調控元件的非編碼RNA基因[2],它們能夠調控真核細胞發育的多個方面,如影響功能基因的表達、調控細胞周期和個體發育等多種行為[3]。ncRNA的發現,打破了長期以來對RNA僅限于DNA的“轉錄信使”這一角色的認識,使科學家重新審視對細胞功能及其進化的認識。非編碼RNA分子在多個物種中不斷被發現和鑒定,揭示出了生物體內存在著一個巨大的且不為人知的“非編碼RNA世界”。同時,基于不同物種體內非編碼RNA分子的研究也拓生出了一門新的科學——RNA組學(RNomics)。目前,RNA組學研究已成為后基因組學時代的一個重要科學前沿[4],尤其是微RNA(microRNA,miRNA)的鑒定和功能研究,已成為繼小干擾RNA(short interfering RNA,siRNA)之后新的研究熱點之一。本研究對miRNA的驗證方法、miRNA靶基因的鑒定方法進行了概述,為進一步開展植物miRNA的功能研究提供借鑒和參考。
1 miRNA研究進展
1.1 miRNA的發現
miRNA于1993年首次在秀麗新小桿線蟲(caenorkabditi solegans)中被發現,當時研究人員在對線蟲突變體進行遺傳分析時,發現了一類長度為22個核苷酸的小分子RNA,它們能夠調控細胞發育的時序,并將其命名為lin-14 RNA[5],進一步研究發現lin-14 RNA雖不編碼蛋白質,卻能與靶基因 lin-14的3′非翻譯區(3′untranslated region,3′UTR)以堿基互補配對的方式結合,并抑制Lin-14基因蛋白的翻譯,從而調控線蟲的細胞發育由第一期向第二期轉化。然而,當Lin-14 RNA首次被發現時,并未引起科研人員太多的關注,直到2000年另一個類似的具有細胞時序調節功能的小分子RNA——let-7 RNA被發現后,這些非編碼RNA才逐步得到人們的關注[6]。由于lin-14 RNA和let-7 RNA具有顯著的細胞時序調控功能,起初被命名為小時序RNA(small temporal RNA,stRNA)。隨著研究的深入,在人和小鼠等哺乳動物中同樣發現了大量具有轉錄后調節功能的小分子RNA。2001年世界不同國家的科研人員將這一類具有轉錄后調節功能的小分子RN再統一命名為miRNA[7-9]。
植物中miRNA于2002年首次在擬南芥小分子RNA文庫中被發現。盡管植物中小分子RNA發現的時間比動物晚了將近10 a,但植物中小分子RNA的鑒定方法要比動物中完善。目前,不僅在擬南芥和水稻等模式植物中發現和驗證了大量的miRNA,而且在其它植物中也發現和驗證了數量可觀的miRNA。隨著生物體內發現的miRNA家族成員的不斷增多,為了更好地對不同物種中發現的miRNA進行登記和命名,國際上建立了miRNA文庫—miRBase[10-11]。截至2015年5月,在miRNA數據庫 (http:microrna.sanger.ac.uk/sequenees/)中注冊提交的miRNA成熟序列已達28 654條,極大地豐富了人們對生物體內非編碼小分子RNA的認識。
1.2 植物miRNA的生成過程
研究發現大部分植物miRNA在基因組上具有一個獨立的轉錄單位,只有少數miRNA在基因組上呈串聯重復存在,在轉錄過程中由一個轉錄單位可加工形成多個miRNA。miRNA在植物體內的生成過程是一個復雜的、涉及多種酶和功能蛋白參與的過程[12],具體步驟如圖1所示:首先植物基因組上具有特定位點的MIR基因,在RNA聚合酶的作用下轉錄成一個或多個較長的初級轉錄產物(pri-miRNA),然后初級轉錄物在Drosha酶的作用下[13],進一步加工形成一個具有特定二級莖環(stem-loop)結構的次級轉錄物(pre-miRNA),這一系列過程是在細胞核內完成的;接著pre-miRNA要在nuclear export的作用下從細胞核運輸到細胞質中,由Dicer酶通過兩步法進行酶切加工,隨后酶切產物在HYL1(HYPONASTICLEAVES1)、HEN1(HUAENHANCER1)、HST(HASTY)等蛋白的作用下,形成成熟的miRNA[14-15]。此外,成熟的單鏈miRNA必須與Argonaute(AGO)蛋白相互作用形成一個RNA誘導沉默復合物(RNA-induced silencing complex,RISC),才能與目標mRNA結合而發揮作用[16-17]。
1.3 miRNA的特點
miRNA是生物體內一類5′端帶磷酸基團,3′端帶羥基基團的內源性小分子RNA。在生物體內miRNA是由非蛋白編碼基因轉錄物形成的莖環結構加工而來,并以堿基互補配對的方式靶向mRNAs,進而影響目標mRNAs的翻譯和穩定性。從目前的研究結果來看,miRNA主要有以下特征:(1)miRNA廣泛存在于生物體內。不僅在模式植物擬南芥[18]和水稻[19]中發現了大量的miRNA,而且在一些重要農作物如玉米[20]、小麥[21]、棉花[22]、及大豆[23]中發現了大量的miRNA,甚至在苔蘚、蕨類等低等植物中也發現了大量的miRNA存在[24]。(2)miRNA沒有開放閱讀框(ORF)及蛋白質編碼基因。在動植物中絕大多數miRNA產生于基因間隔和非蛋白編碼區,只有極少數miRNA產生于基因的啟動子區,它們是由不同于mRNA的轉錄單元表達產生的[25]。(3)miRNA具有典型的長度范圍,長度約為20~24 nt。大多數植物miRNA的長度都集中在21~23 nt,但miRNA的前體長度變化較大,有時長度甚至超過1 kb[26-27]。(4)不同物種中miRNA基因位點通常成簇存在。如研究發現,一些miRNA在生成過程中通常使用一個共同的前體結構,即來源于物種基因組上的同一個miRNA基因簇,并由該前體結構加工形成多個不同的成熟miRNA[28]。(5)miRNA具有典型的序列保守性。與蛋白編碼基因相比,miRNA在進化上更具保守性。如在擬南芥中發現的42個miRNA家族也廣泛存在于水稻、玉米和高粱等其它作物中[29]。同時,miRNA的序列保守性,也為從其它物種中鑒定并克隆miRNA提供了方便。(6)miRNA的表達具有時序性和組織特異性的特征[30-31]。miRNA在不同組織或器官中的表達量差異很大,在同一器官的不同發育時期表達量也有很大差異,表明在生物體的不同發育階段,需要開啟和關閉不同的miRNA來調控其發育進程。(7)大多數miRNA的成熟體都是從前體5′端或3′端的一條臂上加工而來,只有少數miRNA的成熟體是由其前體的兩條壁上同時加工產生[32]。(8)成熟miRNA的 5′端有一個磷酸基團,3′端為羥基,且miRNA的起始堿基多為尿嘧啶核苷酸[33-34]。(9)與動物miRNA的加工和成熟過程需要Drosha、Dicer和Exportin-5等蛋白所不同的是,植物miRNA的生成過程必須經過Dicer-likel(DCL1)、Hasty(HST)、Hyponastie leaves l(HYLI),以及Hua enhancerl(HENI) 等蛋白的加工過程[35-37],這些蛋白在植物miRNA的加工和成熟過程中起著至關重要的作用,它們可直接影響植物體內miRNA的生成、種類、以及分布。(10)大多數植物miRNA與其靶基因幾乎完全互補,而動物miRNA與其靶基因則不完全配對[38]。此外,植物miRNA與靶基因的結合位點不僅限于靶基因的3′非翻譯區(untranslated region,UTR),還可以位于轉錄區域[39-40]。植物中miRNA及靶基因的高度互補性也為相關miRNA的靶基因鑒定和生物信息學預測提供了很大便利。
2 miRNA的鑒定方法
前人研究表明,在哺乳動物中miRNA的異常表達與癌癥[41]、神經紊亂[42]、以及心臟疾病的發生密切相關[43-44],如在大腸癌和乳腺癌細胞中,miRNA的表達量較正常組織細胞有較大改變,人們推測miRNA可能在癌變過程中起到原癌基因和抑癌基因的作用[45]。在植物體內miRNA能夠顯著調控其多個生物學進程,對植物的生長發育至關重要。如與miRNA代謝及其作用機制相關的幾個重要基因的突變體,均體現出發育過程中多向性的缺陷[46-47]。因此,對生物體不同組織或樣本中miRNA的表達特征進行檢測和分析,將有助于人們進一步了解相關miRNA與生物體發育的關系,并為相關miRNA的功能研究提供信息和依據。
由于成熟miRNA具有片段小、細胞中表達水平普遍偏低、且同一miRNA家族成員間僅有1-2個堿基的差別等特點,這些不利因素給miRNA的檢測帶來了較大困難和挑戰。盡管如此,前人還是根據miRNA的表達特征發明了以下5種miRNA檢測方法:Northern blotting法[48]、微陣列法[49]、克隆和測序法[50]、以及實時熒光定量PCR法[51]。根據miRNA的檢測過程中是否需要擴增樣本,又可將以上5種方法大體歸為兩類:一類是不需要樣本擴增的探針直接雜交法,如Northern blotting法;另一類是基于樣本miRNA擴增的方法,如實時熒光定量PCR法。總之,無論何種檢測方法,一種理想的miRNA定量檢測方法都應具有靈敏度高、特異性強、通量高和成本低等優點,才能適應生物學的發展需求。
2.1 基于探針雜交技術的Northern blotting法
基于探針雜交技術的Northern blotting法是目前最為經典的一種miRNA檢測方法,該方法可以直接對樣品中的miRNA進行檢測,而不需要對樣本中miRNA進行預擴增。它的基本原理是先將標記過的miRNA探針固定在尼龍膜上,然后再將樣本RNA與經過標記的探針進行雜交,最后洗滌多余的雜交探針后進行信號檢測。該方法在miRNA的檢測中最為普遍,其優點是可同時對多個不同組織中的miRNA進行檢測,且檢測成本較低。由于經典的Northern blotting方法是基于固相探針雜交技術,所以該檢測方法最大的缺點是雜交速度慢、靈敏度低;同時,該方法在檢測過程需要耗費大量的RNA樣本,對于那些RNA產率很低和容易降解的樣品不太實用。
為了克服Northern blotting探針雜交法特異性差和靈敏度低的缺點,人們發明了用鎖核酸(LNA)修飾的雜交探針來取代傳統探針的方法,即改進的Northern blotting方法[52]。由于LNA是一類寡核苷酸衍生物,它對核酸分子具有較高的親和性,可滲入到核酸序列的任何位置,因而可以提高LNA探針與目標miRNA分子結合后的雙鏈熱穩定性,使得檢測的靈敏度和特異性顯著提高[53]。
2.2 基于微陣列芯片的高通量檢測方法
雖然傳統的Northern blotting法在miRNA檢測中較為常見,但該方法操作步驟繁瑣,不能同時對樣品中多個不同的miRNA進行檢測分析,其miRNA的檢測通量明顯不夠。為了克服這一缺點,人們又發明了miRNA微陣列芯片技術(Microarray),它可以在同一塊芯片上固定多個與miRNA互補的探針,通過對樣本中相關miRNA進行雜交后,最后進行相關miRNA的信號檢測[54]。盡管該方法實現了miRNA的高通量檢測分析,但該方法只能檢測小RNA數據庫中已經提交的miRNA序列,不能對樣品中新的未知miRNA進行檢測,導致樣品中一些miRNA表達信息的缺失,這是該方法的顯著缺陷。
2.3 克隆測序法
基于探針技術的Northern blotting法和微陣列芯片法都是對已知序列的miRNA進行定量檢測。而克隆測序法則可以對樣品中各種不同類型的miRNA進行檢測分析。克隆測序法的基本原理是:首先富集樣品中的小分子RNA,然后通過反轉錄構建樣品相關的小分子RNA的cDNA文庫,接著對cDNA文庫進行PCR擴增,最后將PCR擴增產物連接到表達載體上進行克隆測序,并根據文庫中序列出現的豐度對相關miRNA進行定量分析。由于克隆法在測序前必須對樣本進行細菌克隆,然后再對每一個克隆進行測序,因而需要消耗較大的初始樣本量(幾百微克RNA),并投入大量的人力和財力[55]。此外,基于克隆測序的方法通常是Sanger測序法,不僅測序效率低,而且費時費工,這些都給深入研究miRNA的功能帶來了諸多不便。
2.4 實時定量PCR法
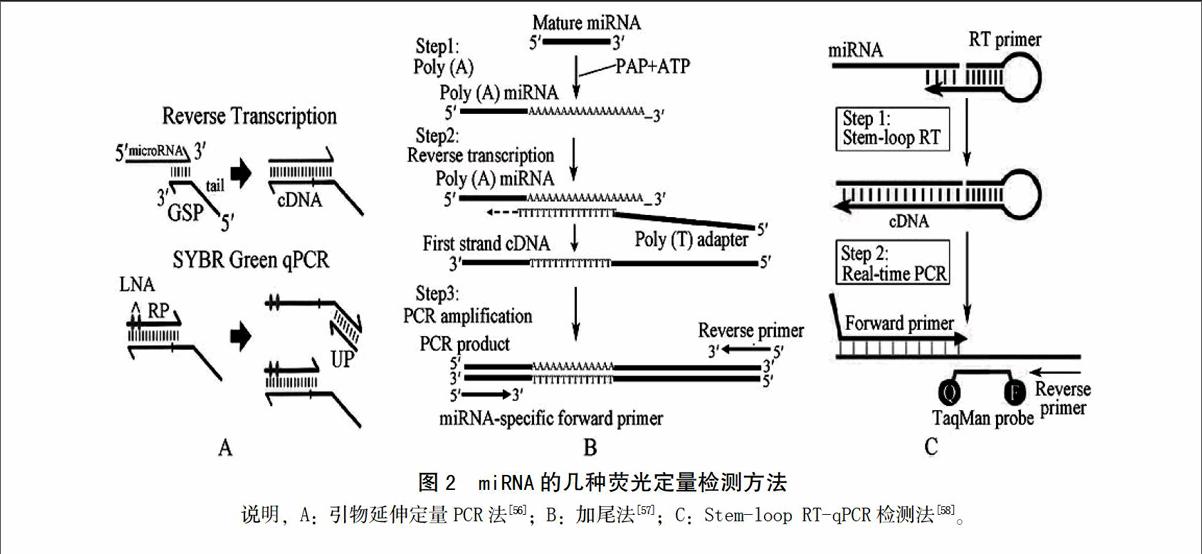
前面幾種方法在檢測過程中都不需要對樣本進行擴增,而實時定量PCR法則需要擴增樣本。目前采用實時定量PCR對miRNA進行定量檢測主要有以下3種方法:第一種方法稱之為引物延伸定量PCR法(PE-qPCR),它的基本原理是在常規引物延伸技術(PE)的基礎上,用一個加尾的特異性引物將miRNA反轉錄成cDNA,然后利用一個鎖核酸(LNA)修飾的miRNA特異性反向引物和一個加尾的通用引物序列,對相關miRNA進行PCR擴增[56];第二種方法稱之加尾法,它的基本原理是先用poly(A)聚合酶對樣品總RNA進行處理,使得樣品中miRNA的3′端加上poly(A)尾巴,然后對其進行反轉錄,使得cDNA序列加上poly(T)接頭,最后再用通用引物和miRNA序列特異引物序列,進行miRNA的PCR擴增[57]。由于該方法在PCR擴增前,需要對miRNA進行加尾反應,因此該方法被稱為加尾法;第三種方法稱之為stem-loop RT-qPCR檢測法,其原理是首先利用一條特殊的莖-環狀引物將miRNA反轉錄成cDNA雙鏈,再利用與miRNA互補的特異性正反向引物和探針,對目標miRNA進行定量檢測。
利用以上3種方法對miRNA進行PCR擴增時,主要采用2種信號檢測模式,即熒光染料法和探針法。利用熒光染料法對miRNA進行PCR擴增時,由于不能進行多重PCR反應,同時引物二聚體、單鏈二級結構以及錯誤擴增產物的出現會增加熒光值,進而影響檢測結果的準確性,限制了其在miRNA定量檢測中的應用;而特異性熒光定量PCR中TaqMan熒光探針法則沒有上述缺點,在實際操作中被廣泛采納[58-59]。目前,從不同物種中miRNA的檢測效果來看,由于熒光探針法在miRNA的PCR擴增中不僅表現出特異性強,靈敏度高,樣品消耗少,定量線性范圍寬等特點[60-62],能準確區分某一miRNA家族中高度同源的不同miRNA序列,甚至還能檢測出只有一個堿基差異的不同miRNA表達水平,因而在miRNA的定量檢測中得以廣泛應用。然而,盡管探針法在miRNA的定量檢測中表現出較大優勢,但在實際應用過程中每一條miRNA序列都需要設計相應的探針,費用相對較高,限制了該技術的大規模應用。
2.5 高通量測序技術
植物中蘊藏著巨大數量的小分子RNA世界,而傳統的Sanger測序法在測序中程序復雜,效率較低,且測序深度有限,在很大程度上制約著植物中miRNA的發現速度。為了克服傳統Sanger測序法的缺點,更好的挖掘物種中未知的miRNA,自2005年科研人員發明了以大規模平行測序(Massively Parallel Signature Sequencing,MPSS)為代表的第二代高通量測序技術,來挖掘植物中蘊藏的miRNA種類[63-65]。盡管第二代高通量測序技術獲得的序列較短,但它們在測序過程中可以對上萬個樣本同時進行測序,極大地提高了測序效率,具有速度快、成本低、覆蓋度深、產出巨大等優點[66-67]。因此,非常適合小分子RNA測序,尤其是對那些拷貝數低的小分子RNA。
尤其值得一提的是,二代測序技術中Solexa測序技術較其它二代測序技術表現出了前所未有的優勢。它能一次檢測上億個核苷酸片段,且測定成本僅為常規毛細管電泳測序技術成本的1%,是二代測序技術中高質量、高通量、低成本的測序法,具有很高的應用前景。該測序技術的基本流程為:先提取高質量的樣品總RNA,然后用聚丙烯酰胺凝膠電泳分離大小為18~30 nt之的小RNA片段,接著用T4 RNA連接酶將該范圍內的小分子RNA加上5′端接頭和3′端接頭,并對兩端加上接頭的小RNA分子進行反轉錄和PCR擴增。最后將PCR擴增后得到的cDNA放置在flow cell上,進行上機測序,從而直接獲得所有miRNA的序列信息。
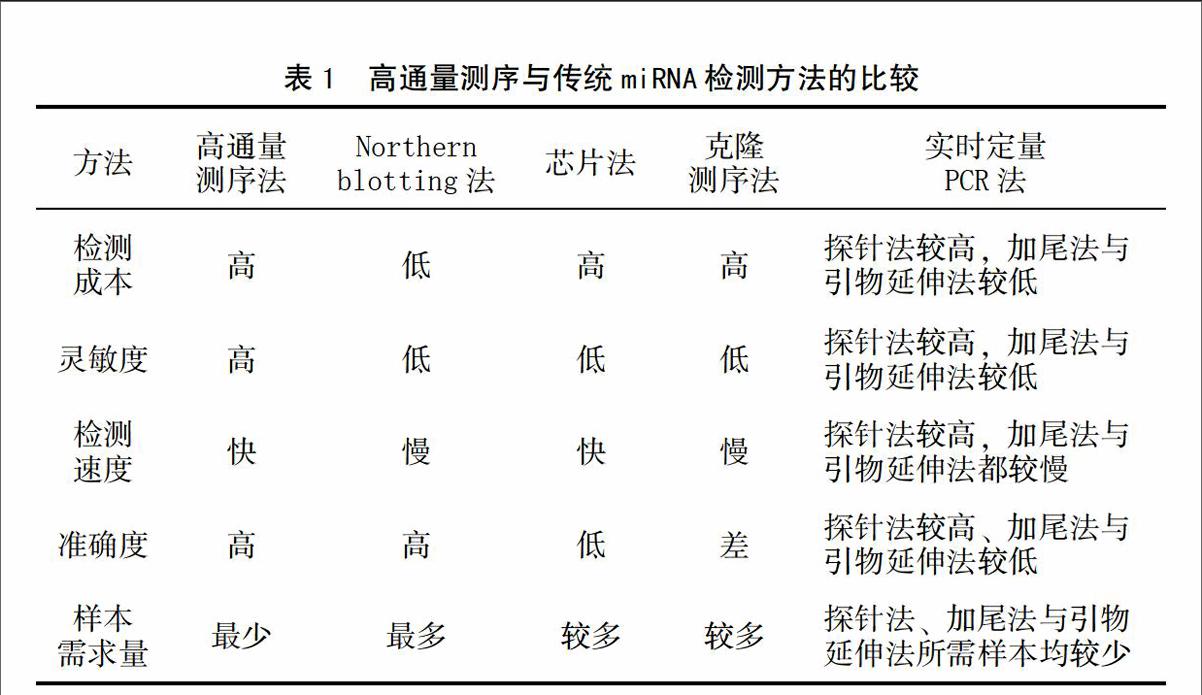
由于植物中miRNA的表達具有時空特異性,并隨著諸多生物學過程的變化而發生變化,而且在植物的不同發育時期、不同生長部位其作用機理表現出較大差異,這就要求miRNA的檢測過程必須具備規模化、高通量、假陽性率低等特征,而新一代高通量測序技術能很好的解決以上問題,較以往的miRNA檢測方法表現出較大優勢(表1)。目前,小RNA數據庫中提交的絕大多數miRNA序列都是通過新一代高通量測序技術發現和鑒定而來。同時,高通量測序技術在植物學研究領域的廣泛應用,不僅提升了植物中miRNA的發掘力度,促進了植物中miRNA的功能研究,提高了人們對不同物種中小分子RNA進化的認識,也為全面、深入研究植物小分子RNA的生物學功能提供最基本的素材。
3 miRNA靶基因鑒定方法
為了更好地了解miRNA的功能及其下游靶基因的相互作用關系,對相關miRNA進行靶基因鑒定就顯得十分必要。目前,主要采用以下3種方法來鑒定相關miRNA的靶基因。
3.1 利用生物信息學預測miRNA的靶基因
植物體內miRNA通常與靶基因進行完全或接近完全的配對來引起mRNA的剪切,從而達到調控基因表達的目的。與動物miRNA相比,植物miRNA與靶基因的互補程度較高,這就為植物miRNA的靶基因預測帶來很大便利。基于miRNA與靶基因的互補配對關系,前人發明了利用生物信息學方法來預測相關miRNA的靶基因。根據前人的研究和總結,miRNA靶基因的生物信息學預測過程中必須遵循一定的流程和規則[68-70],具體規則如下:(1)miRNA與靶基因不得超過4個堿基錯配(G-U配對認為0.5個錯配);(2)miRNA與靶基因的匹配過程中不能有超過2處的相鄰位點堿基錯配;(3)miRNA與靶基因復合體中,從miRNA的5′端起第2~12個位點不得有相鄰位點發生錯配;(4) miRNA與靶基因的匹配過程中不得發生第10或11位點堿基錯配。大多數miRNA都在第10或第11位點對mRNA進行切割,因此這兩個位點不允許有堿基錯配;(5)miRNA與靶基因匹配中,從miRNA的5′端起,第1~12個位點的種子序列之間不得有超過2.5個堿基錯配;(6)miRNA與靶基因匹配后的最小結合自由能(MFE),不能小于該miRNA與靶基因完全互補結合時最小接合自由能的75%。
早期miRNA的功能研究中,許多miRNA的靶基因都是通過生物信息學預測鑒定而來,當時為相關miRNA的功能研究帶來了一定便利。由于傳統的利用生物信息學預測miRNA靶基因的方法主要依靠計算機模擬進行驗證,該方法存在一定的局限性,如相關miRNA的靶基因鑒定結果會出現數據海量,假陽性高,準確性差等弊端。因此,該方法只適合對物種間保守的miRNA進行靶基因鑒定,而對于那些未知的新miRNA進行靶基因鑒定時效果很差,這些不利因素限制了miRNA的后續研究進程。
3.2 利用5′ RLM-RACE技術鑒定miRNA的靶基因
為了從實驗水平直接驗證miRNA的靶基因,人們隨后又發明了基于RNA連接反應的5′ Race技術來鑒定相關miRNA的靶基因[71]。該技術的原理是將提取的RNA連接上5′ Race接頭,在反轉錄酶和GeneRacer Oligo dT 引物的作用下合成cDNA單鏈,然后cDNA單鏈在GeneRacer 5′ Primer(5′-CGACTGGAGCACGAGGACACTGA-3′)和GeneRacer 3′ Primer(5′-GCTGTCAACGATACGCTACGTAACG-3′)的作用下產生非特異性基因的5′ Race擴增產物文庫。接著,使用GeneRacer 5′ Nested Primer和Gene-specific primers進行相關特異性基因的5′ Race產物擴增。最后,對特異性基因的5′ Race擴增產物進行膠回收、克隆、測序分析。
該方法雖能準確鑒定某一特定組織中相關miRNA的靶基因,但在實際應用過程中,仍存在一些不利因素[72-73]。如該技術在操作過程中需要消耗大量的樣品RNA,對于那些采集比較困難的樣品中miRNA的靶基因鑒定不太現實。其次,5′Race試劑盒的價格偏高,致使相關miRNA靶基因的鑒定成本過高。此外,在實際操作過程中,每次僅能從樣品中鑒定有限數目的靶基因,而不能對該組織中的全部miRNA進行靶基因鑒定,使得組織樣品中很多重要的靶基因切割位點信息遺失。因此,該技術只適合小范圍對樣品中的miRNA進行靶基因鑒定,而不能對樣品中絕大多數miRNA的靶基因進行分析,這些不利因素限制了該技術在miRNA功能研究中的大規模應用。
3.3 利用降解組測序技術鑒定miRNA的靶基因
考慮到生物信息學預測miRNA靶基因時存在假陽性偏高、準確性差等弊端,而5′ Race技術又存在成本偏高,不能對組織中全部miRNA的靶基因進行高通量分析的缺點,這些不利因素困擾著miRNA的研究進程。近年來隨著新一代高通量測序(High-throughput Sequencing) 技術的出現和發展,出現了一種新的稱為降解組測序的方法(Degradome Sequencing) ,該技術結合了高通量測序技術與生物信息學分析的優勢,能夠很好的解決miRNA靶基因鑒定過程中存在的弊端和缺陷[74-76],使之在miRNA的研究中得以大規模應用。
降解組測序的原理是基于植物體內絕大多數miRNA是通過剪切作用來實現對靶基因的調控,且miRNA對靶基因的剪切位點通常發生在與mRNA互補區域的第10或第11位堿基位點上。如果mRNA受到相關miRNA的剪切作用,mRNA被剪切后就會產生兩個斷片——即含有帽子結構的5′端剪切片段和帶有poly A尾巴的3′端剪切片段。由于mRNA被剪切后產生的3′端剪切片段含有5′單磷酸自由基團和3′poly A尾巴,該斷片可被RNA連接酶連接,其連接產物可用于下一步的高通量測序;而含有帽子結構的5′端剪切片段因缺少5′單磷酸基團,無法被RNA連接酶連接,因而不能進入下一步的測序實驗。緊接著使用HiSeq2000測序儀對連接產物進行測序,得到50 nt長度的原始數據。原始數據再經去接頭、去污染、去低質量等步驟后獲得“干凈”的序列——即稱為降解序列。降解序列經過一系列的注釋之后,就可得到mRNA的降解片段。最后,獲得的mRNA降解片段通過與miRNA的比對分析后推測出其互補序列,并借助于生物信息學工具target finder做一個反向預測來尋找mRNA-miRNA的配對關系。綜合這兩方面的結果,可以直觀地發現在mRNA序列的某個位點會出現一個波峰,而該峰值對應的mRNA位點正是候選miRNA的剪切位點。
利用降解組測序鑒定miRNA的靶基因具有以下特點:(1)高通量性:一次測序可以檢測到1 000萬條以上的序列信息;(2)高準確性:從幾個到幾十萬個拷貝的精確計數;(3)高分辨率:可以精確檢測到單堿基的差異;(4)重復性好:深度測序保證了檢測的隨機性,不需要技術重復等特點。目前,該方法已被成功應用于水稻、棉花、玉米等重要農作物miRNA靶基因的篩選和鑒定[77-80]。同時,該方法的問世也為miRNA靶基因的鑒定和功能研究帶來了革命性的變革,有利地推動了動植物miRNA的研究進程。
降解組測序技術的出現突破了傳統miRNA靶基因鑒定技術的局限性,為科研人員深入開展miRNA的功能研究提供了更加靈活和有效的分析方法,該技術檢測到的靶基因數量和準確率相對于生物信息學預測有較大提高,而且能大大縮小后續實驗驗證的范圍,其實際效果是傳統的研究方法所無法比擬的(表2)。在實際操作中,借助于生物學實驗手段對樣本中miRNA的靶基因進行高通量測序,可以快速確認上游miRNA的剪切位點及其相互作用的靶基因種類,進而得知樣本中miRNA與下游靶基因的調控通徑。同時,結合不同手段的生物信息學分析,如基因本體論分析(Gene Ontology,GO)、代謝通路(Pathway)分析與表型性狀分析,可以從不同角度對植物miRNA的調控機制進行整體分析。目前,降解組測序技術已被廣泛應用于植物生長發育調控以及逆境脅迫相關的生物學響應機制研究中,成為研究miRNA及其靶基因相互作用關系,進一步探究植物生長發育調控和脅迫相關的調節通路、以及從基因表達的轉錄水平和轉錄后水平構建調控網絡的一種全新而有效的方法。
4 展望
miRNAs的發現是生命科學領域的一個重大突破,它拓展和豐富了人們對生物體內基因表達和小分子RNA調控功能的認識,促使生物學家重新反思對細胞進化的理解和認知。自1993年動物中發現第一個miRNA lin4以來,miRNA領域的相關研究得以飛速發展,并逐步成為生物學研究領域追蹤的熱點之一。現已證實生物體細胞中存在著數量龐大的miRNAs,它們是基因表達調控網絡中豐富而重要的組成部分。在動物研究領域,諸多研究表明,miRNA與人類一些重大疾病的產生密切相關,理解miRNA介導的基因沉默機制,可對諸如癌癥和心腦血管疾病產生的根本原因提供新的見解與治療方案;在植物研究領域,miRNA與一些重要農作物產量形成的分子機制密切相關,通過對一些物種中重要農藝性狀相關miRNA及其靶基因進行鑒定和功能分析,對于詮釋一些重要器官中特定miRNA的生物學功能與代謝機制,破解重要農作物產量形成中的分子調控機制具有重要意義。隨著miRNA及其靶基因鑒定技術的進一步完善和深入,以及大規模高通量測序技術與平臺的廣泛應用,必將促使人們開始對各個物種中特有的miRNA、不同發育狀態下的miRNA、以及各種脅迫條件的miRNA進行挖掘和鑒定,以此來全面探究該物種中miRNA在不同條件及不同發育時期的功能。同時,隨著miRNA及靶基因研究手段的進一步完善,借助于轉基因技術過量表達特定的miRNA或獲得靶基因缺失突變體對miRNA的功能進行驗證技術的進一步完善,這些研究必將推動整個生物學研究領域的飛速發展。
縱觀近年來miRNA及其靶基因檢測與鑒定技術的進展,雖然鑒定并提交至小RAN數據庫中的miRNA數量呈指數增加,但許多miRNA的功能仍然未知,一些提交至小RNA數據庫中miRNA的真實性仍需進一步確認。在miRNA的鑒定與功能研究領域,仍存在許多未解的困惑和未知的領域。總的來說,問題主要包括以下6個方面:其一、關于物種中miRNA種類上限的問題。迄今人們不清楚任何一種物種中miRNA的種類究竟有多少,某一特定物種中發現的miRNA種類距離該物種中全部miRNA種類的上限還有多少? 其二、關于miRNA的靶基因問題。研究發現很多已經鑒定的miRNA根本找不到其作用的靶基因,或者它的靶基因功能未知。其三、關于miRNA與靶基因的接合效率和作用方式問題。在miRNA與靶基因的匹配過程中,究竟什么因素影響了miRNA與UTR位點的接近性和效率? 在什么條件下,miRNA對靶基因進行裂解或者抑制翻譯? 其四、關于miRNA與靶基因的接合位點問題。研究發現很多情況下,多個不同的miRNAs會作用于同一靶基因,這些miRNA結合在同一靶基因位點上,它們如何起作用? 其五、關于miRNA靶基因功能冗余和疊加的問題。研究發現,許多miRNA具有相似的功能,它們的靶基因功能也相似。在進化中這些miRNA靶基因功能冗余和疊加的本質和意義是什么? 其六,關于miRNA與其它幾種內源性小分子RNA作用機制的區別與相互聯系問題。如生物體內miRNAs與siRNAs都可以導致基因表達沉默的產生,但究竟在多大程度上,miRNAs生成通路與siRNAs生產通路使用相似的同源進化基因,何種因素介導了miRNA復合體由細胞核向細胞質轉運,這些問題都有待進一步研究和探討。
參考文獻
[1] International Human Genome Sequencing Consortium. Finishing the euchromatic sequence of the human genome[J]. Nature, 2004, 31(7011): 931-945.
[2] Reinhart B J, Bartel D P. Small RNAs correspond to centromere heterochromatic repeats[J]. Science, 2002, 297(5 588): 1 831-1 831.
[3] Malone C D, Hannon G J. Small RNAs as Guardians of the Genome[J]. Cell, 2009, 136: 656-668.
[4] 屈良鵠. RNA 組學: 后基因組時代的科學前沿[J].中國科學C 輯,2009,39:1-2.
[5] Lee R C, Feinbaum R L, Ambros V, et al. Elegtms heterochronic gene lin-4 encodes small RNAs with antisense complementarity to Lin-l4[J]. Cell, 1993, 75(5): 843-854.
[6] Reinhart B J, Bartel D P. Small RNAs correspond to centromere heterochromatic repeats[J]. Science, 2002, 297(5 588): 1 831-1 831.
[7] Lagos-Quintana M, Rauhut R, Yalcin A, et al. Identification of tissue-specific microRNAs from mouse[J]. Curr Biol, 2002, 12(9): 735-739.
[8] Pasquinelli A E, Reinhart B J, Slack F, et al. Conservation of the sequence and temporal expression of let-7 heterochronic regulatory RNA[J]. Nature, 2000, 408(6 808): 86-89.
[9] Lee R C, Ambros V. An extensive class of small RNAs in Caenorhabditis elegans[J]. Science, 2001, 294(5 543): 862-864.
[10] Griffiths-Jones S, Grocock RJ, Van Dongen S, et al. miRBase: microRNA sequences, targets and gene nomenclature[J]. Nucleic acids research, 2006, 34(1): 140-144.
[11] Griffiths-Jones S, Saini H K, van Dongen S, et al. miRBase: tools for microRNA genomics[J].Nucleic acids research, 2008, 36(1): 154-158.
[12] Wierzbicki AT, Haag JR, Pikaard CS. Noncoding transcription by RNA polymerase Pol IVb/Pol V mediates transcriptional silencing of overlapping and adjacent genes[J]. Cell, 2008, 135: 635–648.
[13] Rehwinkel J, Natalin P, Stark A, et al. Genome-wide analysis of mRNAs regulated by Drosha and Argonaute proteins in Drosophila melanogaster[J]. Mol Cell Biol, 2006, 26: 2 965-2 975.
[14] Bernstein E, Caudy A A, Hammond S M, Hannon G J. Role of a bidentate ribonuclease in the initiation step of RNA interference[J]. Nature, 2001, 409(6 818): 363-366.
[15] Jones-Rhoades M W, Bartel D P, Bartel B. MicroRNAs and their regulatory roles in plants[J]. Annu Rev Plant Biol, 2006, 57: 19-53.
[16] Hammond S C, Bernstein E, Beach D, et al. An RNA-directed nuclease mediates Post transcriptional gene silencing in Drosophila cells[J]. Nature, 2000, 404(6 775): 293-296
[17] Hutvagner G, Zamore P D. RNAi: nature abhors a double strand[J]. Curr Opin Genet Dev, 2002, 12(2): 225-232.
[18] Sunkar R, Zhu J K. Novel and stress-regulated micro-RNAs and other small RNAs from Arabidopsis[J]. Plant Cell, 2004, 16(8): 2 001-2 019.
[19] Sunkar R, Girke T, Jain PK, et al. Cloning and characterization of microRNAs from rice[J]. Plant Cell, 2005, 17(5): 1 397-1 411.
[20] Mica E, Gianfranceschi L, Pe M E. Characterization of five microRNA families in maize[J]. J Exp Bot, 2006, 57(11): 2 601-2 612.
[21] Yao Y Y, Guo G G, Ni Z Y, et al. Cloning and characterization of microRNAs from wheat (Triticum aestivum L.) [J]. Genome Biol, 2007, 8(6): 1-13.
[22] Kwak P B, Wang Q Q, Chen X S, et al. Enrichment of a set of microRNAs during the cotton fiber development[J] .BMC Genomics, 2009, 10(1): 1-11.
[23] Zhang B, Pan X, Stellwag E J. Identification of soybean microRNAs and their targets[J]. Planta, 2008, 229(1): 161-182.
[24] Arazi T, Talmor NM, Stav R, et al. Cloning and characterization of micro-RNAs from moss[J].Plant J, 2005, 43(6): 837-848.
[25] Tang G, Reinhart B J, Bartel D P, et al. A biochemical frame work for RNA silencing in plants[J]. Genes & development, 2003, 17(1): 49-63.
[26] Allen E, Xie Z, Gustafson AM, et al. microRNA-directed phasing during trans-acting siRNA biogenesis in plants[J]. Cell, 2005, 121(2): 207-221.
[27] Jung J H, Seo P J, Park C M. MicroRNA biogenesis and function in higher plants[J]. Plant Bio technol Rep, 2009, 3(2): 111-126.
[28] Sunkar R, Zhu J K. Novel and stress-regulated micro-RNAs and other small RNAs from Arabidopsis[J]. Plant Cell, 2004, 16(8): 2 001-2 019.
[29] Axtell M J, Bartel D P. Antiquity of microRNAs and their targets in land plants[J]. Plant Cell, 2005, 17(6): 1 658-1 673.
[30] Bartel D P. MicroRNAs: genomics, biogenesis, mechanism, and function[J].Cell, 2004, 116(2): 281-297.
[31] Wienholds E, Kloosterman W P, Miska E, et al. MicroRNA expression in zebrafish embryonic development[J]. Science, 2005, 309(5 732): 310-311.
[32] Ambros V. The functions of animal microRNAs[J]. Nature, 2004, 431(7 006): 350-355.
[33] Lau N C, Lim L P, Weinstein E G, et al. An abundant class of tiny RNAs with probable regulatory roles in Caenorhabditis elegans[J]. Science, 2001, 294(5 543): 858-862.
[34] Ambros V, Bartel B, Bartel DP, et al. A uniform system for microRNA annotation[J].RNA, 2003, 9(3): 277-279.
[35] Papp I, Mette MF, Aufsatz W, et al. Evidence for nuclear processing of plant micro RNA and short interfering RNA precursors[J]. Plant physi, 2003, 132(3): 1 382-1 390.
[36] Bartel B, Bartel DP. MicroRNAs: at the root of plant development[J]. Plant Physiology, 2003, 132(2): 709-717.
[37] Lund E, Güttinger S, Calado A, et al. Nuclear export of microRNA precursors[J].Science, 2004, 303(5 654): 95-98.
[38] Liave C, Xie Z, Kasschau KD, et al. Cleavage of Scarecrow-like mRNA targets directed by a class of Arabidopsis miRNA[J].Science, 2002, 297: 2 053-2 056.
[39] Floyd S K, Bowman J L. Gene regulation: ancient microRNA target sequences in plants[J]. Nature, 2004, 428(6 982): 485-486.
[40] Wu S, Huang S, Ding J, et al. Multiple microRNAs modulate p21Cip1/Waf1 expression by directly targeting its 3′ untranslated region[J]. Oncogene, 2010, 29(15): 2 302-2 308.
[41] Cho W C S. OncomiRs: the discovery and progress of microRNAs in cancers[J]. Mol Cancer, 2007, 6(1): 60.
[42] Schetter A J, Leung S Y, Sohn J J, et al. MicroRNA expression profiles associated with prognosis and therapeutic outcome in colon adenocarcinoma[J]. Jama, 2008, 299(4): 425-436.
[43] Zimmerman AL, Wu S. MicroRNAs, cancer and cancer stem cells[J]. Cancer letters, 2011, 300(1): 10-19.
[44] Kong Y W, Ferland-McCollough D, Jackson TJ, et al. microRNAs in cancer management[J].The lancet oncology, 2012, 13(6): 249-258.
[45] Di Leva G, Garofalo M, Croce CM. MicroRNAs in cancer[J]. Annual review of pathology, 2014, 60: 167-179.
[46] Lu C, Fedoroff N. A mutation in the Arabidopsis HYL1 gene encoding a dsRNA binding protein affects responses to abscisic acid, auxin, and cytokinin[J]. Plant Cell, 2000, 12(12): 2 351-2 365.
[47] Schauer S E, Jacobsen S E, Meinke D W, et al. DICER-LIKE1: blind men and elephants in Arabidopsis development[J].Trends in plant science, 2002, 7(11): 487-491.
[48] Pall G S, Codony-Servat C, Byrne J, et al. Carbodiimide-mediated cross-linking of RNA to nylon membranes improves the detection of siRNA, miRNA and piRNA by northern blot[J]. Nucleic Acids Res, 2007, 35(8): 1-9.
[49] Liu C G, Calin G A, Meloon B, et al. An oligonucleotide microchip for genome-wide microRNA profiling in human and mouse tissues[J]. Proc Natl Acad Sci USA, 2004, 101(26): 9 740-9 744.
[50] Chen C, Ridzon D A, Broomer AJ, et al. Real-time quantification of microRNAs by stem–loop RT-PCR[J]. Nucleic acids research, 2005, 33(20): 179-187.
[51] Shi R, Chiang V L. Facile means for quantifying microRNA expression by real-time PCR[J]. Biotechniques, 2005, 39(4): 519-525
[52] Varallyay E, Burgyan J, Havelda Z. MicroRNA detection by northern blotting using locked nucleic acid probes[J]. Nat Protoc, 2008, 3(2): 190-196.
[53] Kubota K, Ohashi A, Imachi H, et al. Improved in situ hybridization efficiency with locked-nucleic-acid-incorporated DNA probes[J]. Appl Environ Microbiol, 2006, 72(8): 5 311-5 317
[54] Castoldi M, Schmidt S, Benes V, et al. miChip: an array-based method for microRNA expression profiling using locked nucleic acid capture probes[J]. Nat Protoc, 2008, 3(2): 321-329.
[55] Lagos-Quintana M, Rauhut R, Meyer J, et al. New microRNAs from mouse and human[J]. Rna, 2003, 9(2): 175-179.
[56] Allawi H T, Dahlberg J E, Olson S, et al. Quantitation of microRNAs using a modified Invader assay[J]. Rna, 2004, 10(7): 1 153-1 161.
[57] Raymond C K, Roberts B S, Garrett-Engele P, et al. Simple, quantitative primer-extension PCR assay for direct monitoring of microRNAs and short-interfering RNAs[J].RNA, 2005, 11(11): 1 737-1 744.
[58] Chen C, Ridzon D A, Broomer A J, et al. Real-time quantification of microRNAs by stem-loop RT-PCR[J]. Nucleic acids research, 2005, 33(20): 179-179.
[59] Varkonyi-Gasic E, Wu R, Wood M, et al. Protocol: a highly sensitive RT-PCR method for detection and quantification of microRNAs[J]. Plant methods, 2007, 3(1): 1-12.
[60] Kramer M F. Stem-Loop RT-qPCR for miRNAs[J]. Current protocols in molecular biology, 2011, 15(10): 1-15.
[61] Hurley J, Roberts D, Bond A, et al. Next-generation microRNA expression profiling technology[J]. Humana Press, 2012: 33-52.
[62] Salone V, Rederstorff M. Stem-Loop RT-PCR Based Quantification of Small Non-Coding RNAs[J]. Small Non-Coding RNAs: Methods and Protocols, 2015, 8(10): 103-108.
[63] Brennecke J, Aravin A, Stark A, et al. Discrete Small RNA-generating loci as master regulators of transposon activity in Drosophila[J]. Cell, 2007, 128(6): 1 089-1 103.
[64] Margulies M, Egholm M, Altman WE, et al. Genome sequencing in microfabricated high-density picolitre reactors[J]. Nature, 2005, 437(7 057): 376-380.
[65] Cokus S J, Feng S, Zhang X, et al. Shotgun bisulphite sequencing of the Arabidopsis genome reveals DNA methylation patterning[J]. Nature, 2008, 452(7 184): 215-219.
[66] Sánchez-León N, Arteaga-Vázquez M, Alvarez-Mejía C, et al. Transcriptional analysis of the Arabidopsis ovule by massively parallel signature sequencing[J]. J Exp Bot, 2012, 63(10): 3 829-3 842.
[67] Nuttle X, Itsara A, Shendure J, et al. Resolving genomic disorder-associated breakpoints within segmental DNA duplications using massively parallel sequencing[J].Nature protocols, 2014, 9(6): 1 496-1 513.
[68] Qiu CX, Xie FL, Zhu YY, et al. Computational identification of microRNAs and their targets in Gossypium hirsutum expressed sequence tags[J]. Gene, 2007, 395: 49-61.
[69] Rhoades MW, Reinhart BJ, Lim LP, et al. Prediction of plant microRNA targets[J]. Cell, 2002, 110: 513-520.
[70] Meyers BC, Axtell MJ, Bartel B, et al. Criteria for annotation of plant microRNAs[J]. Plant Cell, 2008, 20(12): 3 186-3 190.
[71] Liave C, Xie Z, Kasschau KD, et al. Cleavage of Scarecrow-like mRNA targets directed by a class of Arabidopsis miRNA.Science, 2002, 297: 2 053-2 056.
[72] Wang C, Han J, Korir NK, et al. Characterization of target mRNAs for grapevine microRNAs with an integrated strategy of modified RLM-RACE, newly developed PPM-RACE and qPCRs[J]. Journal of plant physiology, 2013, 170(10): 943-957.
[73] Shangguan L, Song C, Han J, et al. Characterization of regulatory mechanism of Poncirus trifoliata microRNAs on their target genes with an integrated strategy of newly developed PPM-RACE and RLM-RACE[J]. Gene, 2014, 535(1): 42-52.
(下轉第36頁)

