Illumina MiSeq平臺高覆蓋率測定干酪中的細(xì)菌微生物多樣性
焦晶凱,莫蓓紅
(乳業(yè)生物技術(shù)國家重點(diǎn)實(shí)驗(yàn)室,光明乳業(yè)股份有限公司乳業(yè)研究院,上海 200436)
內(nèi)蒙古呼倫貝爾草原以畜牧業(yè)為主,是傳統(tǒng)奶制品的發(fā)源地之一,發(fā)酵乳制品種類繁多,奶酪是奶制品中最普遍的食品,為蒙古族人所喜愛,奶酪多以手工家庭制作為主,不添加任何商業(yè)菌株,以自然發(fā)酵為主,發(fā)酵菌株來自原料乳以及周圍的環(huán)境,這些菌株賦予了奶制品特殊的風(fēng)味和質(zhì)地。已有研究顯示,傳統(tǒng)手工干酪中菌種豐富,包含乳桿菌(Lactobacillussp.)、明串珠菌(Leuconostocssp.)、腸球菌(Enterococcisp.)、嗜熱鏈球菌(Streptococcus thermophilus)和乳酸乳球菌(Lactococcus lactis)等[1-4]。然而此前研究微生物多樣性通常使用Sanger第一代測序技術(shù),主要以DNA單鏈為模板,加入脫氧核糖核苷三磷酸(deoxy-ribonucleoside triphosphate,dNTP)和熒光雙脫氧核苷三磷酸(ddNTP)進(jìn)行聚合酶鏈?zhǔn)椒磻?yīng)(polymerase chain reaction,PCR)擴(kuò)增,然后進(jìn)行電泳分離和激光誘導(dǎo)熒光顏色區(qū)分,獲得堿基組成序列,但其成本高、測序通量低、信息不精確[5]。
第二代測序Illumina MiSeq方法分析有效的避免了通量低、操作復(fù)雜和準(zhǔn)確率低等缺陷[6-9],相對于Roche 454焦磷酸測序,具有操作簡單、成本較低的優(yōu)勢,并且采用邊合成邊測序原理,結(jié)果可信度高,MiSeq高通量測序平臺集中了Roche 454[10-11]和Illumina HiSeq 2500的優(yōu)點(diǎn),不僅可實(shí)現(xiàn)對多樣品的多個(gè)可變區(qū)同時(shí)測序,而且在測序速度和測序通量上都有進(jìn)一步提升,目前此平臺已在微生物多樣性群落結(jié)構(gòu)研究方面受到了廣大學(xué)者的認(rèn)可[12-13]。全基因組測序?qū)θ媪私庖粋€(gè)樣本的微生物組成、分子進(jìn)化等有著非常重要的意義。SCHMIDT P A等[14]使用Illumina高通量測序測定了土壤中真菌微生物多樣性,并得到了較為理想的結(jié)果。趙爽[15]使用Illumnina高通量技術(shù)成功分析了對噴施氯苯嘧啶醇后的草坪根際土壤中真菌群落的多樣性。
本研究使用Illumina MiSeq第二代測序方法測定來自內(nèi)蒙古呼倫貝爾的4個(gè)地區(qū)的不同干酪樣品,對其中微生物組成進(jìn)行詳盡的分析,同時(shí)分析了不同樣品間物種差異和進(jìn)化關(guān)系等。
1 材料和方法
1.1 材料與試劑
干酪樣品來自于內(nèi)蒙古呼倫貝爾的4個(gè)地區(qū),分別是哈吉(編號C)、陳巴爾虎旗(編號D)、東烏珠爾(編號E)和西烏珠爾(編號F),這些樣品均為牧民手工制作、自然成熟干酪,保存在4℃環(huán)境下,移至-70℃長期保存。
1.2 儀器與設(shè)備
Pico Green:美國Life Technologies公司;DP328糞便基因組DNA提取試劑盒:天根生化科技(北京)有限公司;AP-GX-500 DNA膠回收試劑盒:美國Axygen公司;P7589 PicoGreendsDNA 定量試劑盒:美國Invitrogen公司;MiSeq測序試劑盒v2、Illumina MiSeq測序儀:美國Illumina公司;DYY-6C電泳儀:北京天誠沃德生物技術(shù)有限公司;凝膠成像系統(tǒng):美國伯樂公司;Pico17離心機(jī):賽默飛世爾科技(中國)有限公司;Aligent 2100電泳生物分析儀:美國安捷倫科技有限公司;BioTek酶標(biāo)儀:美國伯騰儀器有限公司。
1.3 試驗(yàn)方法
1.3.1 DNA提取
干酪樣品中的微生物總DNA是用糞便基因組DNA提取試劑盒,根據(jù)說明書提取DNA。
1.3.2 PCR擴(kuò)增
擴(kuò)增16S rDNA的V4區(qū)域,引物520F:5′AYTGGGYDTAAAGNG3′,引物802R:5′TACNVGGGTATCTAATCC3′。PCR條件預(yù)變性是94℃5min,然后執(zhí)行94℃30s、50℃30s、72℃30s共27個(gè)循環(huán),退火溫度72℃,時(shí)間是5min,最后保持在4℃條件下。1%瓊脂糖電泳檢測,切割回收,使用DNA膠回收試劑盒回收。
1.3.3 各樣本定量
采用BioTek酶標(biāo)儀對各個(gè)樣品定量,取1μL樣品加入24μL TE和25μL PicoGreen(稀釋200倍)置于costar 96孔半量酶標(biāo)儀上,在激發(fā)光485/20nm,發(fā)射光528/20nm下測定熒光強(qiáng)度。
1.3.4 DNA序列修飾
通過3′-5′核酸外切酶及聚合酶的共同作用,修復(fù)帶有突出末端的DNA片段。在修復(fù)平整的DNA片段3′端引入單堿基“A”,接頭3′末端含有單堿基“T”,從而保證DNA片段和接頭能夠通過“A”“T”互補(bǔ)配對連接,并防止接頭連接DNA片段的過程中,DNA插入片段彼此相連,在連接酶的作用下,孵育含有標(biāo)簽的接頭與DNA片段,使其相連。利用PCR選擇性的富集兩端連有接頭的DNA片段,同時(shí)擴(kuò)增DNA文庫。PCR應(yīng)盡量使用較少的環(huán)數(shù),避免PCR擴(kuò)增中文庫出現(xiàn)錯誤。
1.3.5 驗(yàn)證和混合文庫
利用PicoGreen和熒光分光光度計(jì)方法定量文庫,使用Agilent 2100對PCR富集片段進(jìn)行質(zhì)量控制,驗(yàn)證DNA文庫的片段大小分布。
1.3.6 上機(jī)測序
將混合好的文庫(10nmol/L)逐步稀釋定量至4~5pmol/L后進(jìn)行上機(jī)測序,使用合成測序法,測定長度2×250bp。
1.3.7 數(shù)據(jù)分析
1.3.7.1 原始數(shù)據(jù)處理與樣品序列數(shù)目統(tǒng)計(jì)
采用雙峰(pair-end)測序,首先對原始數(shù)據(jù)進(jìn)行質(zhì)量控制,舍棄低質(zhì)量序列(50個(gè)連續(xù)堿基平均質(zhì)量>Q30,不允許有N)。用軟件Flash連接通過質(zhì)量控制的序列對應(yīng)的兩端序列進(jìn)行。設(shè)計(jì)無法連接的序列。對連接上的序列進(jìn)行過濾(連續(xù)相同堿基<6;模糊堿基N<1),獲得最終用于分析的序列。
1.3.7.2 OTU列表生成
應(yīng)用Qiime,根據(jù)序列的相似度,將序列歸為多個(gè)操作分類單元(operational taxonomic unit,OTU)。Qiime調(diào)用uclust對序列進(jìn)行聚類,選取每個(gè)類最長的序列為代表序列。采用RDP-classifier,以RDP數(shù)據(jù)庫的序列為訓(xùn)練集,對OTU代表序列進(jìn)行注釋。得到每個(gè)OTU的分類學(xué)信息。
1.3.7.3 稀釋曲線及豐度分布
根據(jù)獲得的OTU數(shù)據(jù),作出每個(gè)樣品的稀釋曲線,以該曲線表明樣品的取樣深度。對各樣品的OTU豐度大小排序,對豐度值取log2的對數(shù)作豐度分布曲線圖,可提現(xiàn)樣品中物種分布的均勻性。
1.3.7.4 群落結(jié)構(gòu)分析
對OTU列表中獲得的分類信息與豐度進(jìn)行整理,在屬層次下對各樣品進(jìn)行物種豐度統(tǒng)計(jì)、聚類分析及PCoA分析,可得到樣品中群落組成結(jié)構(gòu)及它們的相似性。
2 結(jié)果與分析
C、D、E、F4種樣品分別讀取了26 776、25 003、17 342、24 709條有效序列和26 704、24 927、17 287、24 640條優(yōu)質(zhì)序列。
2.1 樣品所含操作分類單元及所含序列的數(shù)目
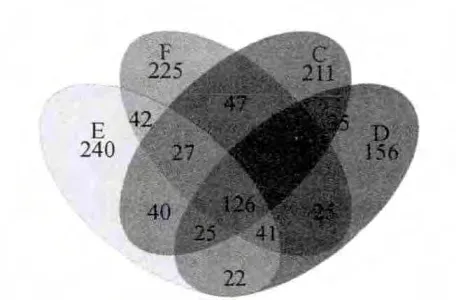
圖1 操作分類單元維恩圖Fig.1 Venn diagram of operational taxonomic unit
如圖1所示,C樣品中含有細(xì)菌種類541個(gè),D樣品中460個(gè),E樣品中含有563個(gè),F(xiàn)樣品中含有593個(gè)。C和F有相同種類230個(gè),C和D有相同種類216個(gè),C和E有相同種類218個(gè),F(xiàn)和D有相同種類222個(gè),F(xiàn)和E有相同種類236個(gè),D和E有相同種類214個(gè)。C、F和D有相同種類156個(gè),C、F和E有相同種類153個(gè),C、D和E有相同種類151個(gè),F(xiàn)、D和E有相同種類167個(gè),在相似度0.97基礎(chǔ)上分類出1 322個(gè)OTU,門1 142,綱1 057,目952,科716,屬432。
2.2 物種豐度及群落結(jié)構(gòu)分析
2.2.1 稀釋曲線
圖2表明每個(gè)樣品的取樣深度,從4種樣品中隨機(jī)抽取的測序條數(shù)如圖2橫軸所示,縱軸則表示基于該測序條數(shù)能構(gòu)建的OUT數(shù)量,當(dāng)曲線趨于平坦時(shí),說明測序數(shù)據(jù)量合理,更多的數(shù)據(jù)量對發(fā)現(xiàn)新OTU的邊際貢獻(xiàn)很小;反之則表明繼續(xù)測序還可能產(chǎn)生較多新的OTU。該分析是基于OTU序列差異水平在0.03即相似度97%的水平上進(jìn)行運(yùn)算的。
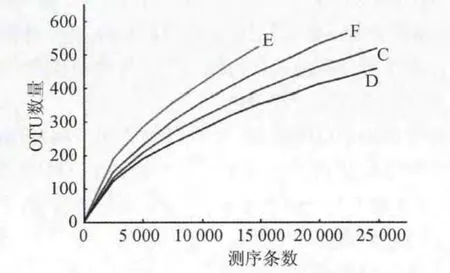
圖2 4種樣品的稀釋曲線Fig.2 Dilution curve of four cheese samples
2.2.2 豐度分布曲線
對豐度值取log2的對數(shù)值得到圖3所示的豐度分布曲線圖。橫軸所示為OTU相對豐度含量等級降序排列,縱軸對應(yīng)的是OTU(物種)所占相對豐度比例,由圖3可以看出4種樣品的曲線斜率較大。
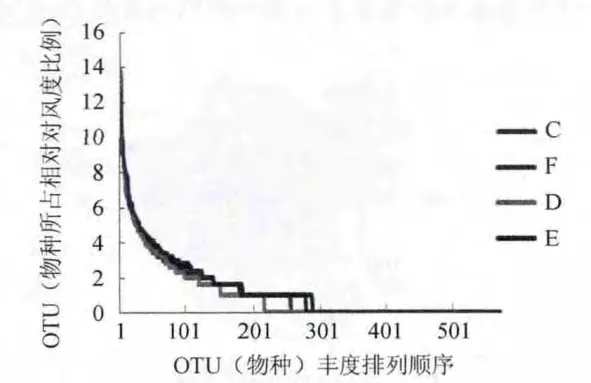
圖3 4種樣品的風(fēng)度分布曲線Fig.3 Rank-abundance curve of four cheese samples
2.2.3 干酪中群落組成分析
在屬的層次下對各樣品物種豐度進(jìn)行統(tǒng)計(jì),如圖4所示,4個(gè)樣品所含物種基本相似,但物種分布差異較大,對所含主要種屬進(jìn)行了分析和歸類。由圖4可知,主要物種包括3類,乳酸桿菌(Lactobacillus)、醋酸菌(Acetobacter)和乳酸球菌(Lactococcus);C樣品與其他3個(gè)樣品的不同在于其含量最多物種為醋酸菌醋酸菌(Acetobacter)而其他3個(gè)樣品為乳酸桿菌(Lactobacillus);F和D樣品較為類似;E樣品相對于其他樣品來說,含有較少的醋酸菌,其他物種含量相對豐富些;此外在4種樣品中還發(fā)現(xiàn)有巨型球菌(Macrococcus)、明串珠菌(Leuconostoc)、鏈球菌(Streptococcus)、腸桿菌(Enterobacteriaceae)和假單胞菌(Pseudomonas)等。

圖4 4種干酪樣品物種分布圖Fig.4 Species distribution in four cheese samples
2.2.4 聚類分析
本實(shí)驗(yàn)在屬的水平上利用Heapmap圖分析了4個(gè)樣品菌群的相似性和多樣性,如圖5所示,所含菌落分為3個(gè)大的分支,其中最為優(yōu)勢的OTU單元為硬壁菌門(Firmicutes);OTU925包含序列數(shù)最多,其34 227條序列均屬于乳酸桿菌(Lactobacillus)。由圖5可知,在OTU861和9254包含序列也較多,分別屬于醋酸桿菌(Acetobacter)和乳酸球菌(Lactococcus),OTU58、OTU717、OTU73、OTU479和OTU 1178等也屬于乳酸桿菌(Lactobacillus),此外序列較多的還包含假單胞菌(Pseudomonas)、腸桿菌(Enterobacteriaceae)、鏈球菌(Streptococcus)和明串珠菌(Leuconostoc)等。

圖5 系統(tǒng)發(fā)育進(jìn)化樹Fig.5 Phylogenetic tree analysis base on 16S rDNA gene of four cheese samples
2.2.5 主坐標(biāo)PCoA分析
如圖6所示,利用各樣品序列間的進(jìn)化信息計(jì)算樣品距離,觀察4種不同干酪樣品多樣性差異。由圖6可知,PCoA分析中這些站點(diǎn)沒有聚在一起,距離相對較遠(yuǎn),表明4個(gè)干酪樣品群落存在一定的差異性,因此,地域?qū)Ω鱾€(gè)群落組成有重要影響。
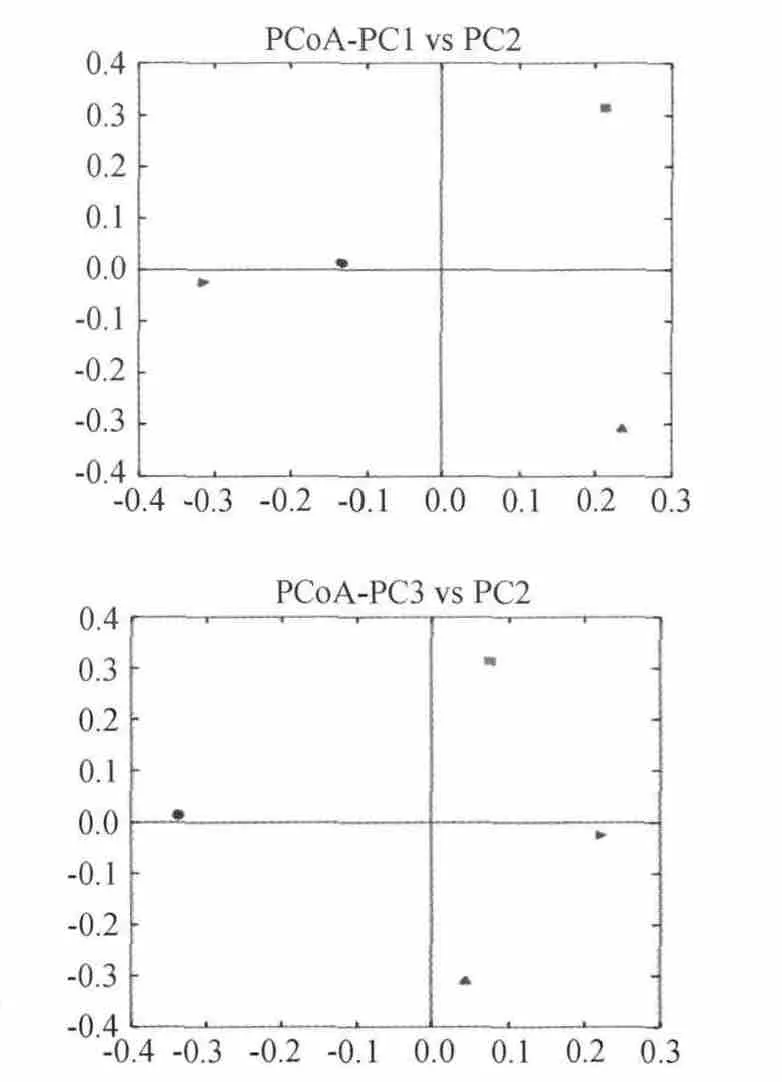
圖6 4種干酪樣品群落主坐標(biāo)分析Fig.6 Principal coordinates analysis of microbiota in four cheese samples
3 結(jié)論
采用Illumina MiSeq測序方法,直接從環(huán)境樣品中擴(kuò)增高變區(qū)進(jìn)行測序,避免了不可培養(yǎng)菌株的不可測性,客觀還原菌群結(jié)構(gòu)及豐度比例,嚴(yán)格控制PCR循環(huán)數(shù),充分發(fā)揮高通量測序的大數(shù)據(jù)量優(yōu)勢。
結(jié)果表明,4個(gè)干酪樣品共分離了細(xì)菌門1 142個(gè),綱1 057個(gè),目952個(gè),科716個(gè),屬432個(gè),主要包括乳酸桿菌(Lactobacillus)、醋酸桿菌(Acetobacter)和乳酸球菌(Lactococcus),本實(shí)驗(yàn)的稀釋曲線分析了4種樣品取樣深度,每個(gè)樣品都呈現(xiàn)相同或相似的變化趨勢,在測序量較少時(shí),每個(gè)樣品OTU數(shù)目呈顯著上升的趨勢,而隨著測序數(shù)量的增加,每個(gè)樣品的OTU數(shù)目增加趨勢逐漸變緩,最后基本達(dá)到飽和。由本實(shí)驗(yàn)4種樣品的豐度曲線可以看出,相對于環(huán)境、土壤等樣品來說曲線較窄,說明干酪中的菌種相對來說較為單一,曲線斜率較大,由此推算4種干酪樣品中存在優(yōu)勢菌株。通過菌落組成分析,F(xiàn)、D、E 3個(gè)樣品的主要優(yōu)勢菌為乳酸桿菌,所占比例均超過了50%,而C樣品中醋酸菌所占比例超過50%,另外一種優(yōu)勢菌是乳酸球菌。PCoA分析可以看出4個(gè)樣品菌群分布大不相同,說明地域?qū)ζ湮⑸锝M成影響較大。
[1]CALLON C,MILLET L,MONTEL M C.Diversity of lactic acid bacteria isolated from AOC Salers cheese[J].J Dairy Res,2004,71(2):231-244.
[2]CHEBE OVá-TURCOVSKá V,?ENI?OVá K,KUCHTA T,et al.Culture-independent detection of microorganisms in traditional Slovakian bryndza cheese[J].Int J Food Microbiol,2011,150(1):73-78.
[3]NIKOLIC M,TERZIC-VIDOJEVIC A,JOVCIC B,et al.Characterization of lactic acid bacteria isolated from Bukuljac,a homemade goat's milk cheese[J].Int J Food Microbiol,2008,122(1-2):162-170.
[4]TERZIC-VIDOJEVIC A,VUKASINOVIC M,VELJOVIC K,et al.Characterization of microflora in homemade semi-hard white Zlatar cheese[J].Int J Food Microbiol,2007,114(1):36-42.
[5]秦 楠,栗東芳,楊瑞馥.高通量測序技術(shù)及其在微生物學(xué)研究中的應(yīng)用[J].微生物學(xué)報(bào),2011,51(4):445-457.
[6]RUTVISUTTINUNT W,CHINNAWIROTPISAN P,SIMASATHIEN S,et al.Simultaneous and complete genome sequencing of influenza A and B with high coverage by Illumina MiSeq Platform[J].J Virol Methods,2013,193(2):394-404.
[7]WILLIAMS S,FOSTER P,LITTLEWOOD D.The complete mitochondrial genome of a turbinid vetigastropod from MiSeq Illumina sequencing of genomic DNA and steps towards a resolved gastropod phylogeny[J].Gene,2014,533(1):38-47.
[8]蔡元鋒,賈仲君.基于新一代高通量測序的環(huán)境微生物轉(zhuǎn)錄組學(xué)研究進(jìn)展[J].生物多樣性,2013(4):402-411.
[9]王興春,楊致榮,王 敏,等.高通量測序技術(shù)及其應(yīng)用[J].中國生物工程雜志,2012,32(1):109-114.
[10]段 曌,肖 煒,王永霞,等.454 測序技術(shù)在微生物生態(tài)學(xué)研究中的應(yīng)用[J].微生物學(xué)雜志,2011,31(5):76-81.
[11]徐曉宇,劉 和.454 測序法在環(huán)境微生物生態(tài)研究中的應(yīng)用[J].生物技術(shù)通報(bào),2010(1):73-77.
[12]董 逸.我國黃、東海典型海域微生物群落結(jié)構(gòu)及其與環(huán)境變化的關(guān)系[D].青島:中國科學(xué)院研究生院(海洋研究所)博士論文,2013.
[13]周林文.MiSeq:新一代個(gè)人化測序儀[J].生物技術(shù)世界,2011(10):14-16.
[14]SCHMIDT P A,BáLINT M,GRESHAKE B,et al.Illumina metabarcoding of a soil fungal community[J].Soil Biol Biochem,2013,65:128-132.
[15]趙 爽.土壤病原真菌的檢測及土壤中真菌群落多樣性的研究[D].南京:南京農(nóng)業(yè)大學(xué)博士論文,2012.

