山羊源奧斯陸莫拉菌的分子鑒定與分析
張素輝,黃勇富,付利芝,沈克飛,徐登峰
奧斯陸莫拉菌(Moraxella osloensis)為革蘭氏陰性球桿菌,是Henriksen于1967年在奧斯陸發現的,該菌是人和動物黏膜上的正常菌群,可從健康成年人的鼻腔和門診病人的耳朵、咽喉、氣管、血液等分離到[1-4]。奧斯陸莫拉菌對頭孢曲松高度敏感,主要在機體抵抗力及免疫力降低時,引起呼吸系統、泌尿生殖等多系統感染,同時可能導致腦膜炎、心內膜炎、肺炎、腹膜炎、陰道炎、骨髓炎、化膿性關節炎及菌血癥等[5]。臨床病例中有近一半患病對象是兒童[6]。目前,國內關于奧斯陸莫拉菌對山羊的危害還未見報道,本研究對重慶市某羊場送檢臨床肺炎病例病山羊的病原微生物進行了分離培養,通過分子檢測方法鑒定,該山羊肺炎病例病原菌為奧斯陸莫拉菌。
1 材料與方法
1.1試驗動物 4只20 d齡的健康羔羊,購自榮昌某羊場。
1.2試驗試劑 小牛血清瓊脂平板培養基,LB培養基均由本實驗提供;PCR反應試劑2×Taq PCR Master Mix、DNA抽提試劑盒購自天根生化科技(北京)有限公司;青霉素等20種藥敏紙片購自自上海衛生技術服務公司生物試劑所;瓊脂糖凝膠回收試劑盒購自廣州東盛生物科技有限公司;16S rDNA的PCR擴增引物為通用引物,預期擴增片段大小為1.5 kb左右,由大連寶生物工程有限公司合成。引物序列為:
F27:5’-AGAGTTTGATCCTGGCTCAG-3’,
R1492:5’-ACGGCTACCTTGTTACGACTT-3’
1.3病原菌的分離純化 實驗室解剖送檢肺炎病例山羊,對肺臟化膿部位進行無菌劃線接種于普通瓊脂平板和小牛血清瓊脂平板上,37 ℃恒溫培養24~48 h,觀察細菌的生長情況,并將分離菌進行純化培養。
1.4致病性試驗 將分離到的病原菌接種到含有小牛血清的肉湯培養基培養中 ,37 ℃,250 r/min搖床培養48 h后,取6 mL菌液4 000 r/min離心5 min,收集菌體,再用生理鹽水稀釋到原有濃度。選取4只健康的20日齡的羔羊,分為兩組,然后選取3只羔羊通過氣管注射上述菌液,2 mL/只,剩余的1只注射生理鹽水作為對照,觀察羔羊的健康情況。
1.5藥敏試驗 參照美國臨床實驗室標準化委員會標準,采用紙片法做藥敏試驗,取200 μL菌液涂布于小牛血清培養基平板上,并貼上藥敏紙片,37 ℃恒溫培養24 h后測定抑菌圈直徑。主要檢測了該菌對頭孢曲松等29種抗生素的敏感性,抑菌圈直徑<10 mm表示其對抗生素具有一定的耐藥性,10~15 mm的為中度敏感,>15 mm的為高度敏感。
1.6病原菌的16SrDNA的 PCR擴增 取1.5 mL純培養菌液離心,按DNA抽提試劑盒說明書提取基因組。在PCR反應體系為:2×TaqPCR Master Mix 25 μL,上、下游引物各1 μL(50 μmol/L),模板DNA 2 μL,加ddH2O至50 μL,在PCR儀中進行擴增。反應條件為:94 ℃預變性4 min,94 ℃變性45 s,55 ℃退火 45 s,72 ℃ 延伸45 s,30個循環,72 ℃延伸10 min,結束反應后4 ℃保存。PCR產物采用l0 g/L瓊脂糖凝膠電泳進行檢測。
1.7PCR產物的純化與測序 將預期目的條帶大小相符的PCR產物經膠回收試劑盒純化后,送往大連寶生物工程有限公司進行DNA測序。
1.8系統進化分析 將分離菌株16S rDNA序列與GenBank中已知的相關核酸序進行分析比對,并利用生物軟件MEGA5通過距離法構建系統進化發育樹。
2 結 果
2.1菌株的鏡檢及培養特性結果 對純化得到的菌株進行革蘭氏染色后發現該菌為革蘭氏陰性菌,呈桿狀,短且寬,近橢圓形,單個、成對或呈短鏈狀,鏡檢結果如圖1所示。分離到的菌株在普通瓊脂平板上生長十分緩慢,24 h后仍觀察不到菌落,說明該菌對營養需求較高;在小牛血清瓊脂培養基上生長48 h后,可觀察到直徑為2.0~2.5 mm的粘性菌落,如圖2所示。
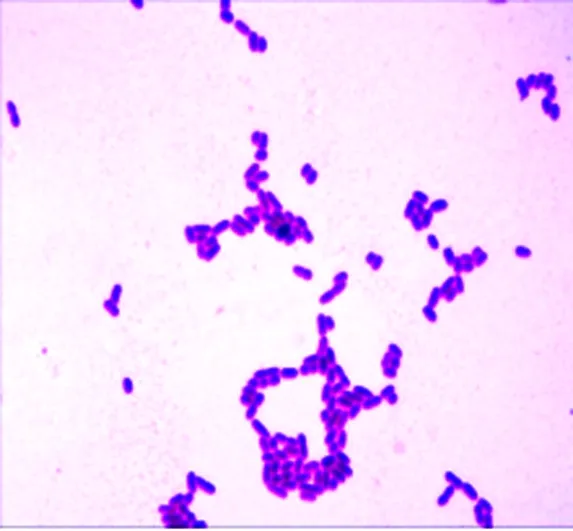
圖1 革蘭氏染色結果
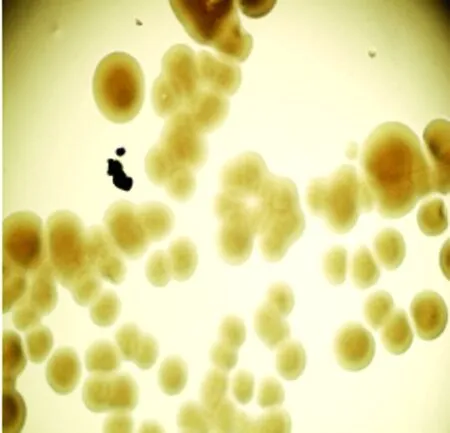
圖2菌落圖片
Fig.2Bacterialcolony
(10× object glass)
2.2致病性試驗 實驗組的3只羔羊被接種病原菌以后,均在7 d左右開始發病,體溫升高到40 ℃~42 ℃,出現眼、鼻黏膜變紅,發干,流鼻液、咳嗽、喘氣等呼吸道癥狀,無死亡現象,對照組的山羊無異常癥狀。對實驗組的羊只進行肺臟病原菌的分離鑒定,通過革蘭氏染色鏡檢發現分離到大量革蘭氏陰性短桿菌,菌體呈單個、成對,亦有短鏈狀。
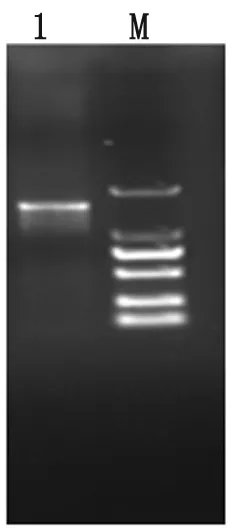
圖3 電泳結果
1: PCR amplification products of 16SrDNA;
M: DNA marker
2.3藥敏試驗 通過對分離的菌株進行藥敏試驗,發現其對頭孢曲松等19種抗生素的敏感性較高,對甲氧芐啶等4種抗生素為中度敏感,對紅霉素等6種抗生素敏感性較弱,表現出一定的抗性。試驗結果見表1。
2.4菌株16S rDNA序列的擴增與鑒定 利用通用引物對該菌進行16SrDNA的擴增,得到大小為1 447 bp的片段,與預期結果一致,電泳結果如圖3所示;擴增產物的核苷酸序列測序結果如圖4所示。
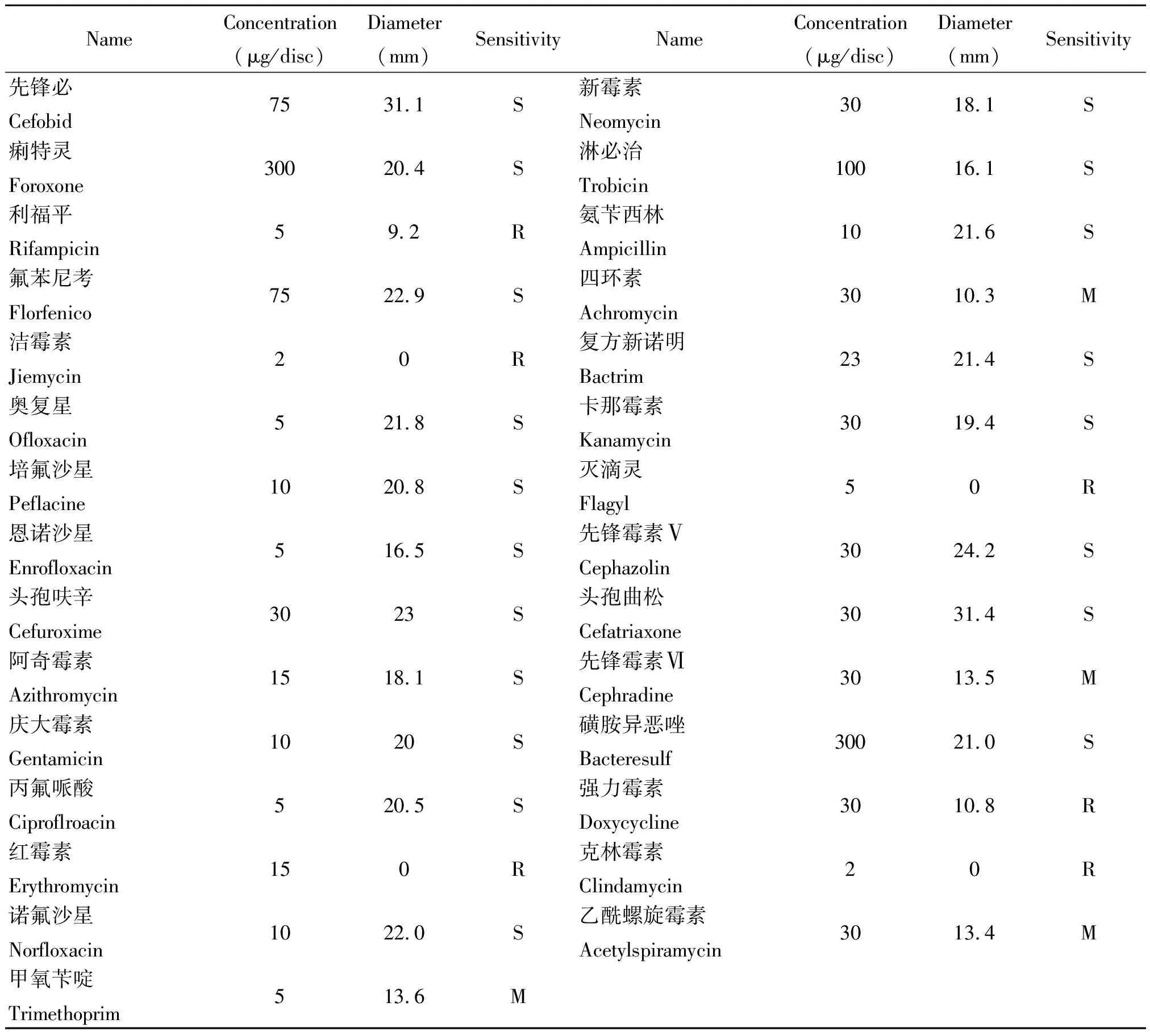
表1 抑菌試驗結果
Note: S--High sensitive; M--Medium sensitive; R--Resistant.

圖4 PCR產物測序結果
2.5系統進化分析結果 將該菌的16SrDNA序列在GenBank進行序列比對,發現其與菌株JN084136同源性達99.9%。將該序列與相關菌屬奈瑟氏菌屬(Neisseria)、莫拉克斯氏菌屬(Moraxella)、俊片菌屬(Lampropedia)、副球菌屬(Paracoccus)的代表菌株16S rDNA進行序列比對分析,各代表菌株相關信息如表2,并利用生物軟件MEGA5構建系統進化樹,系統發育進化結果如圖5所示。
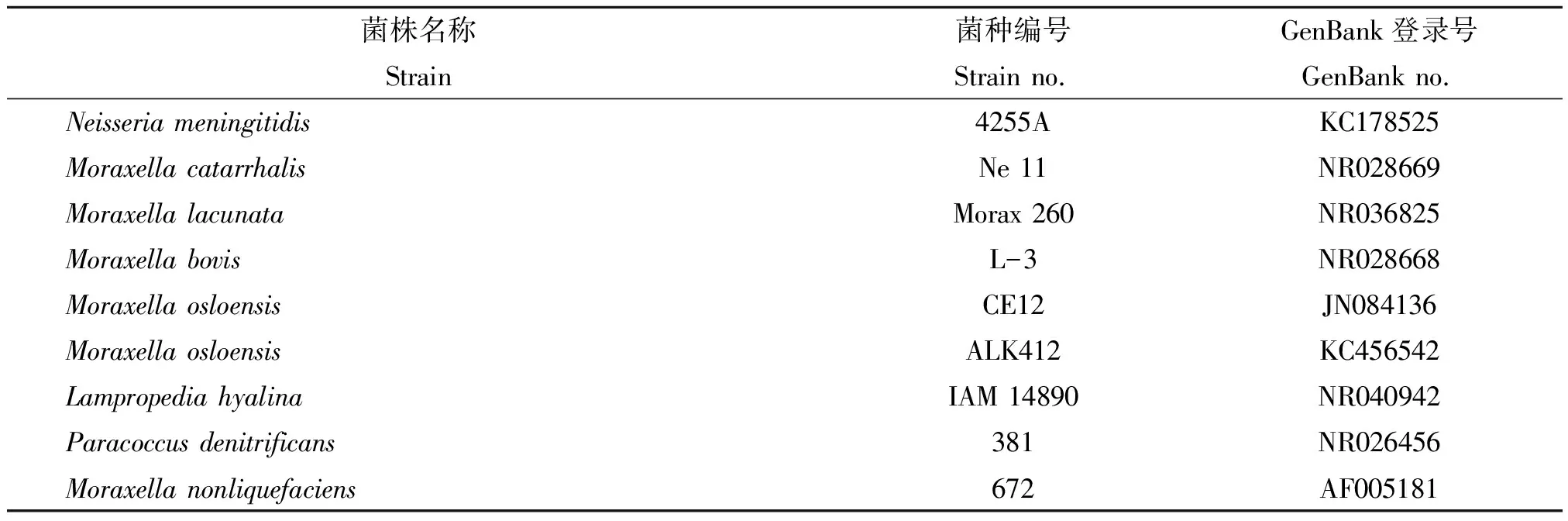
表2 系統發育樹的代表菌株及16rDNA序列信息表
3 討 論
隨著分子生物學的發展,現代細菌分類鑒定從傳統的表型分類進入基因型分類水平,16S rDNA分析技術已成為研究生命系統進化及細菌分類的常用工具,16S rDNA系統發育學分析技術通過比較細菌核糖體DNA片段的同源性實現對細菌的分類學鑒定,理論上具有很好的可靠性[7]。本研究基于16S rDNA序列的同源性的比對表明,分離到的病原菌與各代表菌株的同源性均較高,其中與菌株JN084136同源性高達99.9%;通過構建系統發育進化樹,可以發現實驗分離菌株與Moraxella的代表菌株聚為一簇,Neisseria和Lampropedia位于另一分支,與實驗分離菌株進化距離最近的菌株是Moraxellaosloensis(JN084136),由此可以確定該分離菌為奧斯陸莫拉菌[8-9]。同時為今后奧斯陸莫拉菌性肺炎的PCR快速診斷方法的建立及該病的防治研究提供了可靠的試驗數據。
目前,在國內關于奧斯陸莫拉菌以及它對山羊的生理機能危害的報道相對較少,可能的原因是該菌致病性不強,不會引起動物大規模的死亡,沒有得到人們特別關注;或者是該病原菌可能對針對肺炎病例的藥物較為敏感,相對易于治愈;同時該菌的對生長條件要求較為嚴格,不易分離。而且該病原菌引起的肺炎病例易與溶血性巴氏桿菌、支原體等病原引起的呼吸道病癥相混淆,故目前臨床上暫無奧斯陸莫拉菌致山羊疾病病例的報道。健康動物機體抵抗力較強時,不會因感染該病原菌而患病,當機體體抗力低下時,該菌會引發類似肺炎的癥狀,本次試驗對羊只接種的菌的濃度較大,導致羊只出現了一定癥狀。奧斯陸莫拉菌可以造成人獸共患病,其對人體的危害還有待于進一步研究。
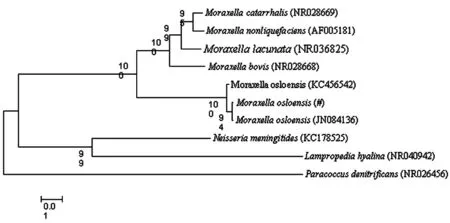
圖5基于16SrDNA序列構建的系統發生樹狀圖(Moraxellaosloensis(#)為分離病原菌)
Fig.5Phylogenetictreebasedon16SrDNAsequences(Moraxellaosloensis(#)isthepathogenicbacterium)
參考文獻:
[1]Berger U, Felsen E. Distribution of species ofMoraxellaandMoraxella-like organisms in the nasopharynx of healthy human adults[J]. Med Microbiol Immunol, 1976, 162: 239-249.
[2]Bovre K. Oxidase positive bacteria in the human nose, incidence and species distribution, as diagnosed by genetic transformation[J]. Acta Pathol Microbiol Scand, 1970, 78: 780-784.
[3]Graham DR, Band JD, Thornsberry C, et al. Infections caused byMoraxella,Moraxellaurethralis,Moraxella-like groups M-5 and M-6, andKingellakingaein the United States, 1953-1980[J]. Rev Infect Dis, 1990, 12(3): 423-431. DOI:10.1093/clinids/12.3.423
[4]Dien Bard J, Lewinski M, Summanen PH, et al. Sepsis with prolonged hypotension due toMoraxellaosloensisin a nonmmunocompromised child[J]. J Med Microbiol, 2011, 60(Pt 1): 138-141. DOI:10.1099/jmm.0.016378-0
[5]Shah SS, Ruth A, Coffin SE. Infection due toMoraxellaosloensis: case report and review of the literature[J]. Clin Infect Dis, 2000, 30(1): 179-181. DOI:10.1086/313595
[6]Han XY, Tarrand JJ.Moraxellaosloensisblood and catheter infections during anticancer chemotherapy: clinical and microbiologic studies of 10 cases[J]. Am J Clin Pathol, 2004, 121(4): 581-587. DOI:10.1309/QBB3AVCMGWA3K1XK
[7]Pettersson B, Kodjo A, Ronaghi M, et al. Phylogeny of the familyMoraxellaceaeby 16S rDNA sequence analysis with special emphasis on differentiation ofMoraxellaspecies[J]. Int J Syst Bacteriol, 1998, 48(Pt 1): 75-89. DOI:10.1099/00207713-48-1-75
[8]Juni E. Simple genetic transformation assay for rapid diagnosis ofMoraxellaosloensis[J]. Appl Microbiol, 1974, 27(1): 16-24.
[9]Coenye T, Goris J, Spilker T, et al. Characterization of unusual bacteria isolated from respiratory secretions of cystic fibrosis patients and description ofInquilinuslimosusgen. nov, sp nov[J]. J Clin Microbiol, 2002, 40(6): 2062-2069. DOI:10.1128/JCM.40.6.2062-2069. 2002

