重組人BMP-2誘導C3H10T1/2間質干細胞定向成骨分化早期基因表達譜分析
左長清 汪宗桂 鐘月春 盧旱云 戴忠 吳鐵 崔燎
1.廣東醫學院藥理學教研室,廣東東莞523808;2.廣東醫學院生物化學教研室,廣東東莞523808
重組人BMP-2誘導C3H10T1/2間質干細胞定向成骨分化早期基因表達譜分析
左長清1汪宗桂2鐘月春1盧旱云1戴忠1吳鐵1崔燎1
1.廣東醫學院藥理學教研室,廣東東莞523808;2.廣東醫學院生物化學教研室,廣東東莞523808
目的研究間質干細胞早期定向成骨分化基因表達譜,為研究基因對早期成骨定向分化調控機制提供實驗基礎。方法分別提取重組人骨形成蛋白2(rhBMP.2)誘導組和對照組C3H10T1/2細胞總RNA,進行擴增標記后,與ArraySTAR小鼠基因芯片雜交,應用生物信息學軟件GeneSpring和GATHER對基因芯片數據進行分析。應用STRING在線軟件對差異表達基因構建蛋白互作網絡并進行網絡分析。結果C3H10T1/2早期成骨分化中,主要富集發育、器官形成等分子功能本體以及細胞因子.細胞因子受體作用信號通路。成骨分化1 d和4 d均上調表達基因42個,下調表達基因45個。網絡分析研究表明:Egfr、Cxcl12等信號分子參與調控rhBMP.2誘導成骨分化。結論篩選的差異表達基因和信號分子對早期成骨分化調控具有重要作用,為進一步全面解析早期成骨定向分化提供實驗基礎。
早期成骨分化;基因表達譜;間質干細胞;生物信息學
成骨細胞主要由骨髓間質干細胞(mesenchymal stem cells,MSCs)分化而來,在其發育分化的整個過程中,不同的基因嚴格按照特定的時間順序開啟和關閉,表現為與分化的階段相一致,支持并且控制著成骨細胞的分化與成熟。因此,骨髓間質干細胞定向成骨分化調控研究在國內外受到高度重視,促進間質干細胞成骨定向分化與成熟,將是治療骨質疏松癥的新的有效方法和手段。但是,目前關于間質干細胞早期成骨定向分化方面的調控機制尚未完全闡明。本實驗室采用重組人骨形成蛋白2(rhBMP.2)誘導小鼠間質干細胞C3H10T1/2定向成骨分化細胞模型,采用小鼠全基因組芯片篩選鑒定差異表達基因譜并進行生物信息學分析,探討rhBMP.2成骨分化調控的差異表達基因集,并進一步通過網絡生物學分析,為探討間質干細胞早期成骨定向分化提供實驗依據。
1 材料與方法
1.1 材料
小鼠間質干細胞C3H10T1/2購自中國科學院上海細胞庫。rhBMP.2(R&D systems),ArraySTAR mouse lncRNA microarray(8×60 K,上海康成生物,芯片包含31423條蛋白編碼mRNA和25376條lncRNA,本文僅分析編碼蛋白mRNA表達變化)。BCIP/NBT堿性磷酸酶染色試劑盒(Sigma)。
1.2 實驗方法
1.2.1 堿性磷酸酶染色鑒定早期成骨定向分化C3H10T1/ 2接種于6孔板中,細胞生長至約90%融合,rhBMP.2誘導組加入含200 ng/mL rhBMP.2培養基,對照組加入不含rhBMP.2的培養基,每2~3天更換一次培養基。當成骨誘導分化7 d后,rhBMP.2誘導組、對照組的單層細胞用PBS清洗2次,然后用體積分數為0.7乙醇室溫固定15 min,BCIP/NBT避光染色30 min,用蒸餾水漂洗細胞3次,倒置顯微鏡下觀察并拍照記錄。
1.2.2 C3H10T1/2早期成骨定向分化基因芯片檢測及其生物信息學分析C3H10T1/2成骨定向分化1 d和4 d后,rhBMP.2誘導組、對照組采用TRIzol提取總RNA,按照說明擴增、標記熒光、純化、與芯片雜交、洗脫。用Agilent Scanner G2505C掃描每張芯片的的信號強度,應用Agilent Feature Extraction軟件(version 11.0.1.1)分析芯片圖像文件,用GeneSpring GX v11.5.1軟件(Agilent)進行數據處理分析,根據基因表達水平篩選出差異2倍以上的基因并采用GATHER軟件[1](http://gather.genome.duke.edu/)進行生物信息學分析富集基因本體(gene ontology,GO)和生物通路。
1.2.3 差異表達基因蛋白互作網絡分析STRING (http://string.db.org/)軟件[2]從各種資源中整合了已知的和預測的蛋白質相互作用(包括直接和間接作用)來生成網絡,其數據來自于基因組、高通量實驗、共表達、已有知識4種資源。本研究采用此數據庫資源對上述差異表達基因進行蛋白互作網絡構建。
2 結果
2.1 間質干細胞C3H10T1/2成骨分化鑒定
間質干細胞C3H10T1/2經rhBMP.2成骨誘導分化7 d后,堿性磷酸酶染色顯示:rhBMP.2誘導組呈藍紫色(圖1),這表明rhBMP.2能誘導C3H10T1/2間質干細胞定向成骨分化。此細胞模型可用于后續實驗研究。

圖1 C3H10T1/2間質干細胞成骨誘導7 d堿性磷酸酶染色(100×)
2.2 C3H10T1/2基因表達譜生物信息學分析
通過對C3H10T1/2成骨分化1 d和4 d芯片結果進行分析,篩選出兩個時間點表達均升高2倍以上的基因42個;表達均降低2倍以上的基因45個(表1)。采用生物軟件進行分子功能基因本體和通路分析發現,顯著性差異分子功能本體為發育、器官形成等,并與細胞因子.細胞因子受體作用通路相關。

表1 C3H10T1/2細胞成骨分化早期差異表達基因集
2.3 一致性差異表達基因編碼蛋白相互作用網絡分析
采用STRING網絡資源對差異表達基因編碼蛋白進行網絡分析,構建了部分基因的基因表達調控與蛋白互作網絡,通過對網絡進行分析(圖2),發現經rhBMP.2誘導后差異表達的Egfr、Cxcl12等信號分子處于蛋白網絡重要交叉點,可能與信號通路交談有關,說明在rhBMP.2誘導C3H10T1/2間質干細胞早期成骨分化過程中,Egfr、Cxcl12等信號分子參與調控BMP.2信號。
3 討論
間質干細胞成骨分化是一個多基因參與的,多步驟的復雜的生物過程。機體一些重要的轉錄因子如Runx2、Dlx2等[3.4]以及眾多的信號通路在成骨分化起到非常重要調控作用[5]。但是,關于BMP.2誘導間質干細胞早期成骨定向分化相關調控機制以及不同信號間的交談分子,目前尚未完全闡明。
基因芯片技術提供了一個用于同時研究一個細胞或組織全基因組表達的較好平臺。本研究對rhBMP.2誘導C3H10T1/2成骨分化1 d和4 d后的基因表達譜進行分析,提取了rhBMP.2誘導后差異表達的基因共87個,其中上調基因42個,下調基因45個。通過文獻分析發現,多個基因已經報道與成骨分化緊密相關,它們在成骨定向、分化、轉分化過程中發揮著重要作用,如Frizzled家族成員FZD9[6]、同源蛋白家族基因Dlx2[7]、轉錄因子ID4[8]、細胞外基質相關基因Ecm1[9]等。其他差異基因,將成為筆者研究BMP.2成骨信號的重要切入點。同時,進行分子功能相關基因本體分析發現,在早期成骨分化過程,富集得分最高的為發育、器官形成、細胞因子.細胞因子受體作用相關信號,這可能與BMP.2誘導成骨分化密切相關。
復雜網絡理論作為一門新興學科,為系統水平上研究生物網絡提供了新的理論依據和平臺。2000年Jeong等[10]在《Nature》上第一次發表利用復雜網絡理論研究代謝網絡的拓撲特性的論文,自此以后,利用復雜網絡理論研究各種生物網絡迅速發展[11.12],主要集中在探索活細胞中所有分子的行為和它們之間的相互作用,以及從局部到整體,生物體如何形成了極其復雜的動力學機制。蛋白.蛋白互作網絡和轉錄調控網絡是生物信息調控的主要實現方式,在整個生命過程中有著非常重要的作用,是決定細胞命運的關鍵因素。通過對差異基因構建蛋白互作和轉錄調控網絡,本研究發現EGFR、CXCL12等信號分子在rhBMP.2誘導間質干細胞早期成骨分化網絡中處于非常重要的網絡節點。研究表明,EGFR信號參與骨原始細胞群維持,成骨分化平衡調控[13],并與前體成骨細胞活性和增殖緊密相關[14]。最近的研究也表明,CXCL12/ CXCR4信號參與骨形成和骨吸收的調控[15],在BMP.9誘導的間質干細胞成骨分化的早期和中期,CXCL12/ CXCR4信號軸也發揮重要作用[16]。本研究提示EGFR、CXCL12信號可能與BMP.2信號“交談”,參與調控BMP.2信號,因此進一步探索這些分子在成骨分化平衡調控中的作用機制,具有重要的意義。同時,趨化因子(C.C模體)配體成員CCL2、CCL7等在網絡中也處于非常重要交叉節點,是否參與BMP.2信號調控成骨分化,筆者正在研究中。
綜上所述,本研究通過對C3H10T1/2成骨分化早期基因表達譜進行分析,找到了一些受rhBMP.2調控表達的基因,這將是BMP.2成骨信號研究的重要切入點。同時,通過網絡生物學分析發現,EGFR、CXCL12等可能參與BMP.2信號調控,控制著間質干細胞骨向分化。
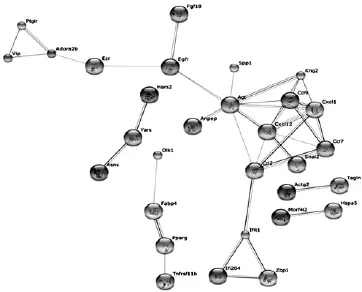
圖2 STRING顯示成骨早期部分差異基因蛋白互作網絡圖
[1]Chang JT,Nevins JR.GATHER:a systems approach to interpreting genomic signatures[J].Bioinformatics,2006,22 (23):2926.2933.
[2]Franceschini A,Szklarczyk D,Frankild S,et al.STRING v9.1:protein.protein interaction networks,with increased coverage and integration[J].Nucleic Acids Res,2013,41 (Database issue):808.815.
[3]Karsenty G.Minireview:transcriptional control of osteoblast differentiation[J].Endocrinology,2001,142(7):2731.2733.
[4]Lian JB,Stein GS,Javed A,et al.Networks and hubs for the transcriptional control of osteoblastogenesis[J].Rev Endocr Metab Disord,2006,7(1.2):1.16.
[5]Zuo C,Huang Y,Bajis R,et al.Osteoblastogenesis regulation signals in bone remodeling[J].Osteoporos Int,2012,23(6):1653.1663.
[6]Albers J,Schulze J,Beil FT,et al.Control of bone formation by the serpentine receptor Frizzled.9[J].J Cell Biol,2011,192(6):1057.1072.
[7]Harris SE,Guo D,Harris MA,et al.Transcriptional regulation of BMP.2 activated genes in osteoblasts using gene expression microarray analysis:role of Dlx2 and Dlx5 transcription factors[J].Front Biosci,2003,8:1249.1265.
[8]Tokuzawa Y,Yagi K,Yamashita Y,et al.Id4,a new candidate gene for senile osteoporosis,acts as a molecular switch promoting osteoblast differentiation[J].PLoS Genet,2010,6(7):1001019.
[9]Mongiat M,Fu J,Oldershaw R,et al.Perlecan protein core interacts with extracellular matrix protein 1(ECM1),a glycoprotein involved in bone formation and angiogenesis[J]. J Biol Chem,2003,278(19):17491.17499.
[10]Jeong H,Tombor B,Albert R,et al.The large.scale organizationofmetabolicnetworks[J].Nature,2000,407(6804):651.654.
[11]Barabasi AL,Oltvai ZN.Network biology:understanding the cell's functional organization[J].Nat Rev Genet,2004,5(2):101.113.
[12]Rual JF,Venkatesan K,Hao T,et al.Towards a proteome. scale map of the human protein.protein interaction network[J].Nature,2005,437(7062):1173.1178.
[13]Zhu J,Shimizu E,Zhang X,et al.EGFR signaling suppresses osteoblast differentiation and inhibits expression of master osteoblastic transcription factors Runx2 and Osterix[J].J Cell Biochem,2011,112(7):1749.1760.
[14]Chandra A,Lan S,Zhu J,et al.Epidermal growth factor receptor(EGFR)signaling promotes proliferation and survival in osteoprogenitors by increasing early growth response 2(EGR2)expression[J].J Biol Chem,2013,288 (28):20488.20498.
[15]Shahnazari M,Chu V,Wronski TJ,et al.CXCL12/CXCR4 signaling in the osteoblast regulates the mesenchymal stem cell and osteoclast lineage populations[J]. FASEB J,2013,27(9):3505.3513.
[16]Liu C,Weng Y,Yuan T,et al.CXCL12/CXCR4 signal axis plays an important role in mediating bone morphogenetic protein 9.induced osteogenic differentiation of mesenchymal stem cells[J].Int J Med Sci,2013,10(9):1181.1192.
Identification and analysis of gene expression profiles for early osteoblastic differentiation on C3H10T1/2 mesenchymal stem cells induced by rhBMP-2
ZUO Changqing1WANG Zonggui2ZHONG Yuechun1LU Hanyun1DAI Zhong1WU Tie1CUI Liao1l.Department of Pharmocology,Guangdong Medical College,Guangdong Province,Dongguan523808,China; 2.Department of Biochemistry,Guangdong Medical College,Guangdong Province,Dongguan523808,China
Objective To investigate the gene expression profiles associated with early osteoblastic differentiation on mesenchymal stem cells,and to provide an experimental basis for the study of gene regulation mechanism for early osteoblastic differentiation.Methods The total RNA was extracted from C3H10T1/2 mesenchymal stem cells between control and rhBMP.2 osteoblastic differentiation group,and then cRNA was labeled with fluorescence labels and hybridized with ArraySTAR mouse genechip.The gene expression profiles were analyzed with bioinformatics software GeneSpring and GATHER.The protein interaction network of differentially expressed genes was constructed and analyzed by online software STRING.Results The molecular function gene ontology(GO)including development and organogenesis and cytokine.cytokine receptor interaction signaling pathway were mainly enriched during C3H10T1/2 early osteogenic differentiation.A total of 42 differentially expressed genes were upregulated and 45 genes were downregulated from rhBMP.2 induced osteoblast 1 and 4 days.Protein network analysis showed that Egfr,Cxcl12 were very important signaling molecules which involved in rhBMP.2 induced osteoblast.Conclusion The screening differentially expressed genes and signaling molecules have an important role for regulation of early osteoblastic differentiation and provide further experimental basis for comprehensive analysis of early osteoblastic differentiation.
Early osteoblastic differentiation;Gene expression profiles;Mesenchymal stem cells;Bioinformatics
R966;R963;R336;R329
A
1673-7210(2014)02(a)-0025-04
2013.11.28本文編輯:程銘)
國家自然科學基金項目(編號81101357);廣東省中醫藥局課題(編號20112145);廣東省東莞市高等院校科研機構科技項目(編號2011108102029);廣東省湛江市科技攻關項目(編號2010C3104003)。
左長清(1977.),男,博士;研究方向:干細胞成骨成脂分化調控,生物信息學。

