門多薩假單胞菌Pseudomonas mendocina P10脂肪酶基因的克隆及其在酵母中的表達
王建榮,陳麗芝,羅長財,鐘開新,聶金梅,李陽源
(廣東溢多利生物科技股份有限公司,廣東珠海519060)
脂肪酶(Lipase,E.C.3.1.1.3),全稱為三酰基甘油酰水解酶(triacylglycerol acylhydrolase),它屬于α/β折疊酶家族,是一種絲氨酸水解酶。脂肪酶催化三酰甘油的酯鍵水解,釋放更少酯鍵的甘油酯或甘油及脂肪酸[1]。脂肪酶主要來源于動植物和微生物中,相對于動植物,微生物分泌的脂肪酶具有更廣的底物專一性、作用溫度以及作用pH范圍。脂肪酶在微生物界分布很廣,目前已發現曲霉[2]、根霉[3]、假單胞菌[4]等多種微生物可產脂肪酶。在眾多來源的脂肪酶中,來源于假單胞菌屬的脂肪酶廣泛應用于各個工業領域,包括食品工業、洗滌工業、手性拆分和藥物合成等領域中。如門多薩假單胞菌的脂肪酶用于洗滌劑添加劑、熒光假單胞菌和銅綠假單胞菌的脂肪酶則被廣泛用于有機合成等[5]。由于來源于假單胞菌的脂肪酶具有良好的應用特性,為了進一步提高假單胞菌分泌脂肪酶的能力,許多學者開始應用分子生物學的方法研究其脂肪酶基因的克隆、表達、調控等。到目前為止已有多種假單胞菌的脂肪酶基因被克隆并且實現了異源表達,如熒光假單胞菌[6]、銅綠假單胞菌[7]、洋蔥勃克霍爾德菌[8]等。本研究采用同源克隆法得到了門多薩假單胞菌的脂肪酶基因并對該基因進行了分析,同時將該基因轉入畢赤酵母X33中,進行了初步的發酵實驗,為進一步研究該基因奠定了基礎。
1 材料與方法
1.1 材料與儀器
表達載體pPICZαA、Pichia pastoris X-33 購自Invitrogen公司;門多薩假單胞菌Pseudomonas mendocina P10 購自中國工業微生物菌種保藏管理中心;大腸桿菌Top10、高保真Pfu酶、T4 DNA連接酶、限制性內切酶EcoRI、NotI、質粒提取試劑盒、細菌基因組提取試劑盒、PCR純化試劑盒 均購自上海生工公司;PCR引物合成以及DNA測序 均由深圳華大基因公司完成;其他生化試劑 均為國產分析純。
Biometra熱密閉PCR儀T-1 德國Biometra公司;Bio-Rad凝膠成像系統 美國Bio-Rad公司;Nano Drop分光光度計ND-1000 美國Nano Drop公司;電泳儀DYY-6C 北京市六一儀器廠。
1.2 實驗方法
1.2.1 門多薩假單胞菌基因組DNA的提取 挑取單菌落接入50mL LB液體培養基中,30℃,150r/min培養過夜,離心取菌體,參照細菌基因組DNA提取試劑盒說明書提取基因組DNA。用Nano Drop分光光度計(ND-1000)測定DNA的濃度,選取A260/A280值在1.8~2.0的樣品,置于冰箱中-20℃保存備用。
1.2.2 脂肪酶基因的克隆 經過分析NCBI上已報道的Pseudomonas mendocina NK-01的全基因組DNA序列(Genebank登錄號為:CP002620.1),設計了一對引物,上游引物:5’-ATCGGAATTCATGGCCAGTCTTCC GCTGTAT-3’和下游引物:5’-ATATGCGGCCGCCTA GCCCTTCATCGGCGCATC-3’(分別含有EcoRI和NotI酶切位點)。以提取的門多薩假單胞菌基因組DNA為模板進行PCR擴增。PCR反應體系:DNA模板1μL(約50ng),上、下游引物各2μL,2×Pfu Master Mix 50μL,用ddH2O補充到總體積為100μL。PCR反應條件:94℃預變性5min后,分別于94℃變性50s,58℃退火50s和72℃延伸1min,共33個循環,最后在72℃延伸10min。PCR產物經凝膠電泳鑒定正確后切膠純化存于冰箱中-20℃備用。
1.2.3 表達載體pPICZαA-pml的構建 pPICZαA質粒和目的基因PCR產物都用EcoRI和NotI進行雙酶切,切膠純化后,用T4DNA連接酶16℃過夜連接酶切產物,然后用CaCl2轉化法轉入E.coli Top10,在含25μg/mL Zeocin的LB培養基平板上涂板,過夜培養。挑取陽性轉化子過夜培養后提取質粒,進行雙酶切鑒定。將陽性質粒提交華大基因公司進行序列測定。
1.2.4 序列分析 核酸和氨基酸序列的比對在NCBI BLAST(http://www.ncbi.nlm.nih.gov)上進行,核酸序列分析用DNAman 5.0完成,蛋白結構與功能的預測在Swissprot(http://www..expasy.ch/sprot)網站上提供的鏈接的平臺上進行。
1.2.5 重組畢赤酵母的構建、誘導表達以及酶活檢測 重組質粒用限制性內切酶SacI進行線性化后,使用電轉化法轉化到畢赤酵母X-33,取轉化產物涂布于含有100μg/mL的Zeocin YPD平板上,30℃培養2~3d,進行菌落PCR鑒定。挑取經菌落PCR鑒定的轉化子,接種于含有50mL BMGY培養基的250mL三角瓶中,30℃,250r/min振蕩培養16~20h至OD600達到2~6。離心收集菌體,再將其重懸浮于BMMY培養基中,稀釋至OD600為0.8~1.0,繼續振蕩培養,每隔24h向BMMY培養基中補加甲醇至終濃度為1.0%進行誘導表達,同時測定重組菌的菌體濃度和脂肪酶酶活。脂肪酶活性定量測定采用堿滴定法,具體步驟參照文獻[9]中報道的方法。
2 結果與分析
2.1 門多薩假單胞菌脂肪酶基因pml的克隆及序列比對
以提取的門多薩假單胞菌基因組DNA為模板,用設計的引物進行PCR擴增,經瓊脂糖凝膠電泳可見PCR產物大小約為820bp,大小與理論值相符(見圖1)。將該目的片段回收并克隆入表達載體pPICZαA,提取重組質粒進行EcoRI和NotI雙酶切鑒定,片段大小正確(見圖2),測序結果表明,pml基因全長為801bp,起始和終止密碼子分別為ATG和TAG,推測其完整的ORF編碼267個氨基酸。利用NCBI網站上的BLAST軟件分析表明,推測的門多薩假單胞菌脂肪酶的氨基酸序列同其他已知細菌的脂肪酶氨基酸序列有較高的相似性。其中最高的為Pseudomonas mendocina NK01(相似性為97%),其次為Pseudomonas mendocina DLHK(相似性為82%)。采用DNAman 5.0進行同源比對分析(見圖3),發現門多薩假單胞菌脂肪酶基因pml的氨基酸序列中含有高度保守的“Gly-X-Ser-X-Gly”五肽功能元件。研究表明,該功能元件在脂肪酶識別和結合底物方面發揮著重要的作用[10]。同時在該脂肪酶氨基酸序列中還發現了構成脂肪酶活性中心催化活性位點的三個氨基酸殘基(Ser-Asp-His),它們分別位于156、212、247位。通過以上分析表明從門多薩假單胞菌中克隆得到了完整的脂肪酶基因。

圖1 PCR擴增電泳圖Fig.1 Electrophoretogram of PCR

圖2 重組質粒的酶切鑒定Fig.2 Digestion and identification of recombinant plasmid
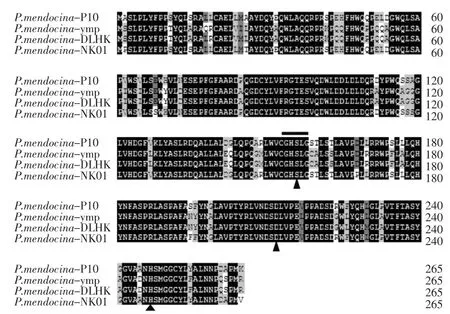
圖3 脂肪酶氨基酸序列多重比對圖Fig.3 The multiplex alignment of the lipase
2.2 門多薩假單胞菌脂肪酶基因pml生物信息學分析
門多薩假單胞菌脂肪酶基因pml編碼297個氨基酸,利用在線軟件Protparam預測表明,該脂肪酶的等電點和相對分子量大小分別為4.82、29.95ku,半衰期理論值為30h,不穩定參數為61.80,屬于不穩定蛋白質。利用在線網站SinglelP4.0預測信號肽定位,結果顯示該氨基酸序列不含有信號肽。采用在線軟件ProtScale分析此蛋白的疏水性和親水性,發現第21位的丙氨酸和22位的谷氨酸的疏水性最強(分值為2.189),位于第41位的精氨酸親水性最強(分值為-2.967)。用SOPMA對此蛋白質的二級結構進行了預測,在該蛋白質的二級結構中無規則卷曲的含量最大為47.74%,其次為α螺旋含量為34.59%,延伸鏈的含量為15.41%,而β轉角的含量最少為2.26%。通過NCBI網站上的conserve domain對該氨基酸序列進行保守結構域分析,結果顯示該序列屬于脂肪酶超家族中的第三類脂肪酶。
2.3 重組畢赤酵母的構建以及篩選
Zeocin抗性平板上長出數百個轉化子(見圖4),隨機挑取單菌落進行菌落PCR,陰性對照和陽性對照分別以畢赤酵母X-33的基因組DNA和構建好的脂肪酶表達載體為模板進行PCR擴增,結果如圖5所示。重組菌和陽性對照都能擴增出一條820bp大小的條帶,與預期大小相符,表明重組質粒已成功整合到了畢赤酵母基因組中。

圖4 擬轉化子Fig.4 Putative transformants

圖5 擬轉化子PCR鑒定Fig.5 PCR detection of putative transformants
2.4 重組轉化子脂肪酶基因的誘導表達及酶活測定
按照方法“1.2.5”對篩選的重組畢赤酵母進行誘導培養,每隔24h取樣測量發酵液的菌體濃度和上清發酵液的脂肪酶酶活。結果如圖6所示,誘導到168h時,菌體濃度達到最高,菌體濃度為432g/L;誘導至192h時,酶活力達到最大為8U/mL。

圖6 重組畢赤酵母生長曲線和產酶曲線Fig.6 Cell growth and lipase production of recombinant yeast
3 結論
采用同源克隆法從門多薩假單胞菌中克隆得到脂肪酶基因,該基因全長801bp,編碼267個氨基酸。通過對該脂肪酶基因的序列及其編碼的氨基酸進行分析發現:該脂肪酶基因與已報道的脂肪酶基因具有很高的相似性,相似性最高可達97%;同時在該脂肪酶氨基酸序列中還存在“Gly-X-Ser-X-Gly”五肽功能元件以及脂肪酶活性中心催化活性位點的三個氨基酸殘基(Ser-Asp-His),通過保守區域分析,該脂肪酶適于脂肪酶超家族中的第三類脂肪酶。將該脂肪酶基因連接到pPICZαA表達載體上轉化Pichia pastoris X-33,脂肪酶基因在Pichia pastoris X-33中實現了分泌性表達。當誘導培養時間誘導到168h時,菌體濃度達到最高,菌體濃度為432g/L;誘導至192h時,酶活力達到最大為8U/mL。
[1]Jaeger K E,Eggert T.Lipases for biotechnology[J].Curr Opin Biotechnol,2002,13(4):390-397.
[2]Shi B,Zeng L,Song H,et al.Cloning and Expression of Aspergillus tamarii FS132 Lipase Gene in Pichia pastoris[J].Int J Mol Sci,2010,11(6):2373-2382.
[3]顏興和,王棟,徐巖.根霉脂肪酶的研究進展[J].工業微生物,2005,35(3):45-49.
[4]張博,楊江科,閆云君.假單胞菌脂肪酶的研究進展[J].生物技術通報,2007,2:52-57.
[5]Gupta R,Gupta N,Rathi P.Bacterial lipases:an overview of production,purification and biochemical properties[J].Appl Microbiol Biotechnol,2004,64(6):763-781.
[6]Chung GH,Lee YP,Jeohn GH,et al.Cloning and nucleotide sequence ofthermostable lipase gene from Pseudomonas fluorescens SIK W1[J].Agric Biol Chem,1991,55(9):2359-2365.
[7]Ogino H,Hiroshima S,Hirose S,et al.Cloning,expression and characterization of a lipase gene(lip3) from Pseudomonas aeruginosa LST-03[J].Mol Genet Genomics,2004,271(2):189-196.
[8]Jorgensen S,Skov KW,Diderichsen B.Cloning,sequence,and expression of a lipase gene from Pseudomonas cepacia:lipase production in heterologous hosts requires two Pseudomonas genes[J].J Bacteriol,1991,173(2):559-567.
[9]Saxena R K,Davidson W S,Sheoran A,et al.Purification and characterization of an Alkaline thermostable lipase from Aspergillus carneus[J].Process Biochem,2003,39(2):239-247.
[10]Tan Y,Miller K J.Cloning,expression,and nucleotide sequence of a lipase gene from Pseudomonas fluorescens B52[J].Appl Environ Microbiol,1992,58(4):1402-1407.

