基于自適應核學習相關向量機的乳腺X線圖像微鈣化點簇處理方法研究*
姚暢 陳后金 Yang Yong-Yi 李艷鳳 韓振中 張勝君
1)(北京交通大學電子信息工程學院,北京 100044)
2)(Department of Electrical and Computer Engineering,Illinois Institute of Technology,Chicago IL 60616,USA)
(2012年12月1日收到;2013年1月29日收到修改稿)
1 引言
乳腺癌是婦女發病率和死亡率最高的惡性腫瘤之一,乳腺癌的早期發現和診斷是提高治愈率和降低死亡率的關鍵[1,2].目前,應用于乳腺癌診斷的檢測方法有:X射線影像技術、計算機斷層攝影術、光聲成像、核磁共振成像、微波成像等技術[3-9].其中,乳腺X線影像技術被認為是最可靠和最有效的方法[9].放射科醫師利用它可以發現乳腺癌觸診而不能發現的早期微小病灶——微鈣化點.在乳腺X線圖像中,微鈣化點是以小亮點出現的鈣沉淀.通常單個的微鈣化點對于乳腺癌的檢測沒有太大的意義,聚合成簇出現的微鈣化點才是早期乳腺癌的重要癥兆(如圖1所示).研究表明,30%—50%確診的乳腺癌患者在早期的乳腺X線圖像篩查中有微鈣化點簇出現[10].因此,乳腺X線圖像中微鈣化點簇的分析處理對乳腺癌的早期防治具有非常重要的意義.

圖1 乳腺X線圖像(左)及其含有的一個微鈣化點簇的放大圖(右)
由于微鈣化點在形狀、大小、分布上都有很大變化性,且其灰度與周圍乳腺組織灰度接近,所以微鈣化點的檢測比較困難.目前,關于微鈣化點的檢測方法已有不少文獻報道,主要可分為:圖像增強方法、隨機建模方法、多尺度分解方法和機器學習方法[8-13].其中,基于機器學習的微鈣化點檢測方法能獲得較好的微鈣化點檢測性能,是當前微鈣化點簇處理方法研究的主要方向.Naqa等[12]將支持向量機(support vector machine,SVM)應用于微鈣化點檢測,采用連續增強學習方法訓練分類器以進一步提高預測能力,實驗結果證明了支持向量機方法的檢測性能要優于神經網絡方法、圖像差分方法和小波多尺度分解方法.Wei等[13]將相關向量機(relevance vector machine,RVM)應用于微鈣化點簇處理,通過實驗仿真證明了RVM在獲得與SVM相同的性能情況下,分類器的計算復雜度較SVM大幅下降,有利于臨床應用.但是,由于RVM最終獲得的相關向量全部來自訓練集中的訓練樣本,且模型采用的核函數的參數只能通過對訓練集樣本采用交叉驗證方法來人工設定,從而導致RVM算法性能的進一步提高受到了影響.
2009年Tzikas等[14]提出了一種基于增量學習的稀疏貝葉斯建模方法,即自適應核學習相關向量機(adaptive kernel learning based relevance vector machine,ARVM)方法.在原理上,ARVM與RVM相似.然而,在RVM方法中,相關向量來自訓練集中的訓練樣本,且模型采用的核函數的參數只能通過對訓練集采用交叉驗證方法來確定.而在ARVM方法中,核函數參數由模型在訓練時自動優化設置,模型核函數允許同時采用不同類型的基函數構成,更適應數據的局部特征.為避免過擬合,模型采用了一種稀疏度先驗方法控制模型有效參數的個數,從而使得模型比RVM更稀疏.
基于此,本文嘗試性地采用ARVM用于乳腺X線圖像微鈣化點簇分析,研究了基于ARVM的乳腺X線圖像微鈣化點簇處理方法.該方法首先將微鈣化點檢測看作一個二分類的監督學習問題,運用ARVM作為分類器判斷圖像中每一個位置是否為微鈣化點;然后采用形態學處理去除干擾噪聲并對判別出的微鈣化點進行分簇.此外,為提高算法的運算速度,實現了一種基于圖像分塊并行運算的ARVM微鈣化點簇快速處理方法.
2 ARVM理論
ARVM是在RVM的基礎上提出的一種核函數參數自適應學習的稀疏貝葉斯建模方法.相比較于RVM,其最大的特點是模型核函數參數在訓練時自動優化設置,且模型核函數允許同時采用不同類型的基函數構成.同時,模型采用稀疏度先驗方法控制模型有效參數的個數,從而使得模型比RVM更稀疏.基于這些特性,ARVM能很好地解決回歸和分類問題.本文采用ARVM來解決二分類問題.
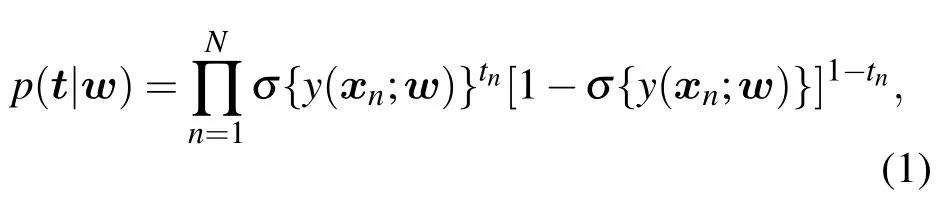
其中
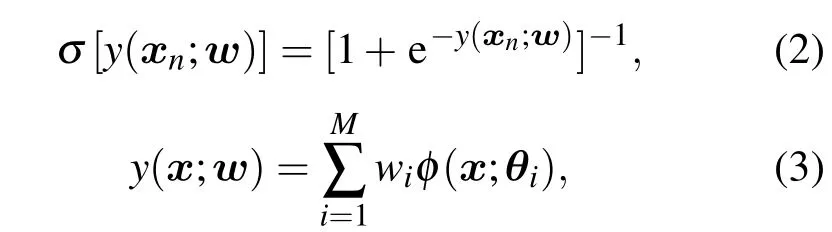
w=(w1,w2,···,wM)T為模型的權值,φ(x;θi),(i=1,···,M)是參數為 θ =(θ1,θ2,···,θM)T的核基函數集.
對于模型權值w的分布,采用不同精度的高斯先驗αi進行控制,
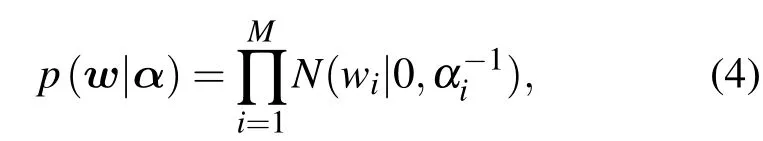
其中 α =(α1,···,αM)T.
對于模型每個噪聲εn,則為具有不同精度βn的高斯噪聲:

利用貝葉斯定理,權值的后驗分布概率為
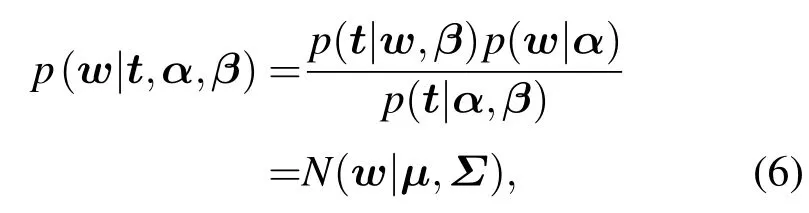
在該算法中,盡管α和β被引入先驗分布,但是并不計算這些隱藏變量的聯合后驗概率而是只計算權值后驗概率p(w|t,α,β),接著計算出后驗概率 p(α,β|t)∝p(t|α,β)p(α,β)最大值時的 α 和 β 的最大后驗概率(maximum a posteriori,MAP).由于假設α和β的無信息先驗,∫更新公式可以通過最大化邊緣似然率 p(t|α,β)= p(t|w,β)p(w|α)d w 獲得.
為了避免過擬合,不同于RVM采用伽瑪(Gamma)先驗分布的方法,ARVM對于α引入超先驗用來直接控制模型有效參數的個數:

其中,S=ΦΣΦTB稱為平滑矩陣,矩陣的跡trace(S)稱為S的自由度,表示模型參數的有效個數,c為稀疏度參數,控制模型期望稀疏度的大小.計算trace(S)得:
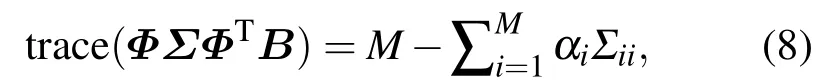
因此,稀疏度先驗式(7)可寫為
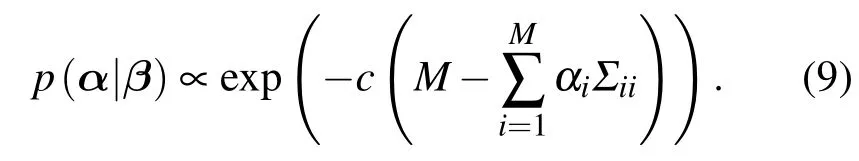
基于超先驗p(α|β),模型超參數α和β通過最大化后驗概率 p(α,β|t)∝ p(t|α,β)p(α|β)p(β)進行更新.由于協方差Σ的計算復雜度為O(N3),算法采用增量方法[15]進行學習.初始假設所有αi=∞,即假設所有基函數由于稀疏度控制為空,然后進行迭代更新,并判斷每次迭代得到的基函數是否滿足條件添加到模型或是否需要從模型刪除.當有基函數添加到模型后,相應的參數αi設定為使后驗概率最大時的值.
在增量方法中,對數化的后驗概率L中僅與參數αi相關的項為
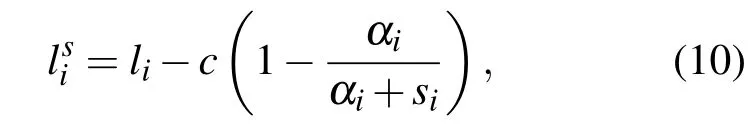
lis的梯度為
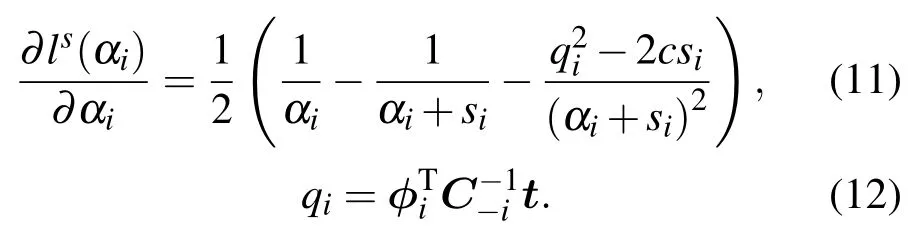
將梯度取零,從而求得lis取最大值時的αi為
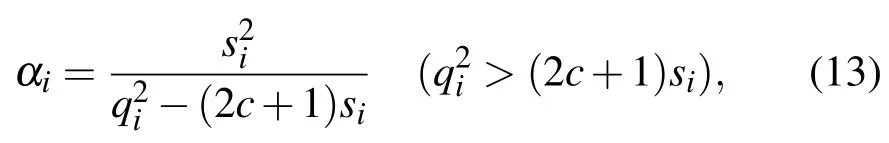
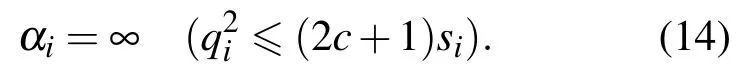
同樣,對于超參數β的更新,也是取后驗概率L對β求偏導后等于零的數值解.此外,對于模型核基函數φ(x;θi)中的不同核函數參數θi,算法通過計算后驗概率L對基函數參數θ的偏導,然后采用擬牛頓法(Broyden Fletcher Goldfarb Shanno,BFGS)求解最優值.
3 基于ARVM的微鈣化點簇處理
本文將乳腺X線圖像中的微鈣化點檢測問題看作一個二分類問題,采用ARVM算法實現對微鈣化點簇的處理.具體來說,包括微鈣化點檢測和微鈣化點分簇兩步操作.其中,微鈣化點檢測通過對預處理后的乳腺X線圖像逐個提取像素點位置的圖像特征,然后采用訓練好的ARVM分類器判斷其是否屬于“微鈣化點”類或“非微鈣化點”類來實現;微鈣化點分簇則是通過對檢測出的微鈣化點采用Kallergi標準來判斷實現.
3.1 圖像預處理
微鈣化點通常與背景中的乳腺組織重疊,且其灰度與周圍乳腺組織灰度接近,必須進行預處理抑制背景噪聲、增強對比度.本文采用文獻[13]的高通濾波方法進行預處理.通過大量實驗,高通濾波器設計為截止頻率ωc=0.125、長度為41的一維有限脈沖響應濾波器,然后采用基于麥克萊倫(McClellan)矩陣的頻率變換將其轉換為二維有限脈沖響應濾波器,如圖2所示.為降低各圖像灰度差異,濾波后的圖像進行了歸一化處理.圖3所示為高通濾波后的圖像.
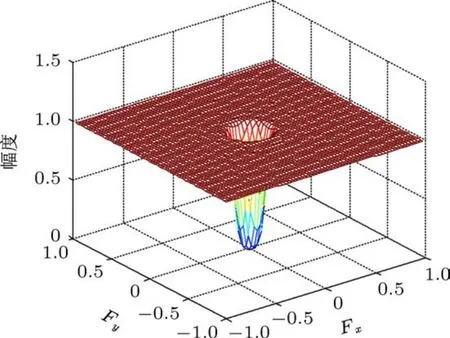
圖2 高通濾波器

圖3 高通濾波后的結果
在ARVM的訓練中,作為金標準的數據為專家手工標定的每個微鈣化點的中心位置.由于專家手工標定的微鈣化點中心不一定在微鈣化點亮度最亮的位置,為減少算法通過計算機找出的微鈣化點中心與專家標定的中心位置的誤差,我們采用線性濾波的方法校正專家手工標定的中心位置.首先設計一個大小為7×7的加權平均濾波器掩模,如圖4(a)所示;然后,對歸一化的圖像中專家標識的微鈣化點中心位置的15×15鄰域圖像進行形態學線性濾波,找出濾波后結果中的最大峰值點,將該點位置標記為校正后的微鈣化點中心,校正過程如圖4(b)所示.
3.2 特征提取
特征提取是微鈣化點檢測的關鍵步驟,特征提取的結果直接影響到分類器的檢測性能.由于微鈣化點在乳腺X線圖像中具有較好的定位性,因此,判斷圖像中一個像素點位置是否是微鈣化點時,可以通過提取其周圍鄰域內的圖像內容進行判別.本文提取乳腺X線圖像中待判別像素點位置的G×G鄰域像素構成特征向量,作為ARVM的輸入,待判別的像素點位置位于G×G窗的中心.具體來說是將每個G行G列(G×G)的圖像窗排列為一個G×G維的行向量.其中,G的選擇條件為:窗口的大小能覆蓋整個微鈣化點像素區域同時又不與鄰近的微鈣化點區域有重疊.本文采用的圖像數據庫中,乳腺X線圖像的精度為0.05 mm/pixel,通過實驗,G取經驗值15最為合適.圖5所示為預處理后的乳腺X線圖像中提取出的含有微鈣化點的特征樣本.
圖4 中心位置校正 (a)濾波器掩模;(b)中心位置校正過程

圖5 提取的含有微鈣化點的樣本
3.3 模型核函數和稀疏度
ARVM的最重要特性是模型在訓練期間能自動優化設置核函數的參數.本文實驗中主要考慮常用的多項式核函數(polynomial kernel,簡記為Poly)和高斯徑向基核函數(Gaussian RBF kernel,RBF).由于核參數的自適應連續優化是基于核參數的偏導進行的,因此,需要計算核函數對其參數的偏導.
1)多項式核函數
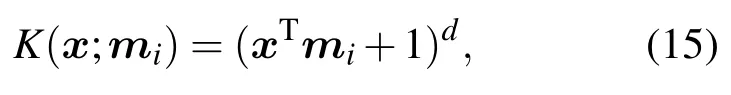
其中d為階數,d>0,mi為模型訓練過程中需要自動優化的核參數.核函數對mi求偏導:

2)高斯徑向基核函數
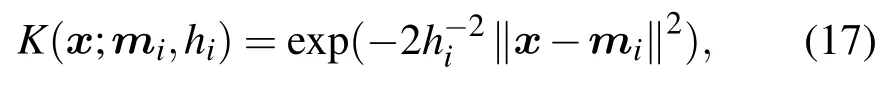
其中,均值向量mi和方差hi為模型訓練過程中需要自動優化設置的核參數.核函數分別對mi和hi求偏導:

核函數參數mi的初始值為隨機挑選的訓練樣本,然后通過擬牛頓法迭代優化,算法最終收斂獲得的mi即為模型的相關向量.
模型的復雜度采用(7)式的稀疏度先驗方法進行控制,其中稀疏度參數c根據如下常用標準取值[14]:

3.4 微鈣化點分簇
通過ARVM分類器檢測出來的微鈣化點進行后續分簇操作時,由于有些檢測出來的相互鄰近的像素點可能是同一個微鈣化點的一部分,因此需要將這些像素點合并到同一個微鈣化點.本文采用形態學方法進行處理,同時將結果中孤立的可疑像素點去除.
形態學處理后的微鈣化點采用Kallergi標準[16]劃分為不同的微鈣化點簇.該標準將檢測出的微鈣化點判別為一個真陽性(true positive,TP)簇時需要同時滿足以下兩個條件:
1)相互鄰近的目標的近鄰距離小于0.4 cm;
2)在面積為1 cm2的范圍內檢測出的微鈣化點中應至少包含有3個真正的微鈣化點.
當一組檢測出的微鈣化點滿足簇判別條件1),但不包含真正的微鈣化點時,該簇稱為假陽性(false positive,FP)簇.
4 微鈣化點簇處理方法的快速實現
訓練好的分類器對測試圖像進行微鈣化點檢測時,將15×15的窗口在圖像上滑動以提取每個像素點的特征向量進行判斷.將提取的特征向量排列為特征矩陣,然后采用分類器進行判別,判別結果再排列回原圖像位置,這樣可以節省大量的計算機運行時間.但由于圖像和特征向量太大,從而使得特征矩陣也變大,算法進行矩陣處理時由于運算量太大導致對機器內存要求變高,進而使得算法執行速度下降.為此,本文提出一種圖像分塊的處理方法.該方法將預處理后的待檢測乳腺圖像劃分為J×J塊子圖像;然后分別提取每個子塊中各像素點的特征向量,即提取每一個像素點的15×15鄰域并排列成225維的行向量,將子塊中各特征向量依次排列為待識別的特征向量矩陣,交由訓練好的ARVM分類器進行判斷,這一步操作也可以通過計算機并行運算快速實現;最后,將J×J塊子圖像的判斷結果合并為整幅圖像的識別結果.分塊方法在提高算法的執行速度的同時不改變算法的檢測結果.圖6所示為J取4時的圖像分塊檢測方法示意圖.
將圖像分塊和ARVM應用于乳腺X線圖像微鈣化點簇處理,本文方法流程如下:
步驟1 利用訓練樣本集訓練ARVM.
1)讀入訓練乳腺圖像,進行高通濾波和歸一化預處理;
2)對預處理后的圖像校正專家手工標定的微鈣化點中心坐標;
3)對中心坐標校正后的圖像提取訓練樣本特征向量,并將其排列為訓練樣本特征矩陣;
4)利用獲得的訓練樣本特征矩陣訓練ARVM.

圖6 圖像分塊方法示意圖
步驟2 利用訓練好的ARVM識別測試圖像中的微鈣化點.
1)讀入待測試乳腺圖像,進行高通濾波和歸一化預處理;
2)對預處理后的圖像采用分塊方法將圖像分為J×J個子塊圖像;
3)依序每次對其中一個子塊圖像提取其待測試特征向量,并將其排列為待測特征矩陣;
4)采用訓練好的ARVM對待測試特征矩陣進行判斷,獲得識別的可疑微鈣化點;
5)重復步驟2中第3),4)步,將獲得的各子塊圖像檢測結果合并為整幅圖像的檢測結果.
步驟3 對獲得的可疑微鈣化點識別結果進行形態學后處理,去掉干擾噪聲.
步驟4 對微鈣化點進行分簇,獲得最終的微鈣化點簇處理結果.
5 實驗仿真及結果分析
實驗采用美國芝加哥大學放射系臨床采集的數據庫[13]來檢驗算法的有效性.該數據庫由66個臨床病例共141幅圖像組成,每幅圖像包含有一個或多個后期經臨床確診的微鈣化點簇.每幅圖像的大小為3000×5000像素,圖像分辨率為0.05 mm/pixel,10位灰度.同時,數據庫提供了每幅圖像經由一組有經驗的放射科專家手工標定的微鈣化點信息以及每幅圖像中乳腺區域的二值掩模.為節省計算時間,與文獻[13]方法一樣,提取每幅圖像中包含專家標定的微鈣化點在內的800×800像素區域進行實驗.
5.1 ARVM訓練
實驗將圖像庫隨機分為兩個獨立的子集,每個子集包含33個病例.其中,來自同一個病例不同視角的乳腺X線圖像屬于同一個子集.將其中一個子集用于訓練ARVM分類器(33個病例,71幅圖像),另一個子集用于測試分類器(33個病例,70幅圖像).這樣,來自同一個病例的所有乳腺X線圖像將要么只用于訓練,要么只用于測試,不會同時用于模型的訓練和測試.
訓練圖像集包含的乳腺X線圖像中總共有1578個微鈣化點.對于每個微鈣化點,提取其中心位置的15×15鄰域窗,并將窗內所有像素點值排列為一個1行225列的行向量,表示為xi.xi即為訓練樣本中類別標簽為“微鈣化點出現”(ti=+1)的正樣本.總共提取有1578個正樣本.同樣地,提取2倍于有微鈣化點出現樣本數的背景像素窗排列為“無微鈣化點出現”(ti=-1)的負樣本(3422個).無微鈣化點出現的負樣本是從訓練集圖像的背景中隨機提取,且不與其他任意樣本窗有重疊.圖7所示為從訓練集提取的部分訓練樣本.
提取完訓練樣本集后,需要確定ARVM所采用的核函數類型以及模型稀疏度參數c的取值才能對分類器進行訓練.本實驗采用5折交叉驗證(5-fold crossvalidation)方法來確定.
交叉驗證[17]是一種統計學上將數據樣本切割成較小子集,先在部分子集上做分析,而后在其他子集上對此分析進行確認及驗證的實用方法.本文采用的5折交叉驗證方法為:
1)將訓練樣本隨機分為5個同樣大小的子集;
2)對于要設置的參數,從5個子集中選擇1個子集保留作為測試集,其余4個子集作為訓練集訓練模型;模型訓練好后用保留的測試集進行驗證,獲得該次的測試結果;

圖7 提取的訓練樣本 (a)“微鈣化點出現”樣本;(b)“無微鈣化點出現”樣本
3)交叉重復5次,每個子集被驗證1次,平均5次的測試結果作為該參數值的泛化誤差.
改變參數的取值,5折交叉驗證方法將獲得相應的泛化誤差,選擇最小泛化誤差對應的取值作為該參數的設定值.
表1給出了不同核函數類型和不同稀疏度值時模型的泛化誤差,其中Poly1,Poly2,Poly3,Poly4分別表示1,2,3,4階的多項式核函數;RBF表示高斯徑向基核函數.從表1可以看出,最小的泛化誤差為0.0385,其對應的核函數為2階多項式核函數,稀疏度參數為lg(N)/2.因此,ARVM中的核函數采用2階多項式核函數、稀疏度參數c=lg(N)/2.
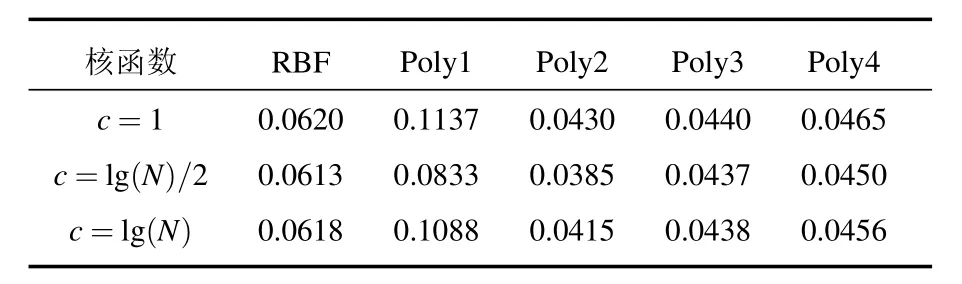
表1 不同參數值的ARVM獲得的泛化誤差
采用訓練樣本集對ARVM進行訓練,直到算法收斂,從而獲得訓練好的分類器.本實驗中當連續10次迭代每次模型后驗概率L的增量可忽略且模型權值非零個數恒定不變時,認為分類器已經訓練好.圖8所示為本實驗采用2階多項式核函數的ARVM收斂過程,由圖8可以看出,算法在迭代到第5次時就已經訓練好了分類器,訓練好的分類器相關向量個數為5,且這些相關向量不是直接來自訓練樣本集,而是模型通過增量方法自適應優化獲得,相關向量如圖9所示.
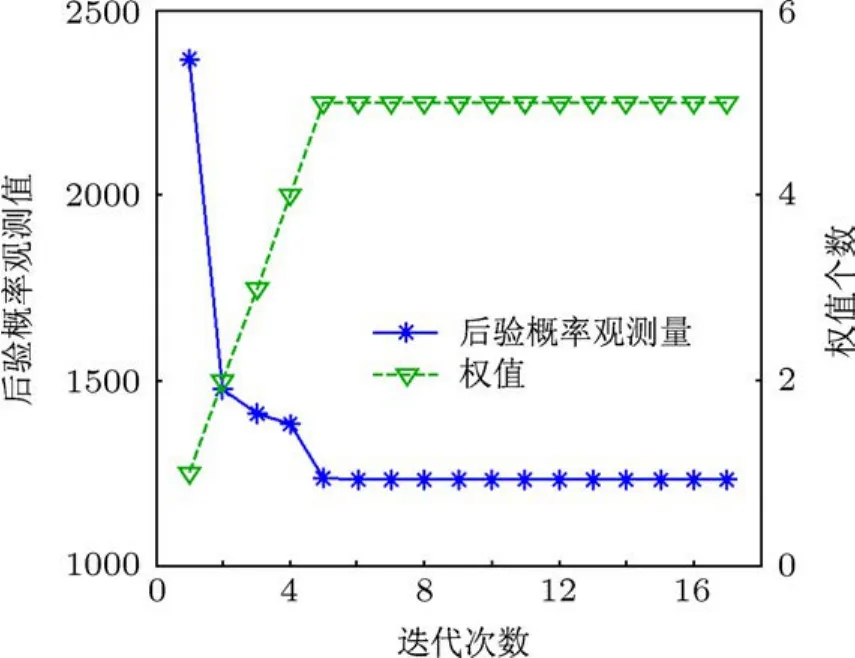
圖8 ARVM收斂過程

圖9 相關向量
5.2 處理結果及算法性能評價
圖10 所示為本文算法對圖1中局部放大圖的微鈣化點檢測結果,其中“°”表示放射科專家標出的真實微鈣化點位置,“+”表示本文算法檢測出的微鈣化點.對圖10結果進行分析可知,盡管本文算法漏檢了兩個真實微鈣化點,且多檢出了兩個假陽性微鈣化點,但是整個檢測出的微鈣化點目標滿足Kallergi微鈣化點簇判別標準,因此成功地檢測出了該微鈣化點簇.
為比較算法性能,實驗同時將文獻[13]中的RVM算法采用相同的測試集進行比較.實驗結果采用自由響應受試者工作特征(free-response re-ceiver operating characteristic,FROC)[18]曲線進行分析和評價.FROC曲線通過計算真陽性簇的正確率(也叫真陽性率)和平均每幅圖像的假陽性簇個數,并將不同閾值下這兩個檢測量的統計值繪制在同一個圖中來評價算法性能.曲線越接近圖的左上角,說明算法對微鈣化點簇的處理性能越好.實驗通過改變判別微鈣化點的閾值來繪制算法處理結果的FROC曲線.圖11所示為近鄰距離Dnn=0.4 cm時,本算法ARVM和文獻[13]的RVM算法對微鈣化點簇處理結果的FROC曲線圖.

圖10 檢測出的微鈣化點,“○”表示專家標出的真實微鈣化點,“+”表示本文算法檢測出的微鈣化點
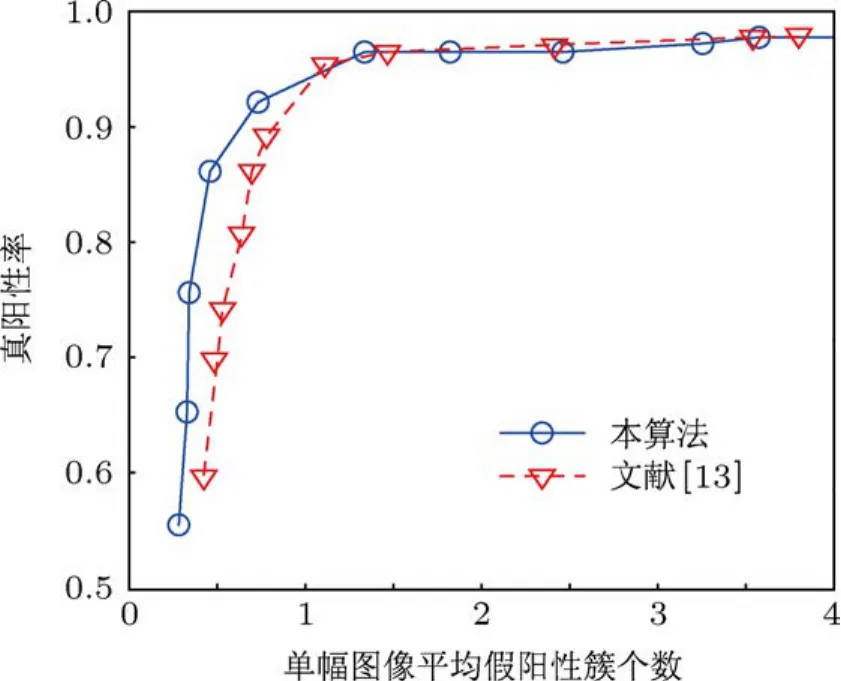
圖11 D nn=0.4 cm時的FROC曲線
由圖11可以看出,當單幅圖像平均假陽性簇個數在[0,1]區間時,本文ARVM算法的真陽性率要高于文獻[13]的RVM算法;單幅圖像平均假陽性簇個數在[1,4]區間時,ARVM與RVM的真陽性率接近.臨床采用計算機輔助診斷系統提供輔助信息時,檢測出的每幅圖像平均假陽性簇個數如果太高則對輔助診斷意義不大,醫生主要關注每幅圖像平均假陽性簇個數小于1時的真陽性簇檢測結果.本文實現的ARVM算法正是在這個區間算法性能要優于RVM算法.特別是當單幅圖像平均假陽性簇個數為0.5時,ARVM算法的真陽性率為87%,而RVM算法的真陽性率只有71%.
由于FROC曲線受微鈣化點簇的判別標準影響,因此,在本文實驗中,改變微鈣化點簇的判別標準,即改變近鄰距離Dnn,然后統計相應的FROC曲線值來觀察算法的性能.圖12和圖13所示分別為Dnn=0.3 cm和Dnn=0.2 cm時的FROC曲線圖.綜合圖11,12和13可以看出,當改變近鄰距離,Dnn的取值從0.2 cm增大到0.4 cm時,ARVM和RVM算法對微鈣化點簇的處理性能都在不斷提高;但在相同的近鄰距離情況下,當單幅圖像平均假陽性簇個數小于1時,本文算法的處理性能都要優于RVM算法,即在相同的假陽性簇個數情況下,ARVM算法的真陽性率要高于RVM算法.
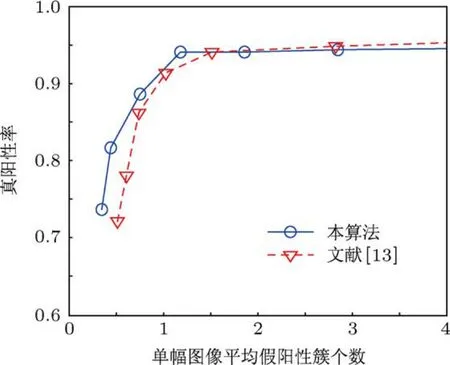
圖12 D nn=0.3 cm時的FROC曲線
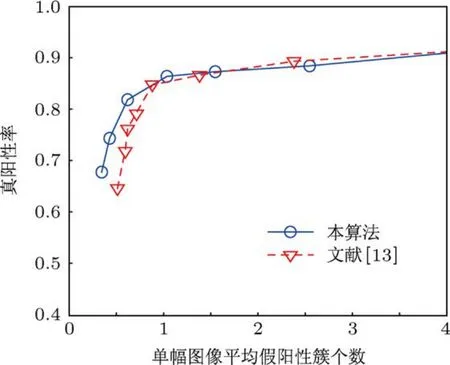
圖13 D nn=0.2 cm時的FROC曲線
由于組織結構特性的差異,女性乳腺通常分為致密型乳腺和脂肪型乳腺.乳腺腺體類型的不同,導致算法對其X線圖像中的微鈣化點簇的處理難度也不同.通常,致密型乳腺X線圖像比脂肪型乳腺X線圖像背景更復雜,對比度更低,處理也更難.實驗為降低測試圖像集中不同類型病例分布對算法性能的影響,采用自助重采樣(bootstrapping)方法[19,20]進行2000次測試檢驗算法的魯棒性,具體執行步驟為:
1)從測試圖像集(70幅圖像)中隨機提取30幅圖像作為該次測試的測試圖像子集;
2)對30幅圖像子集采用訓練好的分類器進行測試,并畫出該次測試結果的FROC曲線;
3)重復步驟1)和2),進行2000次測試;
4)畫出分類器測試結果的平均FROC曲線.
圖14所示為本算法對2000個通過自助重采樣獲得的測試圖像子集處理結果的FROC曲線圖.其中,每一條藍色曲線表示算法對1個測試圖像子集處理獲得的FROC曲線,共2000條;中間曲線表示2000次測試結果的平均FROC曲線,曲線上每個操作點的數據條表示在當前平均每幅圖像檢測到的假陽性微鈣化點簇的情況下,算法對不同組合測試圖像子集處理獲得的微鈣化點簇真陽性率的波動情況,數據條長度表示真陽性率的標準方差大小.數據條越短,波動性越小,魯棒性越強.
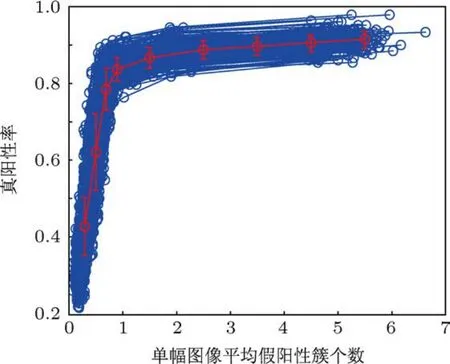
圖14 本算法2000次自助重采樣測試的FROC曲線圖
實驗對本文ARVM算法和文獻[13]的RVM算法在不同近鄰距離下進行自助重采樣測試,并對獲得的平均FROC曲線進行比較.圖15,16和17所示分別為Dnn=0.2,0.3和0.4 cm時2000次自助重采樣測試獲得的平均FROC曲線圖.
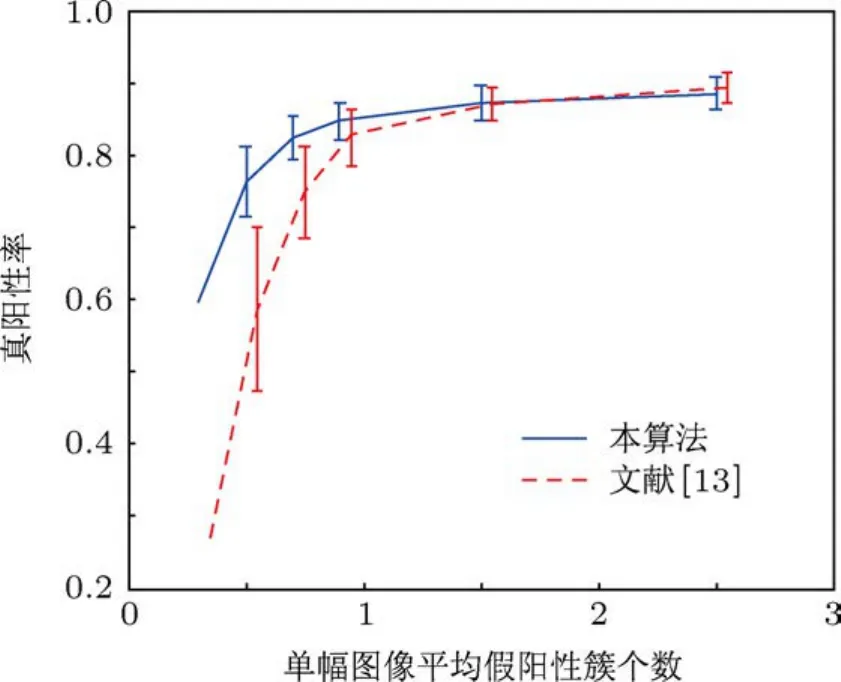
圖15 D nn=0.2 cm時2000次自助重采樣測試結果
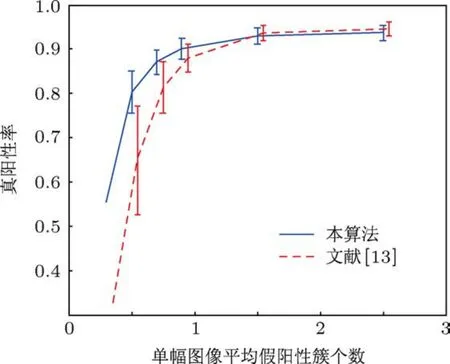
圖16 D nn=0.3 cm時2000次自助重采樣測試結果
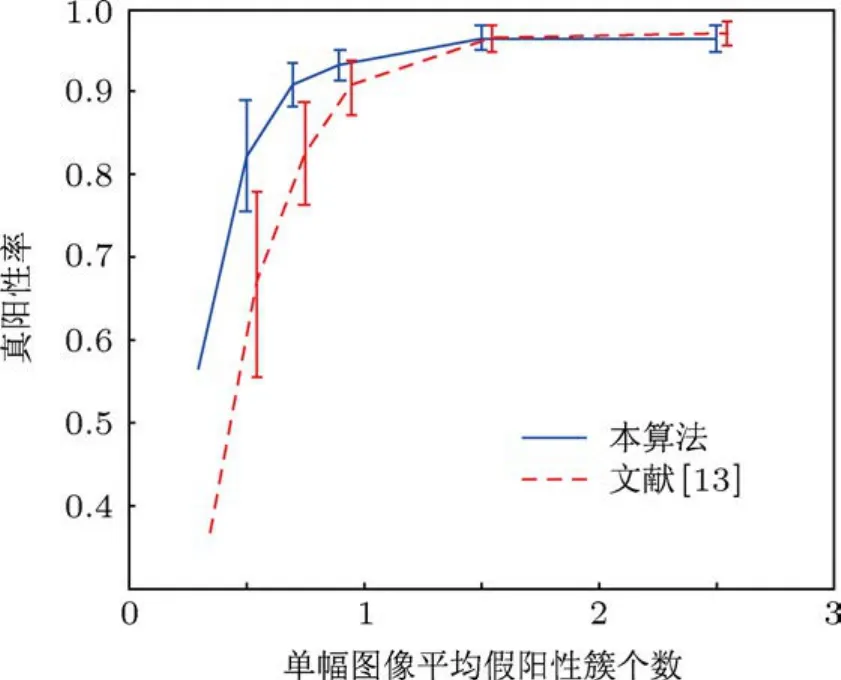
圖17 D nn=0.4 cm時2000次自助重采樣測試結果
從圖15,16和17可以看出,不論在哪種近鄰距離情況下,當單幅圖像平均假陽性簇個數小于1.5時,ARVM算法獲得的真陽性率總要高于RVM算法;同時從數據條的長度可以看出,對于不同組合類型的測試圖像集,ARVM算法真陽性率的波動性要比RVM算法小,具有更強的魯棒性.
5.3 算法運行效率和稀疏性比較
圖像分塊大小J的取值一方面與測試圖像的大小有關,另一方面與實驗機器配置有關,并不是J取值越大,算法效率越高.本實驗在雙核、主頻為1.60 GHz,內存為1 G的臺式計算機Matlab R2010a平臺上進行,測試圖像區域大小為800×800像素.圖18所示為J不同取值情況下ARVM檢測完一幅圖像的時間.由圖18可見,J取值為200時能獲得最快的檢測速度.
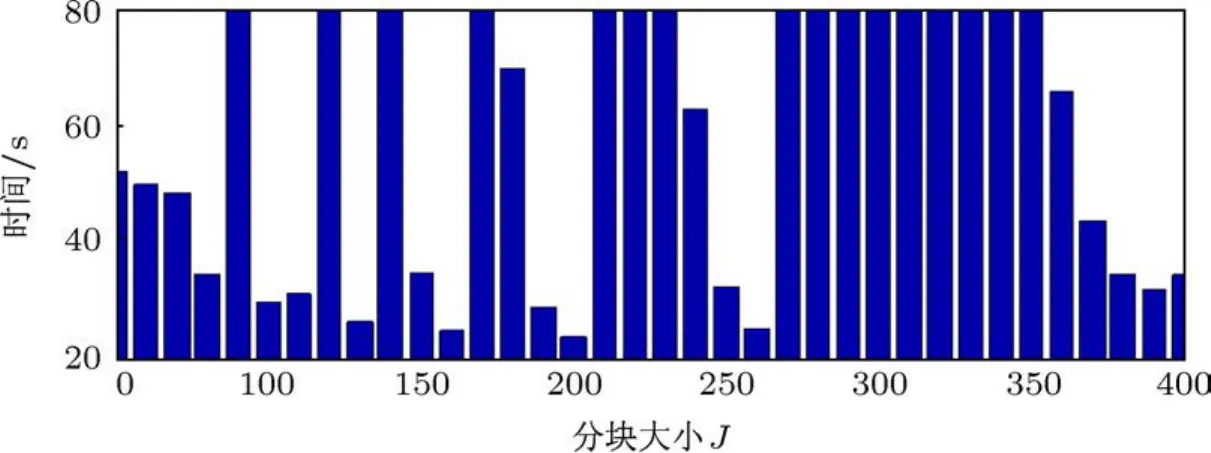
圖18 J不同取值情況下ARVM測試時間統計圖

表2 算法執行時間和相關向量數
為比較算法的計算復雜度和運行效率,本算法ARVM和文獻[13]中RVM的運行時間和模型相關向量個數如表2所示.其中,訓練時間指在訓練階段交叉驗證方法中平均每次的訓練時間,測試時間指在測試階段訓練好的分類器對每幅乳腺X線圖像的平均測試時間.從表2可以看出,相關向量個數由RVM的72個降到了ARVM的5個,相關向量個數的減少意味著獲得的分類器越稀疏,從而使得模型測試時的計算復雜度越低,計算速度越快.這可從分類器的測試時間體現出來,當對整幅圖像逐個像素點遍歷檢測時,RVM的測試時間為129.8 s,而ARVM的測試時間為80.4 s;當采用圖像分塊方法檢測時,RVM的測試時間為62.5 s,而ARVM的測試時間縮短為24.1 s.此外,從表2還可以看出,ARVM的訓練時間長于RVM,這是因為ARVM算法自動優化模型參數時要花費更多的時間.由于模型通常在用于測試前提前訓練好,因此訓練時間對于模型的臨床應用影響不大.
6 結論
利用自適應核學習相關向量機具有模型核參數自動優化設置、核基函數類型可以任意組合以及模型更稀疏的特性,本文將自適應核學習相關向量機應用于微鈣化點簇處理,研究了基于自適應核學習相關向量機的乳腺X線圖像微鈣化點簇處理方法,同時為提高模型的運算速度,實現了一種基于圖像分塊的自適應核學習相關向量機的微鈣化點簇快速處理方法.通過實驗仿真和算法性能分析,結果表明基于自適應核學習相關向量機的微鈣化點簇處理方法在較低的單幅圖像平均假陽性簇個數時能獲得比相關向量機方法更好的處理性能,模型更稀疏,同時實現的快速方法能進一步降低微鈣化點簇的處理時間,有利于臨床應用.
[1]Ahmed M H,Magda E 2011 IEEE Reviws in Biomedical Engineering 4 103
[2]Zhang X S,Gao X B,Wang Y,Zhang SJ2010 J.Infrared Millim Waves 29 27(in Chinese)[張新生,高新波,王穎,張士杰2010紅外與毫米波學報29 27]
[3]Liu G D,Zhang Y R 2011 Acta Phys.Sin.60 074303(in Chinese)[劉廣東,張業榮2011物理學報60 074303]
[4]Xiang L Z,Xing D,Guo H,Yang SH 2009 Acta Phys.Sin.58 4610(in Chinese)[向良忠,邢達,郭華,楊思華2009物理學報58 4610]
[5]Zhang H 2004 Acta Phys.Sin.53 2515(in Chinese)[張航2004物理學報53 2515]
[6]Xu X H,Li H 2008 Acta Phys.Sin.57 4623(in Chinese)[徐曉輝,李暉2008物理學報57 4623]
[7]Xiao X,Xu L,Liu B Y 2013 Acta Phys.Sin.62 044105(in Chinese)[肖夏,徐立,劉冰雨2013物理學報62 044105]
[8]Che L L,Zhang G Y,Song L,Cao W F 2011 Chin.J.Med.Phys.28 2467(in Chinese)[車琳琳,張光玉,宋莉,曹衛芳2011中國醫學物理學雜志28 2467]
[9]Tang J,Rangayyan RM,Xu J,Naqa IEl,Yang Y Y 2009 IEEETrans.Inform.Technol.Biomed.13 236
[10]Jing H,Yang Y Y,Nishikawa RM 2011 Phys.Med.Biol.56 1
[11]Jiang J,Yao B,Wason A M 2007 Comput.Med.Imag.Graph.31 49
[12]Naqa IE,Yang Y Y,Wernick M N,Galatsanos N P,Nishikawa R M 2002 IEEETrans.Med.Imag.21 1552
[13]Wei L,Yang Y Y,Nishikawa R M,Wernick M N,Edwards A 2005 IEEETrans.Med.Imag.24 1278
[14]Tzikas D G,Likas A C,Galatsanos N P 2009 IEEE Trans.Neural Networks20 926
[15]Tipping M,Faul A 2003 Proceedingsof the Ninth International Workshop on Artificial Intelligence and Statistics Key West,USA,January 3—6,2003 p1
[16]Kallergi M,Carney GM,Gaviria J1999 Med.Phys.26 267
[17]Muller K R,Mika S,Ratsch G,Tsuda K,Scholkopf B 2001 IEEE Trans.Neural Networks12 181
[18]Bunch PC,Hamilton JF,Sanderson GK,Simmons A H 1978 J.Appl.Photogr.Eng.4 166
[19]Samuelson FW,Petrick N 2006 Proceedingsof 3rd IEEEInternational Symposium On Biodedical Imaging Arlington,USA,April 4—6,2006 p1312
[20]Xing HY,Qi ZD,Xu W 2012 Acta Phys.Sin.61 240504(in Chinese)[行鴻彥,祁崢東,徐偉2012物理學報61 240504]

