MOLMAP指數及其在變異性預測中的應用
張慶友 龍海林 馮秀林 索凈潔 張丹丹 李靜亞
許力壯2 許 祿3,*
(1河南大學化學化工學院環境與分析科學研究所,河南開封475004;
2深圳市人民醫院,廣東深圳518020;3中國科學院長春應用化學研究所,長春130022)
MOLMAP指數及其在變異性預測中的應用
張慶友1龍海林1馮秀林1索凈潔1張丹丹1李靜亞1
許力壯2許 祿3,*
(1河南大學化學化工學院環境與分析科學研究所,河南開封475004;
2深圳市人民醫院,廣東深圳518020;3中國科學院長春應用化學研究所,長春130022)
分子映射(MOLMAP)指數是以分子的化學鍵描述符為基礎,通過Kohonen自組織映射依據一定的算法而衍生.化學鍵描述符是由化學鍵的物理化學性質,如兩端原子的電荷差和拓撲性質,鍵連雜原子數量等所組成.本文將分子映射指數應用于4075個有機物質(Ames實驗結果:2305個結構有誘變性,1770個結構無誘變性)的變異性預測.通過隨機森林,分別采用三種類型的指數建立模型:(1)采用不同維數的分子映射指數;(2)采用全局分子描述符;(3)將分子映射指數與全局分子描述符相結合.整個數據集的集外(out-of-bag)交叉驗證的正確預測率達到85.4%.為了檢驗模型的穩定性,采用所建模型預測源于另一數據庫的472個化合物,正確預測率為86.7%,與此前的研究相比,兩個預測結果均有所提高.
分子映射指數;Kohonen自組織映射;隨機森林;誘變性;結構-活性關系
1 引言
MOLMAP指數在化合物反應性預測和化學反應的分類預測均有應用.14-18本文作者和Airesde-Sousa19合作,采用Gasteiger等20-22提出的7個經驗的化學鍵物理化學性質,例如共振穩定性和化學鍵裂解能等,依據一定的算法生成MOLMAP指數,并實現了有機物質的變異性預測.本文在此基礎上進一步探索化學鍵性質的描述,采用27種物理化學性質及41種拓撲性質描述化學鍵,然后生成分子MOLMAP指數,并通過隨機森林應用于一個較大型的數據集(4075個物質),預測物質的變異性.此外還采用該模型預測了源于另一數據庫的數據集,檢驗所建模型的穩定性.
2 數據集
數據集由4083個有機物質所組成,10由于所用軟件ChemAxon23的Marvin不能計算其中的8個分子,因而采用余下4075個有機物質作為研究對象,相應的Ames實驗結果為:2305個結構是誘變性物質,1770個結構是非誘變性物質.測試集源于另外一個數據庫,是由Young等24收集的472個化合物,其中305個結構為誘變性物質,167個結構是非誘變性物質.
3 方法
3.1 化學鍵描述符
本文計算了68種化學鍵性質,其中包括化學鍵的電荷差、化學鍵的軌道電負性差、鄰接原子的最大及最小極化率、鍵端原子的電荷密度等27種物理化學性質和化學鍵的類型、鍵端原子的類型、鍵連雜原子的數目等41種拓撲性質,23由每個化學鍵衍生一個68維的矢量.
眾所周知,化學反應的基本特征就是化學鍵的生成和斷裂,而物質的變異性與化學反應密切相關,故本文通過Kohonen自組織映射采用化學鍵的性質衍生MOLMAP指數.
3.2 Kohonen自組織映射
Kohonen自組織映射(SOM)是由Kohonen25在1982年首先提出,它是一種無管理的人工神經網絡,SOM是由二維神經元所組成,每一個神經元代表一個向量,其長度與輸入向量的維數(本文為化學鍵描述符)相同,26,27見圖1.在應用過程中,首先采用訓練集的化學鍵描述符對SOM進行訓練,然后,遞交測試集的化學鍵描述符到被訓練的SOM進行預測,其過程簡述如下,
該工廠于3月份投產,目前生產板材厚度為2~35 mm。其日產量為650 m3,由此Action Tesa公司成為印度最大的MDF生產商,年產能 51.1 萬 m3。
SOM訓練時,首先進行隨機數的初始化,其次,提交化學鍵描述符矢量至SOM,并計算其與各神經元的歐式距離,找到與該描述符最接近的神經元,稱為贏元,然后,調整贏元和鄰近神經元的權值,使其與輸入向量更相似,調整幅度隨著與贏元的距離增大而減小.多次遞交訓練集中所有的描述符矢量,重復上述操作達到預先指定的次數時停止.經過上述訓練,相似的神經元將處于鄰近的位置.若遞交未知的化學鍵描述符到被訓練的SOM,則所得贏元代表該化學鍵.SOM的特點為相近的輸入向量將落入相同的或相近的神經元,由此描述符相似的化學鍵將落入相同或者鄰近的位置.
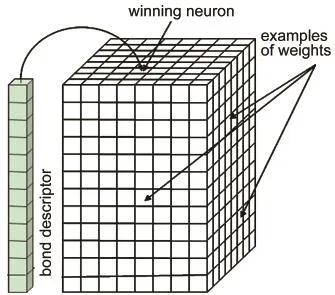
圖1 Kohonen自組織映射(SOM)模型Fig.1 Kohonen self-organization mapping(SOM)model
3.3 分子MOLMAP指數生成
分子MOLMAP指數是通過Kohonen提出的自組織映射,由分子中所有化學鍵描述符所衍生. MOLMAP指數是一個基于化學鍵類型的分子指數,相同類型的化學鍵是指物理化學性質或拓撲性質相似的化學鍵.分子MOLMAP指數的生成過程簡述如下:28(1)計算數據集中所有化合物的化學鍵描述符;(2)從中隨機選取一定數量的化學鍵作為訓練集,本文為4999個化學鍵及其描述符;(3)以此4999個化學鍵訓練SOM,得到被訓練的人工神經網絡;(4)從數據集中提取所有的化學鍵描述符,并遞交到已經被訓練的SOM,其贏元代表相應的化學鍵.
如遞交一個分子的所有化學鍵描述符到上述被訓練的SOM,然后依據這一分子的所有化學鍵在SOM上的分布,進一步可將SOM上的輸出轉換為數值形式.即將每個化學鍵的贏元(與該化學鍵描述符最相似神經元)賦值為1.0,則考慮到鄰近神經元的相似性,那么贏元周圍的8個鄰接神經元譬如可賦值為0.3,由此可轉化為數字的形式.另外,如果分子中幾個化學鍵落入同一個神經元,則所得到的數值相加.
圖2為一個通過Kohonen SOM生成144(12× 12)維的MOLMAP指數示例.圖2a所示為一個有機分子的所有化學鍵經訓練后在SOM中的映射,其中有4個等價的Cl―C鍵落入第1行第9列.若按照前述規則轉化為數值形式,則見圖2b,其中第1行第9列包含4個Cl―C鍵,因而其數值為4.0=4×1.0.最后以從左向右的順序一列接一列的裁剪,并依次把列列首尾相接就得到分子MOLMAP指數,本例為144 (12×12)維.可見,分子MOLMAP指數的維數僅僅與二維神經元的維數有關,而與分子中化學鍵的數量無關.該指數的每一個變量對應著SOM的一個神經元,每一個神經元可以看成一種新化學鍵類型.本文采用前述68維的化學鍵描述符,故同一類型的化學鍵是指具有相似物理化學和拓撲性質因而落入同一神經元的化學鍵.
為了檢驗MOLMAP指數的維數對模型預測能力的影響,本文分別采用了維數為625(25×25)、900 (30×30)和1296(36×36)的SOM生成MOLMAP指數.
3.4 全局分子描述符
除前述MOLMAP指數外,本文還計算出35種分子的物理化學性質和拓撲性質,23構成35維的全局分子描述符矢量(global molecular descriptor).其中包括原子數目、化學鍵的數目、分子質量、芳香原子數目、極化率、NH鍵的數目、NH2鍵的數目、氧原子的數目、氮原子的數目、最小原子電荷、最大原子電荷、氫原子的最小電荷、氫原子的最大電荷、重原子數目、羥基數目、氫鍵受體個數、氫鍵給體個數、logP、直鏈數目、脂肪環數目、芳香環數目、最小環的大小、雜環數目、雜芳香環數目、可旋轉鍵數目、分子表面積、分子極性面積、分子最大投影面積、分子最小投影面積、折射率、平均分子極化度、芳香鍵數目、共振結構的數目、芳香稠環的數目和脂肪稠環的數目.其中一些性質已經廣泛應用于構效關系研究中.29,30
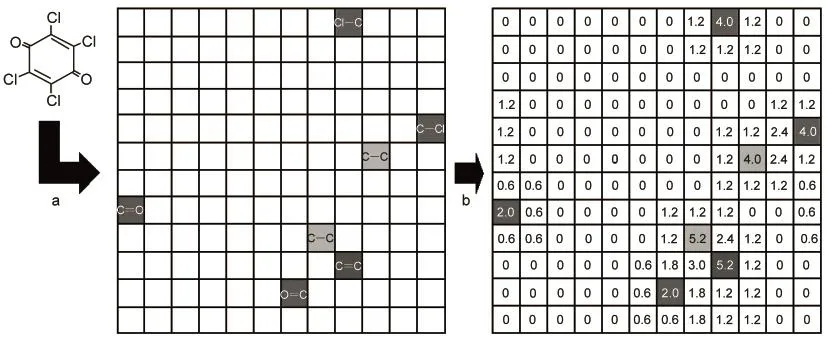
圖2 分子MOLMAP指數的生成Fig.2 Generation of the MOLMAPdescriptor for a molecule
3.5 隨機森林
隨機森林(random forest)是Breiman在Bagging算法之后,提出的一種利用樹的集合進行分類預測和回歸預測的組合算法,31,32隨機森林程序來源于P-program的2.10.1版本,33該算法已經在實踐中得到成功的應用.34
隨機森林通過隨機的方式生成大量的樹來建立數學模型,35每一棵樹類似于一個分類回歸樹,但不進行修剪.對于每一棵樹,在采用訓練集進行訓練時首先隨機選取一個變量子集,然后從選取的子集中選擇變量來劃分結點,對輸入矢量的每一個變量找到一個最佳的分割點(能夠最有效分類的值),其中分類效果最好的變量作為從父結點到子結點的判據,然后根據該結點內的多數票來決定該結點屬于哪一類.每棵樹均獨立的進行訓練,并用于未知樣本的預測.隨機森林中樹的數量由人為設定(本文為1000棵樹),每一棵樹的預測相當于一票,最終的結果由所有樹的多數票決定.如某化合物在900棵樹中被預測為變異性物質,100棵樹中被預測為非變異性物質,則該化合物被預測為變異性物質.隨機森林提供了變量重要性的量化評價,評價主要依據兩種方式:(1)隨機地交換某變量的值,由所引起的誤分類變化評價該變量;(2)采用某變量進行結點分類,由所產生子節點的分類效果來量化該變量的重要性.變量重要性可以作為變量選擇的依據,同時變量重要性結果有助于發現與活性密切相關的化合物結構特征,從而提供對活性研究有啟示意義的信息.
在訓練隨機森林過程中,每一棵樹均把訓練集隨機分成兩部分,一部分數據作為訓練集,另一部分則作為測試集,并綜合每棵樹所建模型對測試集的預測結果來評價整個訓練集,稱為集外(OOB)交叉驗證.
4 結果與討論
采用訓練集的4075個有機物質訓練隨機森林,并分三種情況建立物質變異性的預測模型:(1)僅采用全局分子描述符;(2)僅采用MOLMAP指數,其中MOLMAP指數的維數為:625(25×25)、900(30× 30)或1296(36×36);(3)MOLMAP指數和全局分子描述符相結合.此外,前述每一個預測模型均應用于獨立測試集的472個化合物.
首先僅采用全局分子描述符建立預測模型,所得結果見表1第2行.其中訓練集的OOB交叉驗證結果為81.7%;獨立測試集的預測結果為77.6%.
本文的MOLMAP指數由兩類化學鍵描述符,即物理化學性質及拓撲性質所衍生.故分別采用化學鍵描述符中的27個物理化學性質描述符和41個拓撲性質描述符建立物質變異性預測模型,則訓練集所有化合物的OOB交叉驗證結果及測試集預測結果見表1與表2,其中表1所示為由化學鍵的物理化學性質衍生MOLMAP指數的預測結果;表2所示為由化學鍵的拓撲性質衍生MOLMAP指數的預測結果.
由于Kohonen SOM采用隨機數進行初始化,為了得到較穩定的預測模型,采用化學鍵描述符訓練SOM三次,每次均得到一個MOLMAP指數,并分別采用三個MOLMAP指數進行預測.對于一個化合物來說,若其中兩個MOLMAP指數的預測結果為變異性,而一個預測為非變異性,則預測該化合物為變異性.即以三個模型的多數票作為最終預測結果.
由表1可知:若僅采用化學鍵的物理化學性質衍生的MOLMAP指數建立預測模型,則訓練集的OOB交叉驗證結果為84.3%-84.7%,獨立測試集的預測結果為84.8%-85.0%,均顯著好于僅采用全局分子描述符的結果.若進一步將MOLMAP指數與全局分子描述符相結合,則訓練集的交叉驗證結果在84.8%-84.9%之間;測試集的預測結果在84.3%-85.0%之間.

表1 基于35維全局分子描述符和由化學鍵物理化學性質衍生的MOLMAP指數的隨機森林預測結果Table 1 Correct predictions of random forest based on 35 global molecular descriptors and MOLMAPdescriptors derived from physiochemical properties of chemical bonds
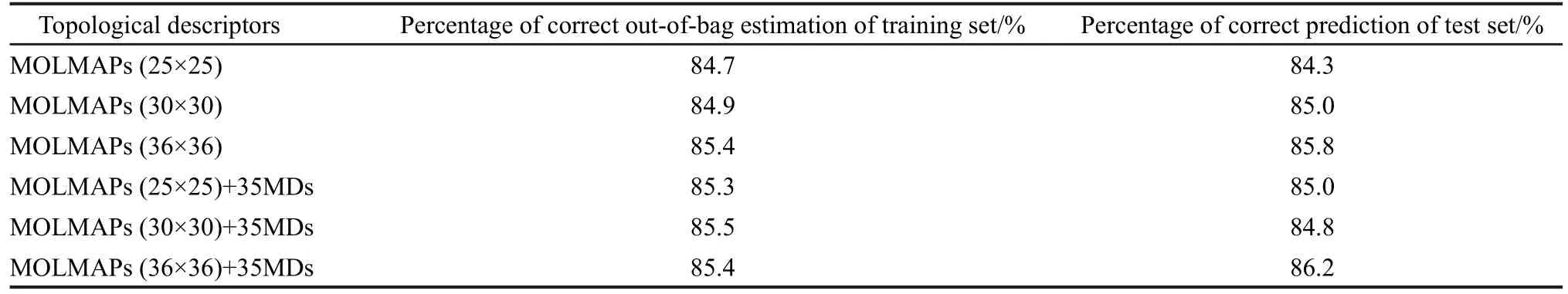
表2 基于35維全局分子描述符和由化學鍵的拓撲性質衍生的MOLMAP指數的隨機森林預測結果Table 2 Correct predictions of random forest based on 35 global molecular descriptors and MOLMAPdescriptors derived from topological properties of chemical bonds

表3 基于35維全局分子描述符和由化學鍵的物理化學性質與拓撲性質衍生的MOLMAP指數的隨機森林預測結果Table 3 Correct predictions of random forest based on 35 global molecular descriptors and MOLMAPdescriptors derived from physiochemical properties and topological properties of chemical bonds
由表2可知:若采用化學鍵的拓撲性質衍生MOLMAP指數與全局分子描述符相結合,則訓練集的交叉驗證結果在85.3%-85.5%之間;測試集的預測結果在84.8%-86.2%之間,所得結果略優于表1的結果.
在此基礎上,合并27維的物理化學描述符和41維的拓撲性質描述符衍生68維的化學鍵描述符,由化學鍵描述符生成MOLMAP指數,并采用前述方法通過隨機森林構建預測模型和進行預測,則所得結果見表3.由表3可知:若MOLMAP指數與35個全局分子描述符相結合,則OOB的交叉驗證結果為85.3%-85.6%,測試集的預測結果為85.6%-86.7%.在此前研究19中同一訓練集的OOB交叉驗證最佳結果為84.1%,測試集為84.5%.可見,本文結果好于該結果.
MOLMAP指數的每一個變量均衍生于Kohonen自組織映射的神經元,而每一個神經元均代表著“化學鍵類型”.根據隨機森林的變量重要性, MOLMAP指數中最重要的兩個變量和第四重要的變量均是由N=O雙鍵所衍生,這些化學鍵主要是來源于硝基,也有少部分來源于C―N=O和O=N―N基團.第三重要的變量由苯并化合物上苯環的碳碳鍵所衍生.這說明該類基團是影響變異性的重要基團.在文獻10中指出,含有上述兩個基團的化合物通常具有變異性,這表明上述模型不僅僅能夠建立較好的模型,還有助于發掘引起變異性的重要化學鍵類型.
綜上可見,采用化學鍵的物理化學描述符衍生MOLMAP指數所建立的模型預測結果最差;若采用化學鍵的拓撲性質則預測能力有所提高;而將二者相結合則預測能力最佳.
5 結論
對于由4075個化合物所組成的比較大型的數據集,本文基于Kohonen自組織映射,采用所提取的68種化學鍵的物理化學性質和拓撲性質,在此基礎上生成625(25×25)、900(30×30)、1296(36×36)三種維數的MOLMAP指數,并結合35維全局分子描述符,通過隨機森林建立變異性預測模型,從而實現了有機物質變異性的自動預測.通過源于另一個數據庫的獨立數據集進行檢驗,模型穩健性較好,比此前的預測能力有所提高,說明通過化學鍵描述符的改進,能夠建立更加穩定的變異性物質預測模型.
(1) Patlewicz,G.;Rodford,R.;Walker,J.D.Environ.Toxicol. Chem.2003,22,1885.
(2) Benigni,R.Chem.Rev.2005,105,1767.
(3) Hansen,K.;Mika,S.;Schroeter,T.;Sutter,A.;Laak,A.T.; Steger-Hartmann,T.;Heinrich,N.Muller,K.R.J.Chem.Inf. Model.2009,49,2077.
(4) Casalegno,M.;Benfenati,E.;Sello,G.J.Chem.Inf.Model. 2011,51,1564.
(5)Ames,B.N.;McCann,J.;Yamasaki,E.Mutation Res.1975,3, 347.
(6) Mortelmans,K.;Zeiger,E.Mutation Res.2000,455,29.
(7) Meier,J.R.Mutation Res.1988,196,211.
(8) Zheng,M.Y.;Liu,Z.G.;Xue,C.X.Zhu,W.L.;Chen,K.X.; Luo,X.M.;Jiang,H.L.Bioinformatics 2006,22,2099.
(9) Liao,Q.;Yao,J.H.;Yuan,S.G.Molecular Diversity 2007,11, 59.
(10) Kazius,J.;McGuire,R.;Bursi,R.J.Med.Chem.2005,48,312.
(11) Helma,C.;Cramer,T.;Kramer,S.;Raedt,L.D.J.Chem.Inf. Comput.Sci.2004,44,1402.
(12) Popelier,P.L.A.;Smith,P.J.;Chaudry,U.A.J.Comput.-Aided Mol.Des.2004,18,709.
(13) He,L.N.;Jurs,P.C.;Custer,L.L.;Durham,S.K.;Pearl,G.M. Chem.Res.Toxicol.2003,16,1567.
(14) Gupta,S.;Matthew,S.;Abreu,P.M.;Aires-de-Sousa,J.Bioorg. Med.Chem.2006,14,1199.
(15) Zhang,Q.Y.;Aires-de-Sousa,J.J.Chem.Inf.Model.2005,45, 1775.
(16) Latino,D.A.R.S.;Aires-de-Sousa,J.Angew.Chem.Int.Edit. 2006,45,2066.
(17) Latino,D.A.R.S.;Zhang,Q.Y.;Aires-De-Sousa,J. Bioinformatics 2008,24,2236.
(18) Latino,D.A.R.S.;Aires-de-Sousa,J.J.Chem.Inf.Model. 2009,49,1839.
(19) Zhang,Q.Y.;Aires-de-Sousa,J.J.Chem.Inf.Model.2007,47, 1.
(20) Gasteiger,J.;Marsili,M.;Hutchings,M.G.;Saller,H.;L?w,P.; R?se,P.;Rafeiner,K.J.Chem.Inf.Comput.Sci.1990,30,467.
(21) Simon,V.;Gasteiger,J.;Zupan,J.J.Am.Chem.Soc.1993,115, 9148.
(22) Gasteiger,J.Mini-Rev.Med.Chem.2003,3,789.
(23) http://www.chemaxon.com/.
(24) Young,S.S.;Gombar,V.K.;Emptage,M.R.;Cariello,N.F.; Lambert,C.Chem.Int.Lab.Syst.2002,60,5.
(25) Kohonen,T.Biol.Cybern.1982,43,59.
(26) Aires-de-Sousa,J.Chem.Int.Lab.Syst.2002,61,167.
(27) http://www.dq.fct.unl.pt/staf/jas/jatoon/.
(28) Long,H.L.;Feng,X.L.;Suo,J.J.;Zhang,D.D.;Li,J.Y.; Zhang,Q.Y.;Xu,L.Computers and Applied Chemistry accepted.[龍海林,馮秀林,索凈潔,張丹丹,李靜亞,張慶友,許 祿.計算機與應用化學,已接受.]
(29) Dai,Z.J.;Zhou,W.;Yuan,Z.M.Acta Phys.-Chim.Sin.2011, 27,1654.[代志軍,周 瑋,袁哲明.物理化學學報,2011,27, 1654.]
(30) Yang,G.B.;Li,Z.R.;Rao,H.B.;Li,X.Y.;Chen,Y.Z.Acta Phys.-Chim.Sin.2010,26,3351.[楊國兵,李澤榮,饒含兵,李象遠,陳宇綜.物理化學學報,2010,26,3351.]
(31) Breiman,L.Machine Learning 1996,24,123.
(32) Díaz-Uriarte,R.;Andrés,S.A.D.BMC Bioinformatics 2006,7, 3.
(33) http://www.R-project.org.
(34) Svetnik,V.;Liaw,A.;Tong,C.;Culberson,J.C.;Sheridan,R. P.;Feuston,B.P.J.Chem.Inf.Comput.Sci.2003,43,1947.
(35) Breiman,L.Machine Learning 2001,45,5.
October 27,2011;Revised:December 19,2011;Published on Web:December 28,2011.
MOLMAP Descriptor and Its Application to Mutagenicity Prediction
ZHANG Qing-You1LONG Hai-Lin1FENG Xiu-Lin1SUO Jing-Jie1ZHANG Dan-Dan1LI Jing-Ya1XU Li-Zhuang2XU Lu3,*
(1Institute of Environmental and Analytical Sciences,College of Chemistry and Chemical Engineering,Henan University,Kaifeng 475004,Henan Province,P.R.China;2Renmin Hospital of Shenzhen,Shenzhen 518020,Guangdong Province,P.R.China;3Changchun Institute of Applied Chemistry,Chinese Academy of Sciences,Changchun 130022,P.R.China)
The molecular mapping of atom-level properties(MOLMAP)descriptor was generated on the basis of chemical bond descriptors of a molecule by Kohonen self-organizing map with a specific algorithm. The bond descriptors were composed of the physiochemical properties of the chemical bond,such as the difference of the charges between the two atoms and topological properties,such as the number of hetero-atoms connected to the two atoms.In this paper,the MOLMAP descriptors were used to predict the mutagenicity of 4075 organic substances(2305 mutagens and 1770 nonmutagens in Ames test).Random forests were used to construct mathematical models with three kinds of descriptors:(1)MOLMAP descriptors of different size;(2)global molecular descriptors;(3)the combination of MOLMAP descriptors and global molecular descriptors.The correct prediction percentage of out of bag(OOB)cross-validation of the whole data set reached 85.4%.To test the stability of the prediction model,it was used to predict the properties of a test set that was composed of 472 compounds collected from another database.The percentage of correct prediction of the test set was 86.7%.The prediction results were improved compared with the results of previous work.
MOLMAP descriptor;Kohonen self-organizing map;Random forest;Mutagenicity; Structure-activity relationship
10.3866/PKU.WHXB201112281
O641
?Corresponding author.Email:luxu@ciac.jl.cn;Tel:+86-431-85262239.
The project was supported by the National Natural Science Foundation of China(20875022),Scientific Research Foundation for the Returned
Overseas Chinese Scholars,Ministry of Education of China(2009(1001)),and International Science and Technology Cooperation of Henan Province, China(114300510009).
國家自然科學基金(20875022),教育部留學回國人員科研啟動基金(2009(1001))及河南省國際科技合作項目(114300510009)資助

