狼尾草屬牧草RAPD反應體系的建立與優化
喬良普 (山東省臨沂市河東區畜牧局 276034) 李麗 (臨沂市科學技術合作與應用研究院)
?
狼尾草屬牧草RAPD反應體系的建立與優化
喬良普 (山東省臨沂市河東區畜牧局 276034) 李麗 (臨沂市科學技術合作與應用研究院)
利用RAPD技術對狼尾草屬牧草進行種質資源遺傳多樣性研究中,對影響PCR擴增效果的一些因素諸如DNA的提取濃度、Taq酶的選擇及其用量、Mg2+濃度、dNTPs的用量以及退火溫度等指標進行反復篩選和優化,結合正交試驗的結果,最終確定RAPD-PCR的反應體系為:總體積20μl,反應體系中含有1.5μl (25mM)Mg2+、0.4μl(10mM)dNTPs、1.2μl(10pM)primer、0.4μl(5U/μl)Taq DNA Polymerase、2.0μl10×PCR Buffer、2.0μl DNA模板,ddH2O補足到20μl。
狼尾草屬 RAPD 反應體系 優化
狼尾草屬(Pennisetum Rich)牧草為一年生或多年生禾本科牧草,主要分布在熱帶和亞熱帶,全世界約有80種,多數原產于非洲[1]。目前我國人工栽培利用的種主要有多年生的象草(P.purpureum Schumach)和一年生的美洲狼尾草(P.americanum Rich.)及其種間雜交種。截止到2008年,經全國草品種審定委員會審定的狼尾草屬品種共8種,其中引進福建省栽培種植面積較大的有5個品種依次為華南象草(P.purpureum cv. Huanan)、雜交狼尾(P.am-ericanum×P.purpureum)、熱研4號王草(P.purpureum× P.americanurn.cv.ReyanNo.4)、桂牧1號雜交象草[(P.americanum×P.purpureum)×P.durpureum cv.Guimu No.1]和摩特矮像草(P.purpureum cv.Mott)。因其廣泛的適應性、高產草量、高蛋白質含量及無融合生殖等特性,在發展草食動物生產中發揮了越來越重要的作用[2]。
RAPD(Random Amplified Polymorphic DNA,隨機擴增多態性DNA)技術是1990年由Welsh and Mcclelland[3]和Williams[4]同時提出的一種DNA檢測技術,是繼RFLP技術之后發展起來的一種以PCR為基礎的分子生物學技術。RAPD標記具有以下優點:所需基因組DNA用量少,而靈敏度高;實驗易于操作,花費少;不必清楚與物種有關的基因組序列以及相關的分子生物學信息;具有通用性。目前,RAPD分子標記已經應用到了棉屬[5]、石蒜屬[6]、杜鵑屬[7]、松毛蟲屬[8]、卷柏屬[9]等植物。本文試圖摸索適合狼尾草屬牧草的RAPD反應體系,為以后其遺傳多樣性的研究提供理論依據及幫助。
1 材料與方法
1.1 材料
材料選自福州狼尾草屬牧草6個品系(粗莖象草、矮象草、細莖象草、紫象草、王草和臺灣二系狼尾草)共12份材料,根據狼尾草屬牧草的分布狀況選取具有代表性的樣本。要求每個樣本的株間距至少在50m以上,每株采摘的鮮嫩葉片都做好標記,塑料袋低溫保存,立即拿到實驗室,進行基因組總DNA的提取。
1.2 主要試驗設備與試劑
主要試驗儀器設備有:PCR儀(美國Bio-RAD公司)、穩壓電泳儀(北京六一儀器廠)、水平電泳槽(北京六一儀器廠)、凝膠成像分析系統(美國Bio-Rad公司)、低溫離心機、紫外可見分光光度計、-80℃超低溫冰箱、移液器(德國Eppendorf)等。試驗所用的Taq DNA聚合酶、DNA Marker、引物等試劑均購自上海生工生物工程公司。
1.3 方法
1.3.1 基因組DNA的提取及檢測 采用植物基因組DNA提取試劑盒(天根生物有限公司)提取DNA,經0.8%瓊脂糖凝膠檢測及紫外分光光度計檢測純度和濃度,在凝膠成像分析系統上拍照,最后稀釋成10ng/μl,放在-20℃冰箱中備用。
1.3.2 RAPD初始擴增體系及PCR擴增程序 初始的擴增反應體系為:總體積20μl,反應體系中含有1.5μl(25mM) Mg2+、0.4μl(10mM)dNTPs、1.2μl(10pM)primer、0.4μl (5U/μl) Taq DNA Polymerase、2.0μl10×PCR Buffer、2.0μl DNA模板,ddH2O補足到20μl。
初步設定的擴增程序為:95℃預變性5min,接下來94℃變性45s,50~60℃退火45s,72℃延伸60s共40個循環,72℃延伸7min,最后4℃保存。
1.3.3 RAPD-PCR擴增反應體系的優化 為了確保試驗的穩定性和準確性,需要對RAPD反應體系進行摸索,本試驗采用正交試驗的方法,確定最終的反應體系。
就狼尾草屬RAPD反應中的Taq DNA Polymerase的濃度、Mg2+濃度、dNTP濃度和引物濃度,選用選用L16(44)正交表,設計的PCR各成分的因素水平(表1)及正交試驗設計表見表2。

表1 RAPD體系的因素水平
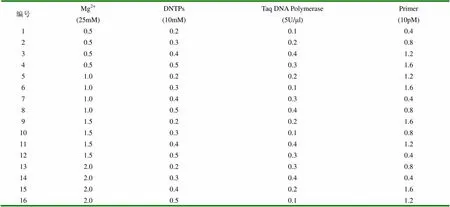
表2 PCR正交試驗設計
以上16個處理,每個處理2次重復,共32個PCR管,按表2加樣。反應體系總體積20μl,每管還包括1×PCR Buffer2.0μl和2μl的模板DNA。
1.3.4 PCR產物電泳檢測 PCR產物經含EB1.4%瓊脂糖凝膠電泳100V電壓大約1h后,在美國Bio-Rad公司生產的型號GelDocXR凝膠成像系統上拍照。
2 結果與分析
2.1 基因組DNA的提取
使用天根植物基因組DNA提取試劑盒提取的DNA,經瓊脂糖凝膠電泳后,拍照的圖如1,從圖可以看到提取的DNA整齊劃一、明亮,且沒有拖帶產生。說明DNA提取的質量較好,沒有DNA降解,也沒有RNA及蛋白質污染,完全可以滿足RAPD-PCR反應的需要。

圖1 為狼尾草屬牧草DNA 的凝膠電泳圖
注:泳道1~6DNA分別為粗莖象草、矮象草、細莖象草、紫象草、王草和臺灣二系狼尾草
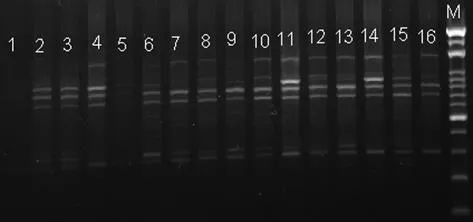
圖2 正交設計RAPD反應體系的擴增
注:1~16泳道為正交試驗的16個處理
2.2 RAPD反應體系的確定
按照表2正交試驗的設計,每管重復一次進行加樣、PCR,將所得的產物凝膠電泳,結果如圖2。從結果明顯看出,隨著dNTPs的增加,擴增產物明顯變清晰,但其特異性差(1~3號);而Taq DNA Polymerase濃度太低時,直接影響PCR擴增,甚至擴增很模糊,第11,14樣本其特異性高,產物穩定可靠。再結合單因素試驗結果,最終確定的20μl RAPD反應體系見表3:

表3 RAPD反應體系 (μl)
2.3 引物退火溫度的篩選
根據正交試驗的結果,選用第11號方案,對引物S228的退火溫度進行選擇,依據引物的Tm值,Tm±5℃生成10個梯度,如圖3可見退火溫度影響到PCR的特異性及產量,溫度高,產物的特異性高。10個梯度均能擴增出條帶,當溫度低時,有些條帶不明顯,背景比較模糊;當溫度在44℃時,引物與模板的特異性最高,且譜帶清晰、多態性較高,當溫度升高或降低時,條帶減少且模糊。因此引物S228最佳的退火溫度選擇是44℃。當某一引物擴增出來的條帶清晰明亮且多態性良好時保留,用做下一步試驗。
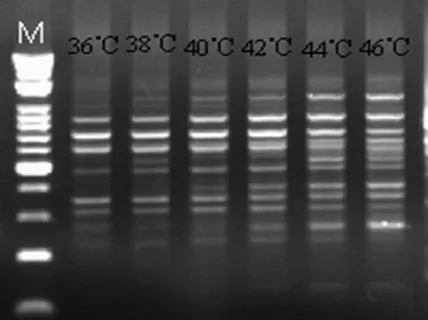
圖3 不同退火溫度對擴增結果的影響

圖4 12份狼尾草屬牧草的RAPD擴增結果
2.4 RAPD反應體系的驗證 經過優化最終確定的RAPD反應體系為:總體積20μl,反應體系中含1.5μl(25mM) Mg2+、0.4μl(10mM)dNTPs、1.2μl(10uM)primer、0.4μl Taq DNA Polymerase、2.0μl 10×PCR Buffer、2.0μl模板,ddH2O補足到20μl。反應程序:95℃預變性5min;4℃變性45s,36~45℃退火45s,72℃延伸60s,循環40次;72℃延伸7min;4℃保存。用引物S228對12份牧草進行擴增,結果如圖4。從圖上可以看到,擴增的條帶清楚,位點明確,多態性高。
3 小結與討論
3.1 DNA的提取是PCR反應的第一步 DNA提取的質量直接影響到后續反應的進行。本文用天根試劑盒提取的DNA純度高,A260nm/A280nm值在1.8左右,沒有RNA及蛋白質污染。DNA純度高,且擴增出來的條帶清晰明亮,多態率高,完全滿足RAPD反應的需要。
PCR反應中的五大要素分別為引物、DNA聚合酶、dNTPs、Mg2+以及模板,PCR產物的特異性主要取決于引物與模板的匹配程度,每個PCR反應應該以最低的引物量產生所需的結果為好。引物濃度過高會引起錯配和非特異性擴增,且增加引物之間形成二聚體的機會。DNA聚合酶濃度過高可引起非特異性擴增,過低合成產物量減少。4種dNTP濃度要相等,任何一種濃度不同,就會引起錯配。dNTPs濃度過低又會降低PCR產物的產量。Mg2+對PCR擴增的特異性和產量有顯著的影響,濃度過高,反應特異性降低,濃度過低會降低Taq DNA聚合酶的活性,使反應產物減少。模板的濃度和純度也是PCR成功與否的關鍵。如果PCR體系中的任何一個成分選擇不合適就會使PCR結果變差甚至失敗。PCR反應的程序也同樣重要,其中退火溫度、循環數、延伸時間、變性時間都會對PCR產物的產量和結果的特異性產生影響。
3.2 反應體系的建立 依據正交試驗的結果,經過反復篩選優化的RAPD-PCR反應體系為:總體積20μl,反應體系中含有1.5μl(25mM)Mg2+、0.4μl(10mM)dNTPs、1.2μl (10pM)primer、0.4μl(5U/μl)Taq DNA Polymerase、2.0μl 10×PCR Buffer、2.0μl DNA模板,ddH2O補足到20μl。
[1] 林潔榮, 劉建昌. 福建南亞熱帶狼尾草屬牧草品比試驗[J]. 福建農林大學學報(自然科學版), 2003,32(1): 110-112.
[2] 卓坤水, 蘇水金, 杜仲清等. 閩引象草(Pennisetum purpureum schum MIN-YIN)的選育[J]. 熱帶作物學報, 2009,30(8): 1196-1200.
[3] Welsh J,Mclelland M.Fingerprinting genomes using PCR with arbitrary primers[J].Nucleic Acids Research, 1990,(18): 7213-7218.
[4] Williams J.G.K,Kubelik A.R.,Livak K.L,et al. DNA polymorphisms amplified by arbitrary primers are useful as genetic markers[J]. Nucleic Acids Research,1990,(18):6531-6535.
[5] 吳玉香. 棉屬遺傳多樣性與栽培棉種間雜交研究[D]. 杭州: 浙江大學, 2007.
[6] 袁菊紅. 中國石蒜屬(LycorisHerb.)種間親緣關系與居群分子標記研究[D]. 南京農業大學, 2007.
[7] 周蘭英. 杜鵑屬植物親緣關系及遺傳多樣性研究[D].四川農業大學, 2008.
[8] 南宮自艷. 松毛蟲屬部分種類遺傳多樣性及影響因素研究[D]. 河北農業大學, 2008.
[9] 萬定榮. 湖北省卷柏屬藥用植物的鑒定和品質研究[D]. 湖北中醫學院, 2006.
(2012–05–29)
S812.8
A
1007-1733(2012)09-0007-03

