谷氨酸棒狀桿菌集成細胞網絡的構建與結構分析
姜金國,宋理富,鄭平,賈士儒,孫際賓
1 天津科技大學生物工程學院,天津 300457
2 中國科學院天津工業生物技術研究所,天津 300308
3 中國科學院系統微生物工程重點實驗室,天津 300308
細胞的各種組成分子在一定的空間內相互作用,協調實現生命的機能。所有的細胞分子及其相互作用,就構成了細胞網絡。細胞網絡是系統生物學的重要研究內容之一,是從全局和整體上把握和理解復雜的代謝生理現象、實現對生命的有效調控、部分改造和從頭設計創造的基礎[1]。盡管生物分子發揮其機能是借助于同一個細胞網絡,但是在傳統研究中,根據研究者研究興趣和生物分子參與的反應類型,往往將細胞網絡人為分解成不同類型的子網絡分別研究,如基因組規 模 代 謝 網 絡 (Genome-scale metabolic network,GSMN)、轉錄調控網絡 (Transcriptional regulatory network,TRN) 和信號轉導網絡(Signal transduction network) 等[2-3]。近年來,研究者已經逐步認識到這種分解所帶來的問題,比如Ma等[4]研究大腸桿菌轉錄調控網絡時,發現其中找不到生物學中最常見的反饋調控,進一步研究表明,這些轉錄反饋調控大部分是通過代謝網絡與轉錄調控網絡協同實現的,僅有7個是發生在轉錄調控這一單一網絡層次上[5]。如今,以單一網絡為基礎的多網絡集成,已成為系統生物學領域的重要發展趨勢,雖然相關的分析軟件還相對缺乏,但是集成分析已經產生了一些重要的結果。Wang等[6]集成了轉錄調控和蛋白質相互作用網絡,認為網絡集成后更接近于真實的生物網絡。Yeang[7]、Herrg?rd[8]和 Chandrasekaran[9]等通過集成調控與代謝網絡,認為集成網絡比單一網絡更能擬合生物的生長狀態。Covert[10]和Lee[11]等整合了信號轉導、代謝和調控網絡,發現多網絡的集成在表型預測時更加準確。
谷氨酸棒狀桿菌是最重要的工業微生物之一,被廣泛應用于氨基酸 (如谷氨酸、賴氨酸等)、維生素 (如泛酸等) 和核酸的工業化生產[12],所生產的產品總量達到每年數百萬噸的規模。谷氨酸棒狀桿菌細胞結構和調控關系相對簡單,對于簡單原料和復雜工業環境適應性好,生長快速,具有較好的分子改造基礎,這些優勢使得谷氨酸棒狀桿菌具備了作為一個通用優秀細胞工廠的基礎,研究者正在研究和改造棒桿菌生產有機酸、醇等大宗化學品[13]。日本學者 Okino等[14]通過改造谷氨酸棒狀桿菌生產丁二酸,其產品濃度和發酵速度都達到了世界領先的水平,成為棒桿菌細胞工廠改造的一個范例。對工業菌株的研究和改造離不開對其細胞網絡的了解和認識,谷氨酸棒狀桿菌的基因轉錄調控和代謝網絡也一直是人們關注的焦點。Ikeda等[15]和Kalinowski等[16]分別完成了谷氨酸棒狀桿菌 Corynebacterium glutamicum ATCC13032菌株的基因組測序,將谷氨酸棒狀桿菌的研究帶入了系統生物學新紀元。Kjeldsen等[17]和 Shinfuku等[18]利用基因組注釋數據庫分別構建了含有約500個反應的谷氨酸棒狀桿菌基因組規模代謝網絡。Brinkrolf等[19]通過對文獻報道數據的整理構建了首個谷氨酸棒狀桿菌轉錄調控網絡。但是目前關于谷氨酸棒狀桿菌的研究大多只是針對單一網絡,由于單一網絡突現的信息割裂的局限性,限制了棒桿菌的研究和開發利用,急需構建基因組規模多網絡集成的細胞網絡。
本研究在前人工作的基礎上,系統整理和分析了谷氨酸棒狀桿菌的轉錄調控信息,對以往報道的代謝網絡進行擴展,并借鑒bow-tie模型[20]的分析方法對谷氨酸棒狀桿菌的代謝網絡進行初步分析,最后以基因-酶-反應關系為接口對轉錄調控網絡和代謝網絡進行集成,為大規模的數據整合和模擬計算分析奠定了基礎。同時文中還以賴氨酸的生物合成為例提出一種從集成細胞網絡中提取代謝調控子網絡的新方法,希望從系統水平上能更好地理解氨基酸等產品的生物合成與調控機制,對工程菌株的定向設計改造提出指導性建議。
1 方法
1.1 TRN數據的采集與處理
從谷氨酸棒狀桿菌調控網絡數據庫CoryneRegNet[21]5.0提取轉錄調控信息,并結合文獻[22-24]對數據補充和校正,去掉不確定的調控關系 sugR (cg2115)→ramA(cg2831) 和 uriR (cg1547)→rbsR (cg1410) (文獻報道中無此調控關系的結論)。用轉錄調控因子 (Transcription factors,TF) 的編碼基因來代替TF本身 (即所有節點均為基因),再根據基因的cg號進行排序,用 VBA程序處理成可視化軟件 Cytoscape[25]和Pajek[26]可識別的文件格式。
1.2 TRN的分層分解
我們借鑒Ma等[27]報道的分層思想對谷氨酸棒狀桿菌的TRN進行分解,首先將出度為0的節點分配到網絡第一層;然后移除這些節點,從剩余的節點中,將出度為0的節點分配到網絡的第二層;依次類推,直到把所有的節點分配到不同的層中。
1.3 GSMN數據采集與處理
1.3.1 數據采集
用Perl程序,從KEGG 數據庫 (http://www. genome.jp/kegg/) 的 Ligand/reaction文件中提取反應數據,結合谷氨酸棒狀桿菌基因組注釋信息(cgb_enzyme和cgb_ko文件),并保留自發反應,便可生成谷氨酸棒狀桿菌GSMN的初始模型。
同時,我們根據文獻數據[17-18]提供的化合物信息,用VBA程序從KEGG/Ligand/compound和glycan數據中搜索對應的化合物編號 (C number和G number),再由化合物編號從KEGG/Ligand/ reaction/reaction數據中搜索到相應的反應號(R number),然后從上述GSMN的初始模型中去掉與文獻數據中反應號重疊的反應,但是保留先前反應的反應對 (Reactant pairs,指底物與產物的對應關系。例如,在反應R00299:ATP+D-葡萄糖<=>ADP+D-葡萄糖 6-磷酸中,D-葡萄糖與D-葡萄糖6-磷酸就是一個反應對) 的信息。例如文獻[18]中的反應:丙酮酸+CoA+NAD+=>乙酰-CoA+CO2+NADH+H+,我們通過VBA程序搜索后發現它與 KEGG中的反應 R00209:丙酮酸+CoA+NAD+<=>乙酰-CoA+CO2+NADH+H+基本匹配,但是KEGG中該反應的可逆性與文獻中的不一致,鑒于文獻中的數據有計算模擬和實驗的支持,而KEGG中的數據僅供參考,所以最終我們把反應 R00209定義為:丙酮酸+CoA+NAD+=>乙酰-CoA+CO2+NADH+H+,反應對信息仍采用KEGG中R00209的反應對 (因為文獻中的反應沒有反應對的信息),即NAD+—NADH、CoA—乙酰-CoA、丙酮酸—乙酰-CoA和丙酮酸—CO2。最后,通過將 KEGG數據和文獻代謝網絡數據相整合,生成谷氨酸棒狀桿菌GSMN模型。
1.3.2 反應方向的重新定義
反應的方向對于生物網絡構建非常重要,但是 KEGG中很多反應的方向并不可靠或沒有定義,對此我們主要以文獻報道數據中的反應方向為主,其余的采用Ma等[28]總結的11條規則重新定義。
1.3.3 反應對的拆分
KEGG中的很多反應并未給出反應對信息,我們根據KEGG對已有反應對的拆分規律RDM (Reaction center, R; Difference region, D; Matched region, M) 模式[29]對信息不全的反應進行拆分。
1.3.4 特殊反應對的刪除
在GSMN中某些化合物出現的頻率很高,被稱為通用代謝物 (Currency metabolites),如ATP、ADP、NADH、H2O、Pi等,它們通常攜帶一些供轉移的小分子化學基團 (如磷酸基、氨基、一碳單位、甲基等)[28]。這些通用代謝物所連接的一些特殊反應對必須去掉,否則整個網絡的路徑將會縮短,不利于后期拓撲結構的分析,而且與實際的生物學意義不符 (如 R00258:L-丙氨酸+2-酮戊二酸<=>丙酮酸+L-谷氨酸中的反應對 L-丙氨酸—L-谷氨酸和 2-酮戊二酸—丙酮酸應該去掉,僅保留L-丙氨酸—丙酮酸和2-酮戊二酸—L-谷氨酸)。
1.4 GSMN的bow-tie結構
為了降低網絡復雜度,谷氨酸棒狀桿菌的GSMN中節點均為代謝物。文中綜合Zhao等[30]和Ma等[20]對代謝網絡的分解方法,首先借助軟件 Pajek求出最大強連通體 (Giant strong component,GSC),然后收縮GSC為單節點,再分別求出 GSC的輸入域 (S) 和輸出域 (P),剩余的節點歸為孤立子集 (Isolated subset,IS) (同一個代謝物在細胞不同部位出現時,分別賦予不同的 ID,例如細胞膜上和細胞內同一代謝物的ID不同)。
1.5 細胞網絡的集成
以基因→酶的關系為接口可以將轉錄調控網絡 (調節子→靶基因) 和代謝網絡 (底物→反應→產物;酶→反應) 整合起來,形成調節子→靶基因→酶→反應和底物→反應→產物的集成細胞網絡模型 (Integrated cellular network,ICN)。
1.6 賴氨酸代謝調控子網絡的提取
1) 在 ICN 數據中用酶的編碼基因將酶替換,然后求葡萄糖的輸出域,從生成的子網絡中提取賴氨酸的輸入域得到節點集A。
2) 將A收縮為單節點 (借鑒Zhao等[30]的方法),并求其 K輸入節點集 (在有向圖中,如果某節點j在K步之內可以到達節點i,那么這些節點j組成的集合就是節點i的K輸入節點集),并從中去掉表示代謝物的節點,僅保留基因節點,組成Genes節點集合。
3) 將 Genes收縮為單節點,從其輸出域中去掉節點集A,得到節點集B。
4) 求A的1近鄰輸入節點集K1,并從K1中提取僅是A的輸入不是B的輸入節點集記為K11;K11中調控關系已明確的節點集記為C,反之記為H;K1中既是A的輸入又是B的輸入的節點集記為K12;K12中調節基因已明確的節點集記為F,反之記為G。
5) 求A的2近鄰輸入節點集K2 (調節子);從K2中提取既調控F又調控C的基因集記為E,僅調控C的記為D。
以上步驟的分析均是借助Pajek軟件和作者自己編寫的VBA程序實現的。
2 結果
2.1 谷氨酸棒狀桿菌TRN的分析
2.1.1 TRN的功能模塊分析
我們最終構建的谷氨酸棒狀桿菌TRN含有660個基因,基于基因功能的生物學意義可以將其劃分為5大功能模塊[19]:A (碳水化合物代謝),B (細胞分裂與分隔),C (常量元素與金屬離子的體內平衡),D (SOS應激反應),E (特殊的生物合成與運輸途徑) (圖1)。
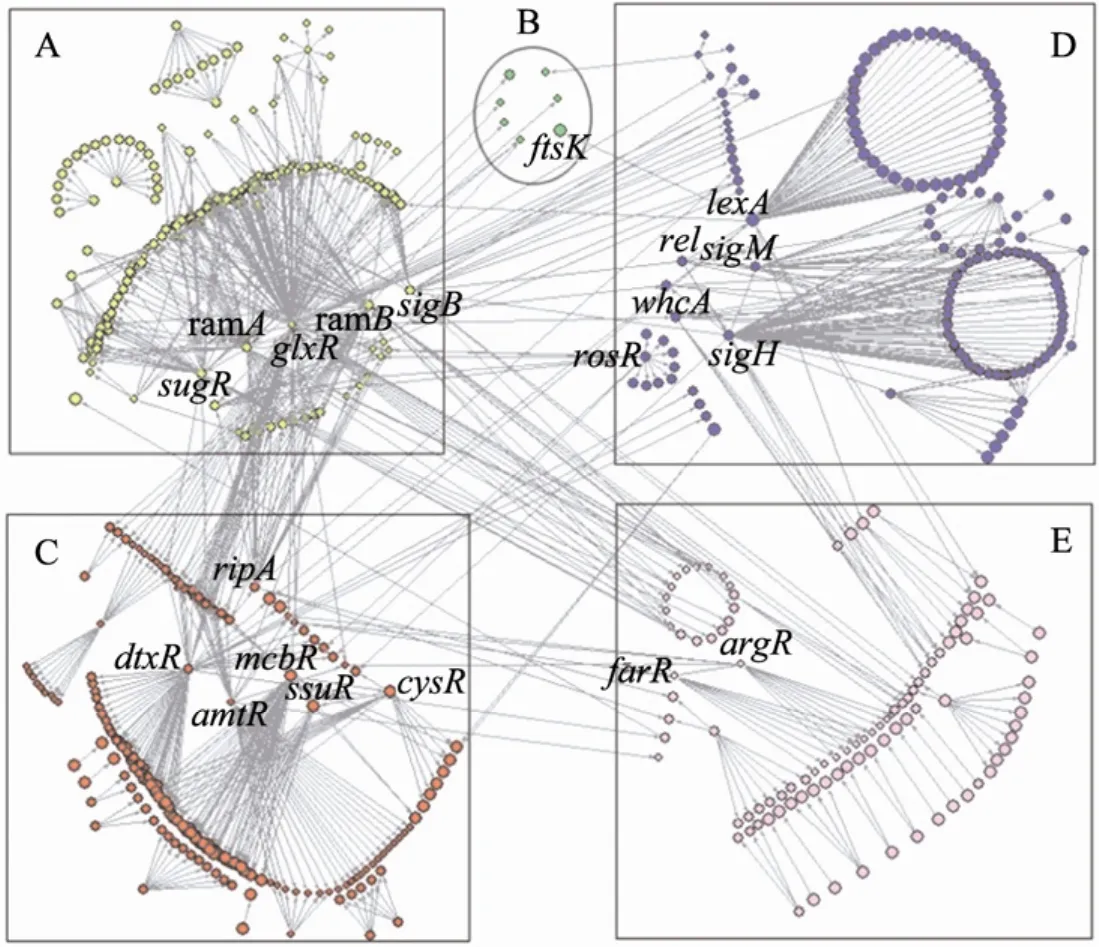
圖1 谷氨酸棒狀桿菌TRN分解Fig. 1 The decomposition of TRN of Corynebacterium glutamicum. (A) Carbohydrate metabolism. (B) Cell division and septation. (C) Macroelement and metal homeostasis. (D) SOS and stress response. (E) Specific biosynthesis and transport pathways.
模塊 A中的調控關系最為復雜 (212個基因),它控制著谷氨酸棒狀桿菌的中心代謝。其中sugR、glxR、ramA、ramB和sigB還具有跨模塊調節的作用,如全局調節子 glxR可以直接調控以上5個模塊中的基因。此外,位于模塊C的dtxR調節子還調控模塊A和模塊D中的基因。模塊E中的ArgR通過阻遏轉氨酶的轉錄從而調控精氨酸代謝,同時抑制谷氨酸脫氫酶的轉錄進而影響谷氨酸轉化和NADP的體內平衡。模塊B中未發現轉錄調節子,基因均受到來自其他模塊調節子的調控,如細胞分裂蛋白基因ftsK的轉錄要受到阻遏蛋白LexA (模塊D) 的抑制。
2.1.2 全局調節子的重新定義
在細胞網絡中,某些調節子可調控20個以上屬于不同轉錄單元的并且從屬于多個功能模塊的靶基因,被稱為全局調節子[31-32]。Brinkrolf等[22]通過搜索調節子直接調控的靶基因和功能模塊的數量,認為在谷氨酸棒狀桿菌TRN中只存在一個全局調節子glxR (cg0350)。而事實上,有些調節子的靶基因同樣也是調節基因。因此,我們引入調節子的輸出域 (Output domain) 來替換其出度 (Out-degree) 重新分析,并用自己編寫的VBA程序廣度搜索了調節子直接和間接調控的總模塊數(表1)。
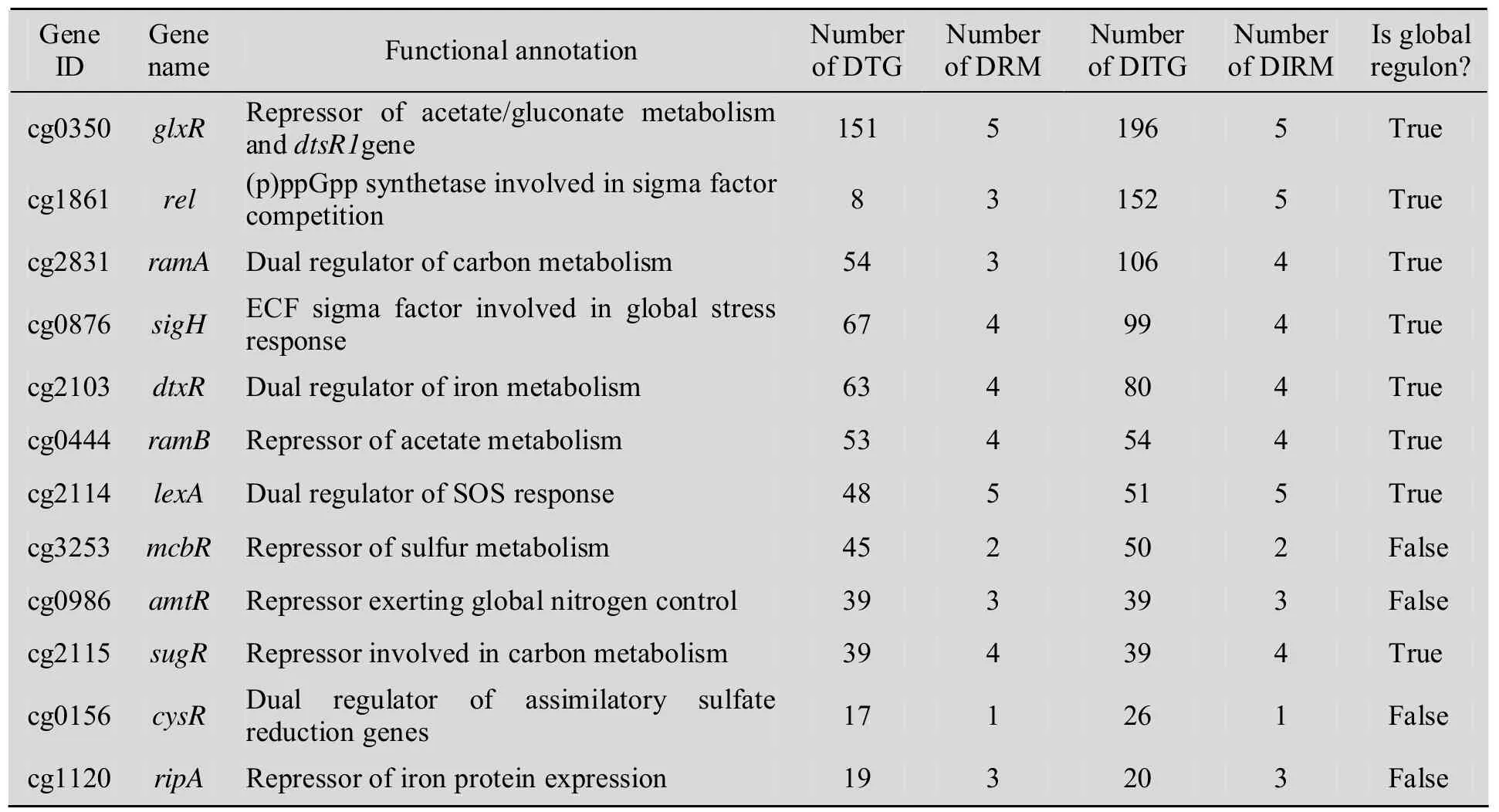
表1 谷氨酸棒狀桿菌調節子調控的目標基因與模塊的統計分析Table 1 Statistic analysis of target genes and modules which are regulated by regulons in Corynebacterium glutamicum
結果我們發現了8個調節子調控影響范圍廣泛 (直接和間接調控的目的基因總數不少于20,調控模塊總數不少于 4),可以認定為全局調節子:glxR (cg0350),lexA (cg2114),ramA (cg2831),ramB (cg0444),sigH (cg0876),sugR (cg2115),rel (cg1861) 和dtxR (cg2103)。雖然有些調節子直接調控的功能模塊并不多,但是它們還可以通過調控其他調節子間接調控一些模塊。如 rel (cg1861) 通過調控調節子sigH (cg0876) 實現對cg2810 (Na+/H+泵轉運系統,模塊C) 的間接調控。這8個全局調節子共調控387個基因,調控范圍覆蓋了整個TRN的59%。
2.1.3 TRN的分層分解
借助網絡分析軟件Pajek我們發現谷氨酸棒狀桿菌TRN中無強連通體存在,這與枯草芽胞桿菌[33]和大腸桿菌[5]有所不同,說明在 TRN中無反饋環 (基因A調控基因B,基因B也直接或間接調控基因A) 的存在,因此可以對TRN按層次分解,如圖2所示。
其中底層的基因最多 (572個),其次為第2層 (68個)。分層后可以清楚地看到,rel (cg1861)和dtxR (cg2103) 分別位于頂層和第4層,是重要的全局調節子。并且基因之間的調控關系還可以跨層實現,如rel (cg1861) 調節子對底層基因cg0877 (rshA) 的調控不僅可以通過sigH間接實現,還能直接完成,這可以很好地解釋谷氨酸棒狀桿菌TRN的平均路徑長度只有1.27,即存在調控捷徑。
2.2 GSMN分析
2.2.1 GSMN模型
經過對公共數據庫和文獻數據的收集和處理,我們最終構建的GSMN模型含有1 384個生化反應 (其中978個為不可逆反應,406個可逆反應) 和1 276個代謝物,如圖3A所示。去除經過通用代謝物的連接后,連接數由原來的3 783縮減為2 803,網絡直徑和平均路徑[28]分別由原來的17和4.74變成了32和9.66,網絡明顯得到了簡化,如圖3B所示。

圖2 谷氨酸棒狀桿菌TRN的分層結構Fig. 2 The hierarchical structure of TRN of Corynebacterium glutamicum.

圖3 谷氨酸棒狀桿菌GSMN模型. (A) 含有經過通用代謝物連接的GSMN. (B) 不含經過通用代謝物連接的GSMNFig. 3 The GSMN model of Corynebacterium glutamicum. All the nodes represent metabolites. (A) GSMN including the connections through currency metabolites. (B) GSMN not including the connections through currency metabolites. The red lines with arrows correspond to irreversible reactions and the blue lines without arrows to reversible reactions.
2.2.2 Bow-tie結構
經過分析,我們發現谷氨酸棒狀桿菌的基因組規模代謝網絡GSMN共102個強連通體 (節點數≥2),其中最大的強連通體GSC包含456個代謝物,占整個代謝網絡的36% (圖4)。GSC的平均路徑為9.08,而整個網絡的平均路徑為9.66,這說明與網絡中的其他模塊相比GSC中節點之間的聯系更加緊密。按照KEGG對代謝反應的類別劃分,我們將GSC分解成10大功能模塊(圖5)。結果顯示GSC幾乎覆蓋了所有的代謝功能,是整個代謝網絡的核心,并且模塊之間并非獨立存在,而是通過“代理”(Broker)[27]或橋(Bridges)[34](表2) 相互聯系。

圖4 谷氨酸棒狀桿菌GSMN的bow-tie結構Fig. 4 The bow-tie structure of GSMN for Corynebacterium glutamicum. GSC: giant strong component; S: substrate subset; P: product subset; IS: isolated subset.
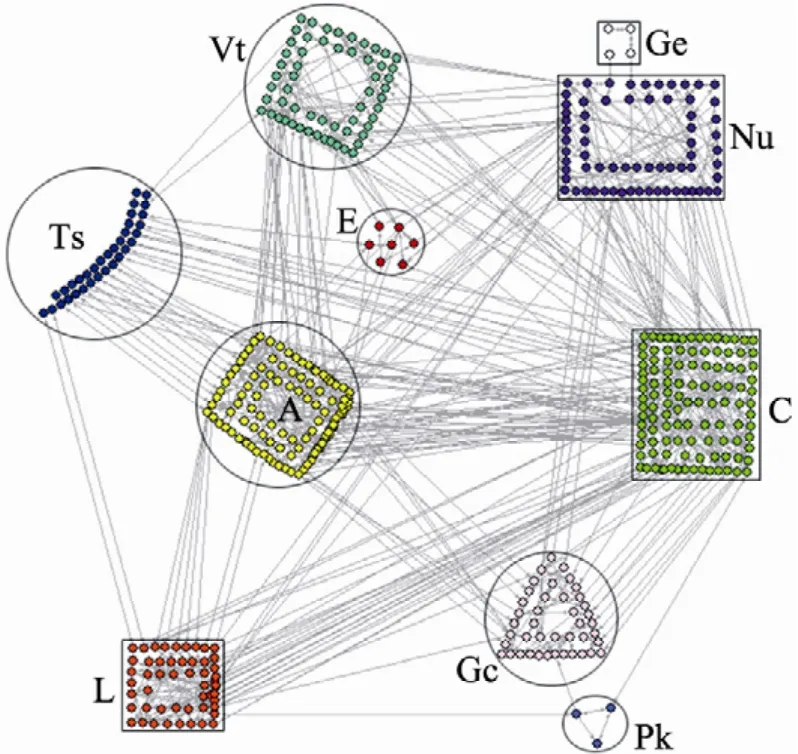
圖5 GSC的分解Fig. 5 The decomposition of GSC. A: amino acid metabolism; C: carbohydrate metabolism; E: energy metabolism; Ts: membrane transport or signal transduction; Gc: glycan biosynthesis and metabolism; Ge: genetic information processing; L: lipid metabolism; Nu: nucleotide metabolism; Pk: metabolism of terpenoids and polyketides; Vt: metabolism of cofactors and vitamins.
2.2.3 GSC的中心性分析
緊密度中心性 (Closeness centrality,CC)[27]和介數中心性 (Betweenness centrality,BC)[35-36]是用來衡量節點地位的重要參數,值越大節點就越重要,后者也常用來尋找網絡的“代理”[27]或橋[34]以及分解網絡[37]。經過計算分析,我們找到了 29個中心代謝物 (Central metabolites)[20](表2)。
很多化合物不僅僅參與了一個功能模塊中的反應,如丙酮酸 (Pyruvate) 不僅是糖酵解和TCA的樞紐,而且連接著碳水化合物代謝和氨基酸代謝,并通過反應R05553 (4-氨基-4脫氧分支酸<=>4-氨基苯甲酸+丙酮酸) 參與葉酸鹽的生物合成。乙酰-CoA (Acetyl-CoA) 是網絡的輸出中心,連接著糖酵解、TCA、肽聚糖生物合成和脂類代謝以及氨基酸代謝。草酰乙酸(Oxaloacetate) 是TCA中重要的代謝物,同時經反應 R00355 (草酰乙酸+L-谷氨酸<=>L-天冬氨酸+α-酮戊二酸) 使碳水化合物代謝與氨基酸代謝連接起來。
2.3 集成細胞網絡的構建
我們以基因、酶、反應和代謝物為節點,相互作用關系為弧 (邊),去除通用代謝物參與的特殊反應對以及文獻和數據庫之間的冗余部分,最終所構建的谷氨酸棒狀桿菌集成細胞網絡包括1 050個基因,785個酶,1 384個反應,1 276個代謝物,88個調節子,999對轉錄調控關系(圖6)。

表2 GSC網絡中中心性最大的幾種中心代謝物Table 2 The top central metabolites with higher centrality in GSC

圖6 谷氨酸棒狀桿菌集成細胞網絡Fig. 6 The integrated cellular network of Corynebacterium glutamicum. pink cirle: gene; blue triangle: enzyme; green diamond: metabolite; green yellow square: reaction; →activationrepression.
利用Pajek軟件從構建的集成細胞網絡中,我們找到了 8個基于轉錄與代謝調控的反饋環路。例如,dtxR編碼的轉錄因子DtxR可以抑制基因hrrA (對hmuO的轉錄有激活作用) 和hmuO的轉錄,hmuO編碼的血紅素氧化酶 (Heme oxygenase,HmuO) 可以催化亞鐵血紅素 Heme生成鐵離子,而鐵離子又是DtxR發揮作用時必需的輔因子,這樣就構成了一個反饋環路,如圖7A所示。該反饋回路對于維持 Heme的體內平衡[38]和鐵代謝[39]具有重要意義。此外,在天冬氨酸家族 (如甲硫氨酸[40-42]) 和一些芳香族氨基酸的生物合成中也同樣存在轉錄與代謝協同作用的復雜調控關系。例如,在色氨酸的合成中,幾個關鍵酶 ANS (Anthranilate synthase)、PRT (Anthranilate phosphoribosyltransferase) 和 TS (Tryptophan synthase) 除了受終產物色氨酸的反饋抑制外,其編碼基因的轉錄還受到色氨酸的衰減作用 (Attenuation) 的控制[43],如圖7B所示。這些發生在細胞內的轉錄與代謝雙層次復合調控關系在單一的代謝網絡[17-18]或轉錄調控網絡[19]中是無法體現的。
2.4 L-賴氨酸生物合成代謝調控子網絡的提取
分別以葡萄糖為底物約束和賴氨酸為產物約束,從集成細胞網絡中提取了賴氨酸生物合成的代謝調控子網絡 (圖8)。它涉及的代謝功能模塊有氨基酸代謝、碳水化合物代謝、能量代謝、膜運輸與信號轉導、多糖合成與代謝、遺傳信息處理、脂類代謝、核酸代謝、萜類與聚酮代謝以及輔因子與維生素代謝,而傳統的賴氨酸合成途徑[44]僅包括糖酵解、TCA以及賴氨酸、蘇氨酸等氨基酸合成途徑。
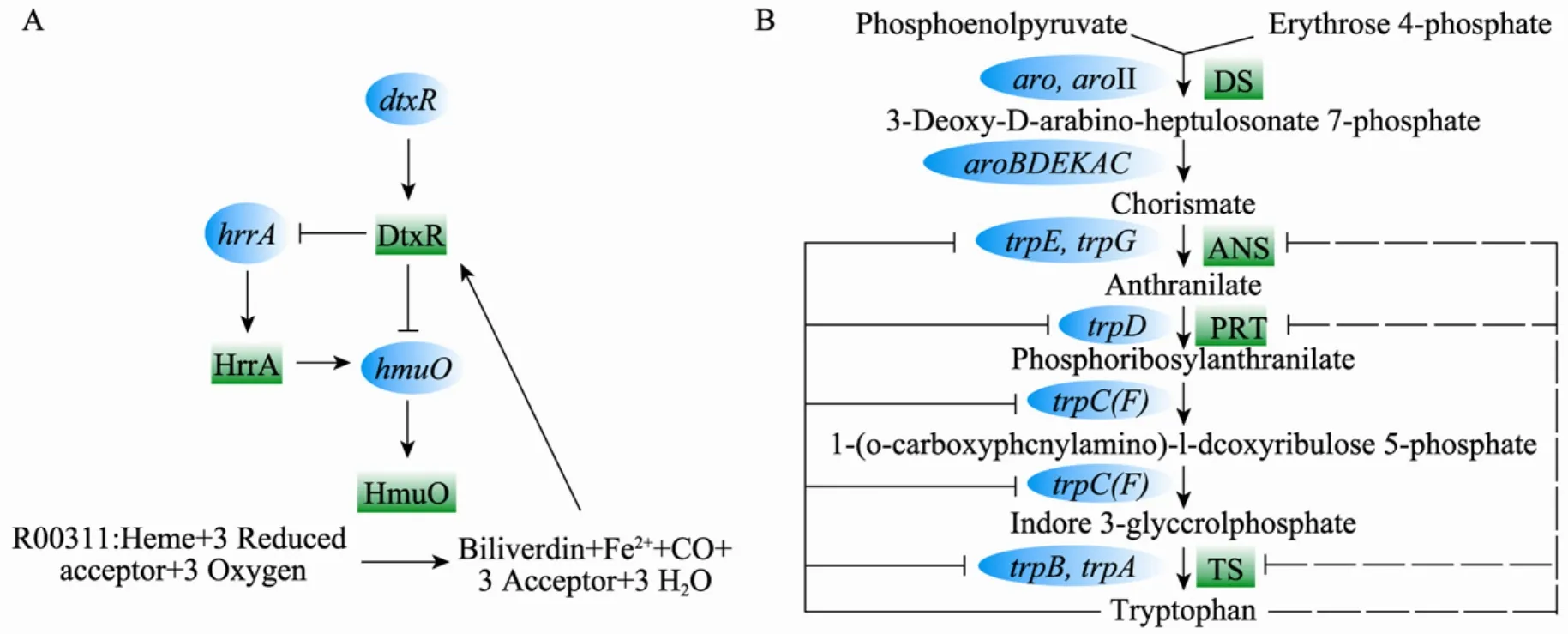
圖7 谷氨酸棒狀桿菌中的復合調控回路. (A) 血紅素鐵代謝的轉錄與代謝調控反饋環. (B) 色氨酸的生物合成與調控Fig. 7 Complex regulation loop in Corynebacterium glutamicum. (A) A feedback loop for transcriptional and metabolic regulation of Heme iron. (B) The biosynthetic pathway of tryptophan and its regulation. DS: 3-deoxy-D-arabino-heptulosonate 7-phosphate synthase; blue ellipse: gene; green box: corresponding protein; →activation; ━┫repression. The dotted lines indicate feedback inhibition.
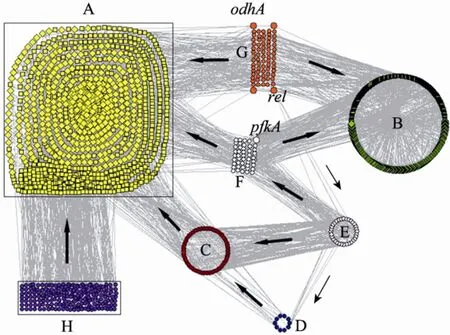
圖8 賴氨酸生物合成代謝調控子網絡Fig. 8 The metabolic and regulatory sub-network for lysine biosynthesis. (A) Lysine biosynthesis. (B) Other biosynthesis. C, F, G and H represent enzyme-coding genes. D and E represent regulons. Circle: gene; diamond: metabolite; square: reaction; arrow: the direction of material interaction.
模塊C中的基因可以編碼催化模塊A中代謝反應的酶,同時受到模塊D和模塊E的調控。模塊F和模塊G中的基因所編碼的酶不僅可以催化賴氨酸生物合成,還可以催化模塊 B中的反應,例如pfkA (cg1409) 編碼的磷酸果糖激酶既是中心代謝途徑的重要限速酶 (R00756,模塊 A),也是塔格糖酵解的重要酶 (R03236,模塊B),同時受到全局調節子glxR和sugR以及sigB (σ因子) 的調控 (模塊 E)。rel (cg1861,PPGPP合成酶,模塊 E) 不僅可以編碼催化R00336 (模塊A)和R00429 (模塊B)的酶,而且還是全局調節子。此外,模塊F中的基因受到模塊E中調節子的調控,而模塊G和模塊H所對應的調節基因,目前為止尚不清楚。
3 討論
關于谷氨酸棒狀桿菌轉錄調控網絡和代謝網絡的研究已有相關文獻介紹,但是對于集成細胞網絡的分析尚未發現有人報道。文中從網絡數據庫和文獻信息提取數據,成功構建了谷氨酸棒狀桿菌的集成細胞網絡模型,網絡涉及1 384個生化反應和999對調控關系。通過后續的網絡分析,發現了一些在單一網絡中很難體現的轉錄與代謝雙層次協同作用的復合調控關系,這為重新設計和優化改造谷氨酸棒狀桿菌,發展新型細胞工廠奠定了基礎。
通過對基因轉錄調控網絡的分析,我們重新定義了8個全局調節子,其調控范圍覆蓋整個轉錄調控網絡的59%。轉錄調控網絡可分為5層,并且有調控捷徑的存在,這可能有利于生物體提高自身對環境變化的適應能力。
我們在谷氨酸棒狀桿菌的代謝網絡中找到了和大腸桿菌類似的bow-tie結構,其中GSC的強連通性可能有利于提高生物體的魯棒性(Robustness)。通過對GSC的進一步分析,我們找到了 29個中心代謝物,它們像橋梁一樣將一個個分開的反應模塊緊密地連接在一起,對于微生物而言,這種結構可能有利于節省空間和資源,使生物代謝更加經濟高效。
最后,基于集成細胞網絡我們提出了一種代謝調控子網絡的提取方法,與傳統意義上的代謝途徑相比,前者的代謝反應更加全面,物質間相互作用關系也更為復雜。應用這種提取方法,我們研究了從葡萄糖合成賴氨酸的代謝調控子網絡,發現在這個復雜的子網絡中,有些基因不僅編碼賴氨酸合成代謝途徑中的酶,同時還是其他代謝反應的酶編碼基因 (如 cg1280 (odhA) 和cg1409 (pfkA)),更有甚者,如rel不僅編碼賴氨酸合成途徑中涉及的酶,同時還是全局調節子。這一結果告訴我們,在運用基因工程手段對菌株進行定向改造時,應該從系統生物學角度出發,通盤考慮轉錄調控和代謝反應,否則某些基因的改變可能會導致細胞整體代謝反應失去平衡,整個細胞生理機能發生改變。
本研究的工作,僅僅是谷氨酸棒狀桿菌系統生物學工作的開始。在本研究提出的集成細胞網絡的基礎上,能夠集成更多的實驗數據,包括轉錄組、蛋白質組、代謝組、通量組數據,對發酵過程的動態數據進行全面地整合分析,將發現更多的代謝調控機制,進一步完善集成型細胞網絡模型。同時,發展整合細胞網絡的模擬計算方法,從代謝和調控的全局開發新型細胞工廠設計方法,提出最佳基因組改造策略,有可能大幅度改進谷氨酸棒狀桿菌的性能,為新型生物制造產業的發展貢獻力量。致謝 感謝英國愛丁堡大學馬紅武博士的修改意見。
[1] Kitano H. Systems biology: a brief overview. Science, 2002, 295(5560): 1662?1664.
[2] Kim TY, Kim HU, Lee SY. Data integration and analysis of biological networks. Curr Opin Biotechnol, 2010, 21(1): 78?84.
[3] Sik KJ, Yun H, Kim HU, et al. Resources for systems biology research. J Microbiol Biotechnol, 2006, 16(6): 832?848.
[4] Ma HW, Buer J, Zeng AP. Hierarchical structure and modules in the Escherichia coli transcriptional regulatory network revealed by a new top-down approach. BMC Bioinformatics, 2004, 5: 199.
[5] Ma HW, Kumar B, Ditges U, et al. An extended transcriptional regulatory network of Escherichia coli and analysis of its hierarchical structure and network motifs. Nucl Acids Res, 2004, 32(22): 6643?6649.
[6] Wang YC, Chen BS. Integrated cellular network of transcription regulations and protein-protein interactions. BMC Syst Biol, 2010, 4: 20.
[7] Yeang CH, Vingron M. A joint model of regulatory and metabolic networks. BMC Bioinformatics, 2006, 7: 332.
[8] Herrg?rd MJ, Lee BS, Portnoy V, et al. Integrated analysis of regulatory and metabolic networks reveals novel regulatory mechanisms in Saccharomyces cerevisiae. Genome Res, 2006, 16(5): 627?635.
[9] Chandrasekaran S, Price ND. Probabilistic integrative modeling of genome-scale metabolic and regulatory networks in Escherichia coli and Mycobacterium tuberculosis. Proc Natl Acad Sci USA, 2010, 107(41): 17845?17850.
[10] Covert MW, Xiao N, Chen TJ, et al. Integrating metabolic, transcriptional regulatory and signal transduction models in Escherichia coli. Bioinformatics, 2008, 24(18): 2044?2050.
[11] Lee JM, Gianchandani EP, Eddy JA, et al. Dynamic analysis of integrated signaling, metabolic, and regulatory networks. PLoS Comput Biol, 2008, 4(5): e1000086.
[12] Eggeling L, Bott M. Handbook of Corynebacterium glutamicum. Boca Raton: CRC Press, 2005: 1?616.
[13] Wendisch VF, Bott M, Eikmanns BJ. Metabolic engineering of Escherichia coli and Corynebacterium glutamicum for biotechnological production of organic acids and amino acids. Curr Opin Microbiol, 2006, 9(3): 268?274.
[14] Okino S, Noburyu R, Suda M, et al. An efficient succinic acid production process in a metabolically engineered Corynebacterium glutamicum strain. Appl Microbiol Biotechnol, 2008, 81(3): 459?464. [15] Ikeda M, Nakagawa S. The Corynebacterium glutamicum genome: features and impacts on biotechnological processes. Appl Microbiol Biotechnol, 2003, 62(2/3): 99?109.
[16] Kalinowski J, Bathe B, Bartels D, et al. The complete Corynebacterium glutamicum ATCC 13032 genome sequence and its impact on the production of L-aspartate-derived amino acids and vitamins. J Biotechnol, 2003, 104(1/3): 5?25.
[17] Kjeldsen KR, Nielsen J. In silico genome-scale reconstruction and validation of the Corynebacterium glutamicum metabolic network. Biotechnol Bioeng, 2009, 102(2): 583?597.
[18] Shinfuku Y, Sorpitiporn N, Sono M, et al. Development and experimental verification of a genome-scale metabolic model for Corynebacterium glutamicum. Microb Cell Fact, 2009, 8: 43.
[19] Brinkrolf K, Brune I, Tauch A. The transcriptional regulatory network of the amino acid producer Corynebacterium glutamicum. J Biotechnol, 2007, 129(2): 191?211.
[20] Ma HW, Zeng AP. The connectivity structure, giant strong component and centrality of metabolic networks. Bioinformatics, 2003, 19(11): 1423?1430.
[21] Baumbach J, Wittkop T, Kleindt CK, et al. Integrated analysis and reconstruction of microbial transcriptional gene regulatory networks using CoryneRegNet. Nat Protoc, 2009, 4(6): 992?1005. [22] Brinkrolf K, Schr?der J, Pühler A, et al. The transcriptional regulatory repertoire of Corynebacterium glutamicum: reconstruction of the network controlling pathways involved in lysine and glutamate production. J Biotechnol, 2010, 149(3): 173?182.
[23] Nentwich SS, Brinkrolf K, Gaigalat L, et al. Characterization of the LacI-type transcriptional repressor RbsR controlling ribose transport in Corynebacterium glutamicum ATCC 13032. Microbiology, 2009, 155(1): 150?164.
[24] Engels V, Lindner SN, Wendisch VF. The global repressor SugR controls expression of genes of glycolysis and of the L-Lactate dehydrogenase LdhA in Corynebacterium glutamicum. J Bacteriol, 2008, 190(24): 8033?8044.
[25] Cline MS, Smoot M, Cerami E, et al. Integration of biological networks and gene expression data using Cytoscape. Nat Protoc, 2007, 2(10): 2366?2382.
[26] Batagelj V, Mrvar A. Pajek-analysis and visualization of large networks//Jünger M, Mutzel P. Graph Drawing Software. Berlin: Springer, 2003: 77?103.
[27] Ma HW, Silva MR, Sun JB, et al. Reconstruction and structural analysis of metabolic and regulatory networks//Choi S. Introduction to Systems Biology. New Jersey: Humana Press, 2007: 124?146.
[28] Ma HW, Zeng AP. Reconstruction of metabolic networks from genome data and analysis of their global structure for various organisms. Bioinformatics, 2003, 19(2): 270-277.
[29] Kotera M, Okuno Y, Hattori M, et al. Computational assignment of the EC numbers for genomic-scale analysis of enzymatic reactions. J Am Chem Soc, 2004, 126(50): 16487?16498.
[30] Zhao J, Tao L, Yu H, et al. Bow-tie topological features of metabolic networks and the functional significance. Chin Sci Bull, 2007, 52(8): 1036?1045.
[31] Rodionov DA. Comparative genomic reconstruction of transcriptional regulatory networks in bacteria. Chem Rev, 2007, 107(8): 3467?3497.
[32] Moreno-Campuzano S, Janga SC, Pérez-Rueda E. Identification and analysis of DNA-binding transcription factors in Bacillus subtilis and other Firmicutes-a genomic approach. BMC Genomics, 2006, 7: 147.
[33] Liu Y, Zhao XM, Ma HW. The structure and decomposition of the transcriptional regulatory network of Bacillus subtilis. Chin J Bioprocess Eng, 2007, 5(3): 58?63.劉楊, 趙學明, 馬紅武. 枯草芽孢桿菌轉錄調控網絡的連接結構及分解. 生物加工過程, 2007, 5(3): 58?63.
[34] Pinney JW, Westhead DR. Betweenness-based decomposition methods for social and biological networks. In: Barber S, Baxter PD, Mardia KV, et al. Interdisciplinary Statistics and Bioinformatics. Leeds, UK: Leeds University Press, 2006: 87?90. [35] Freeman LC. A set of measures of centrality based on betweenness. Sociometry, 1977, 40(1): 35?41. [36] Brandes U. A faster algorithm for betweenness centrality. J Math Sociol, 2001, 25(2): 163?177.
[37] Holme P, Huss M, Jeong H. Subnetwork hierarchies of biochemical pathways. Bioinformatics, 2003, 19(4): 532?538.
[38] Frunzke J, G?tgens C, Brocker M, et al. Control of heme homeostasis in Corynebacterium glutamicum by the two-component system HrrSA. J Bacteriol, 2011, 193(5): 1212?1221.
[39] Brune I, Werner H, Hüser AT, et al. The DtxR protein acting as dual transcriptional regulator directs a global regulatory network involved in iron metabolism of Corynebacterium glutamicum. BMC Genomics, 2006, 7: 21.
[40] Rey DA, Pühler A, Kalinowski J. The putative transcriptional repressor McbR, member of the TetR-family, is involved in the regulation of the metabolic network directing the synthesis of sulfur containing amino acids in Corynebacterium glutamicum. J Biotechnol, 2003, 103(1): 51?65.
[41] Rey DA, Nentwich SS, Koch DJ, et al. The McbR repressor modulated by the effector substance S-adenosylhomocysteine controls directly the transcription of a regulon involved in sulphur metabolism of Corynebacterium glutamicum ATCC 13032. Mol Microbiol, 2005, 56(4): 871?887.
[42] Kr?mer JO, Heinzle E, Schr?der H, et al. Accumulation of homolanthionine and activation of a novel pathway for isoleucine biosynthesis in Corynebacterium glutamicum McbR deletion strains. J Bacteriol, 2006, 188(2): 609?618.
[43] Ikeda M. Towards bacterial strains overproducing L-tryptophan and other aromatics by metabolic engineering. Appl Microbiol Biotechnol, 2006, 69(6): 615?626.
[44] Wittmann C. Analysis and engineering of metabolic pathway fluxes in Corynebacterium glutamicum. Adv Biochem Eng Biotechnol, 2010, 120: 21?49.