茶陵野生稻及其導入后代間的遺傳差異RAPD分析
蔣斌元,張 齊,康 敏,王勝利,龔建華,洪亞輝
(1.湖南農業大學生物科學技術學院,湖南 長沙 410128;2.株洲市農業科學研究所,湖南 株洲 412000)
水稻是一種重要的糧食作物,是人類主要的糧食來源,提高水稻產量以及防治病蟲害一直是科研工作者們努力的方向[1]。普通野生稻是栽培稻的近緣祖先,保有許多寶貴的基因資源,是水稻突破性育種與生產的重要基因源[2-4]。在茶陵已發掘的新石器時代大塘文化遺址中發現了已碳化的谷物堆,由此可見,茶陵地區谷物種植歷史悠久,茶陵野生稻具有重要的研究價值。湖南農業大學細胞生物學研究室與株洲農業科學研究所合作,將茶陵野生稻的基因組DNA采用花粉管通道法[5]導入受體栽培稻R9810、RCP08-29,其中R9810這一系中獲得的后代已培育到第4代,表型各異,表現出明顯的耐寒能力并且具有對株洲當地稻瘟病菌小種的抗性。筆者以茶陵野生稻及其導入后代為研究對象,輔以受體材料為對照,采用RAPD方法[6-9]研究總基因組導入后在遺傳上所造成的影響,旨在配合大田表型篩選,為后續有目的性地篩選試驗材料提供科學依據。
1 材料與方法
1.1 材料
供試材料為茶陵野生稻(W)、超級稻R9810(C)、 導入后代D4(1:ys09203;2:ys09004;3:ys090015;4:ys090026;5:ys090027c)
1.2 方法
1.2.1 總DNA的提取 將供試材料洗凈剪碎分別置于研缽中,加入液氮研磨破碎細胞,使用CTAB法[10]提取DNA,電泳檢測。
1.2.2 RAPD擴增 經過多次篩選確定使用引物(表1)與25 μL反應體系,PCR反應程序為94℃預變性 5 min;94℃變性 40 s,36℃復性 40 s,72℃延伸1 min,共計40個循環;最后72℃延伸7 min。引物濃度為10 μM。隨機引物由華大基因合成,利用隨機引物對各樣品基因組DNA進行擴增,凝膠電泳檢測擴增產物。
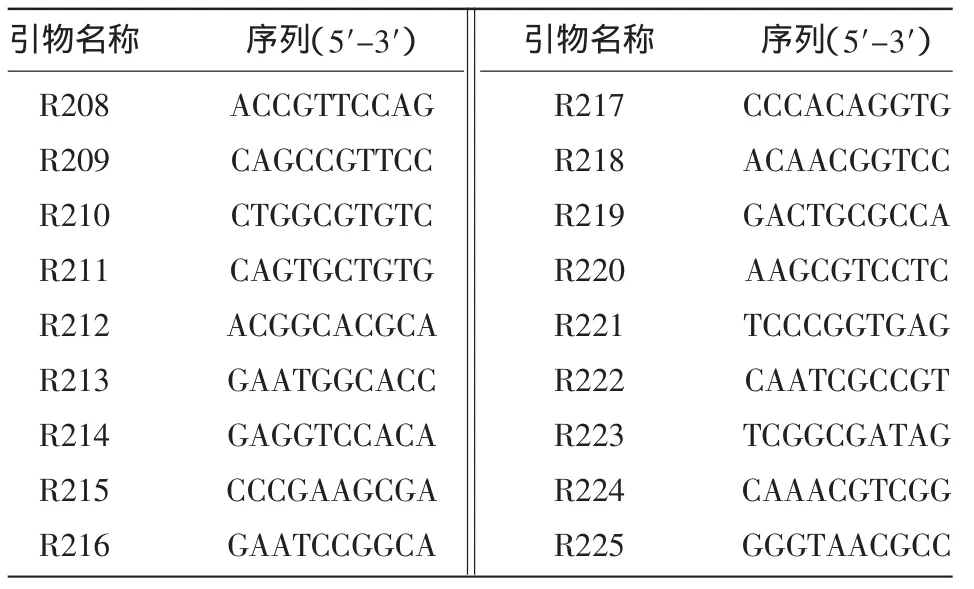
表1 RAPD隨機引物序列
1.2.3 數據分析 對凝膠電泳圖譜進行統計,使用統計類軟件NTSYSpc 2.1,基于非加權組平均法(UPGMA)對所得結果進行聚類分析[11-13]。
2 結果與分析
2.1 水稻基因組DNA的提取
提取的水稻基因組DNA經1.0%瓊脂糖凝膠電泳檢測后,顯示條帶清晰(圖1)。這說明所提取的水稻基因組DNA大分子較為完整,雜質和DNA的降解均比較少,符合RAPD所需要的PCR模板要求。經蛋白核酸測定儀測量,所提取的基因組DNA的A260/A280值為2.10,基本符合DNA的純度要求。
2.2 RAPD分析
用篩選出的18個RAPD隨機引物對各樣品基因組DNA進行擴增,圖2為部分RAPD電泳圖。
對顯示的凝膠電泳圖譜進行統計,使用1和0表示帶的有和無(表2)。使用統計類軟件NTSYSpc 2.1基于UPGMA算法對所得結果進行聚類分析,做出聚類圖(圖3)。基于該聚類圖所顯示的遺傳關系,發現茶陵野生稻總DNA經花粉管通道法導入栽培稻后,其導入后代在遺傳關系上表現各異,有仍然趨向于受體R9810的,如5號導入后代,也有隨機插入DNA片段后在遺傳距離上發生了改變的,如 1、2、3、4 號導入后代。

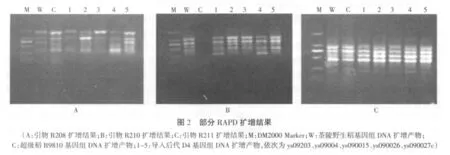

表2 隨機引物RAPD圖譜條帶讀取結果

3 討 論
采用花粉管通道法將茶陵野生稻的總基因組DNA導入栽培稻后,在表型上獲得了許多變異,筆者使用RAPD法在分子水平上對基因組導入所引起的變異進行了分析說明。研究結果表明,茶陵野生稻在進化上處于比較原始的位置,受體R9810與5號導入后代有著較近的親緣關系,5號材料在遺傳上并未出現很大的改變,而1、2、3、4號導入后代在遺傳上則受到了較大的改變,這些變化可能是由于DNA片段的隨機插入而引起,亦或者是僅限于所選引物進行擴增才會顯示的結果。總而言之,RAPD法能在某種程度上顯示出采用花粉管通道法將供體基因組DNA導入受體后所產生的后代在基因組水平的變化,能為后續選育試驗提供一定的參考。與表現型相結合,通過對花粉管通道法導入總DNA后獲得的大量后代進行歸類研究,能提高分類的準確性。
[1]康美花,曹豐生,陳紅萍,等.水稻稻瘟病抗性基因研究進展及其在育種上的應用[J].江西農業學報,2010,22(2):95-98.
[2]鄧啟云,袁隆平,梁鳳山,等.野生稻高產基因及其分子標記輔助育種研究[J].雜交水稻,2004,19(1):6-10.
[3]盧寶榮,葛 頌,桑 濤,等.稻屬分類的現狀及存在問題[J].植物分類學報,2001,39(4):373-388.
[4]呂學蓮,白海波,蔡正云,等.普通野生稻(Oryza rufipogon)DNA導入栽培稻后代的發芽耐鹽性研究 [J].安徽農業科學,2010,38(36):20556-20558.
[5]龔蓁蓁,沈慰芬,周光宇,等.受粉后外源DNA導入植株技術-通過花粉管通道進入胚囊 [J].中國科學(B輯),1988,(6):611-614.
[6]梁美霞,李景富,許向陽.RAPD技術在蔬菜遺傳育種上的應用[J].分子植物育種,2003,1(5-6):737-740.
[7]許 麗,李明瑩,林 風.DNA分子標記及其在作物遺傳育種中的應用[J].沈陽師范大學學報(自然科學版),2006,24(4):466-469.
[8]Mengoni A,Gori A,Bazzicalupo M.Use of RAPD and microsatellite(SSR)variation to assess genetic relationships among populations of tetraploid alfalfa[J].Medicago Sativa Plant Breeding,2000,119(4):311-317.
[9]Ying X.Detection of Purity of Thai Hom Mali Rice by RAPD[J].Agricultural Science&Technology,2011,12(11):1565-1568.
[10]Murray M G,Thompson W F.Rapid isolation of high molecular weight plant DNA [J].Nucleic Acids Res,1980,(8):4321-4325.
[11]薛慶中.DNA和蛋白質序列數據分析工具(第二版)[M].北京:科學出版社,2010.
[12]Nozaki T,Kumazaki A,Koba T,et al.Linkage analysis among loci for RAPDs,is ozymes and some agronomic traits in Brassica campestris L[J].Euphytica,1997,95:115-123.
[13]漆小泉,朱德蔚,沈 鏑,等.大白菜和紫菜薹自交染色體組DNA 的 RAPD 分析[J].園藝學報,1995,22(3):256-262.

