四川牦牛奶和曲拉中九株乳酸菌的分子鑒定
包秋華, 呂嬙, 于 潔, 王宏梅, 張和平, 孫天松
(內蒙古農業大學 乳品生物技術與工程教育部重點實驗室,內蒙古 呼和浩特 010018)
四川牦牛奶和曲拉中九株乳酸菌的分子鑒定
包秋華, 呂嬙, 于 潔, 王宏梅, 張和平, 孫天松*
(內蒙古農業大學 乳品生物技術與工程教育部重點實驗室,內蒙古 呼和浩特 010018)
為準確鑒定分離自我國四川鮮牦牛奶和曲拉中9株乳酸菌到種水平,作者運用16S r DNA序列分析、rec A基因特異擴增和hsp60-RFLP等多種分子技術對分離自我國四川地區鮮牦牛奶和曲拉中的9株乳酸菌進行分類鑒定。結果證實,16S r DNA序列分析法可將9株乳酸菌初步歸類為植物乳桿菌群(4株),腸膜明串珠菌(4株),瑞士乳桿菌(1株)。由于16S r DNA序列分析法不能區分植物乳桿菌和戊糖乳桿菌,為了進一步鑒定植物乳桿菌群中的4株菌,繼續采用recA基因特異擴增和hsp60-RFLP技術對其細分,結果表明rec A基因特異擴增和hsp60-RFLP的方法均能很好地把植物乳桿菌群中的4株菌鑒定到種水平,且均為植物乳桿菌。
乳酸菌;分類鑒定;16S r DNA;recA基因;hsp60基因;RFLP
乳酸菌是一類革蘭氏陽性、細胞形態為桿菌或球菌、過氧化氫酶為陰性、不形成內生孢子、利用可發酵糖產生50%以上乳酸的細菌的通稱[1]。廣泛地存在于自然界、動植物體、乳制品和發酵食品中,且大多數為非致病菌,屬于有益微生物,具有改善腸道菌群、增強人體免疫力、抗腫瘤、降低血液膽固醇、降血壓等功能[2],是人體正常菌群。牦牛乳和曲拉具有獨特的生態學特性和較高的經濟價值,同時含有種類豐富的微生物,如可檢測到很多屬、種的乳酸菌[3-5]。
乳酸菌的分類鑒定方法很多,總的來說可分為經典方法和分子生物學方法。經典方法包括形態特征、生理生化反應及血清學反應等,這些方法都是基于微生物表面受體的特異性,屬于表型分類法。由于乳酸菌大多屬于兼性厭氧菌,且其對營養要求具有復雜性;傳統食品是一個復雜的環境系統,其中可能會存在許多人們尚未發過的乳酸菌菌種;傳統的表型、生物化學方法鑒定微生物耗時耗力等原因,最終可能導致鑒定結果不穩定[6]。而分子生物學方法是在分子水平上對生物個體的核酸和蛋白質進行研究,根據獲得的基因型信息對生物個體進行分類,從分子和基因水平來認識乳酸菌的遺傳結構、組成和分類。目前,許多新的分子標記技術被用來進行乳酸菌的鑒定:如16S r DNA序列分析[7]和基于聚合酶鏈式反應(Polymerase Chain Reaction,PCR),即PCR技術的分子標記技術等,其中基于PCR技術的分子標記又可分為隨機擴增多態性 DNA (Randomly Amplified Polymorphic DNA)、擴增片段長度多態性(Amplified Fragment Length Polymorphism)、限制性片段長度多態性(Restriction Fragment Length Polymorphism)等[8-10]。其中16S r DNA是細菌系統分類學中最有用的和最常用的分子鐘,其進化具有良好的時鐘性質,在結構和功能上具有高度的保守性,素有“細菌化石”之稱。16S大小適中,約1.5 kb左右,方便用于PCR擴增和測序,故被細菌學家及分類學家所接受,成為系統發育關系分類的標準方法。16S rDNA結構由可變區和恒定區組成,不同細菌的恒定區序列基本保守,而可變區序列則因細菌而異。所以,可以根據其恒定區的序列設計引物將16S r DNA擴增出來,利用可變區序列的差異來對不同的。
,16S r D NA序列分析也是最常用的方法之一,對有些相近種需要采用其他分子標記技術做補充。
作者以分離自四川地區的鮮牦牛奶和采用傳統方法制作曲拉樣品中分離得到的乳酸菌為研究對象,首先運用16S r DNA序列分析將其初步鑒定,部分乳酸菌屬于植物乳桿菌群,由于植物乳桿菌(Lactobacillus plantarum)和戊糖乳酸菌(Lactobacillus pentosus)之間的16S r DNA序列同源性為99%以上,難以從16S r DNA的系統發育樹上區分,故繼續采用recA基因特異擴增和hsp60-RFLP相結合的技術,對乳酸菌分離株進行鑒定。
1 材料與方法
1.1 材料
菌株:由內蒙古農業大學乳品生物技術與工程教育部重點實驗室提供,是從四川牧民家庭采集的鮮牦牛奶和以傳統方法制作的曲拉中分離得到的9株乳酸菌,菌號分別為IMAU80248,IMAU80249,IMAU80250,IMAU80251,IMAU80252,IMAU80286,IMAU80297,IMAU80323,IMAU80325。標準菌Lactobacillus plantarumATCC 14917,Lactobacillus pentosusATCC 8041:購于美國標準菌種收藏所(ATCC)。
1.2 試劑與儀器
10 g/d L脫脂乳培養基、TPY液體培養:參照文獻[11]制備;Taq DNA聚合酶、限制性內切酶等:購自大連寶生物工程有限公司;5810R型Eppendorf冷凍離心機、ND-1000型微量紫外分光光度計、PTC-200型梯度基因擴增儀:均購自基因有限公司。
1.3 所用引物
PCR擴增乳酸菌的16S r RNA、recA和hsp60基因引物見表1,所有引物均由上海桑尼生物科技有限公司合成。
2 實驗方法
2.1 乳酸菌基因組總DNA提取及純度的檢測
將分離純化的真空冷凍干燥保存的9株乳酸菌接種于5 m L滅菌的10 g/d L脫脂乳培養基中,37℃、培養24~48 h,活化培養三代。經革蘭氏染色鏡檢確定為純培養物后,將分離株接種于3 m LTPY液體培養基中,置37℃培養24 h。離心收集菌體后按液氮凍融-CTAB法[8]提取基因組總DNA,并采用微量紫外分光光度計測定基因組DNA的濃度和純度。

表1 擴增乳酸菌16S r RNA、rec A和hsp60基因擴增引物Tab.1 Primers used for amplification of 16S r RNA,rec A and hsp60 gene in Lactic acid bacteria
2.2 乳酸菌16S rDNA擴增、測序及系統發育樹的構建
將提取的基因組DNA稀釋到100 ng/μL左右作為PCR擴增的模板,采用通用引物27f和1495r[7,9]擴增9株乳酸菌的16S r RNA 基因。PCR反應體系(50μL)的配制:10×PCR Buffer 5μL,上下游引物各1.5μL (10μmol/μL),模板 DNA 1.5μL(100 ng/μL),d NTP Mix 4μL,r Taq酶0.5 μL,超純水補充至50μL。擴增程序為:94℃5 min預變性;94℃1 min,58℃1 min,72℃ 2 min,30個循環;72℃延伸10 min。用1.0 g/d L的瓊脂糖凝膠電泳檢測,并將16S r DNA擴增產物純化后送上海桑尼生物技術有限公司測序。
將 測 序 得 到 的 序 列 用 BLAST (http://www.ncbi.nlm.nih.gov/blast/)在 GenBank中搜索,與待測菌株同源性最高的已知分類地位的菌種初步確定待測菌株的種屬。然后從GenBank數據庫中下載已知的乳酸菌標準菌株的16S rDNA序列,使用軟件Mega4.0作16S rDNA序列系統發育樹。
2.3 rec A基因鑒定L.plantarum群的乳桿菌
RecA基因對于L.plantarum群的特異性較高,因此以相應的L.plantarum群的recA基因設計相應的引物[12],分別以planF、pentF為正引物,p REV為反引物。PCR反應體系(25μL)的配制:10×PCR Buffer 2.5μL,上下游引物各1.0μL(10 μmol/μL),模板 DNA 1μL (100 ng/μL),d NTP Mix 2μL,r Taq酶0.3μL,超純水17.2μL。擴增循環參數為:94℃3 min預變性;94℃變性30 s,56℃復性10 s,72℃延伸30 s,共30個循環;循環結束后72℃延伸5 min。
2.4 hsp60 基 因 擴 增 產 物-RFLP 標 記L.plantarum 群的乳桿菌
采 用 表1中 引 物 H729和 H730[13]擴 增L.plantarum群的乳桿菌hsp60基因。PCR反應體系(25μL)的配制:10×PCR Buffer 2.5μL,上下游引物各1.0μL(10μmol/μL),模板DNA 1μL(100 ng/μL),d NTP Mix 2μL,r Taq酶0.3μL,超純水17.2μL。擴增條件為:95℃3 min預變性;95℃變性30 s,40℃復性90 s,72℃延伸1 min,共40個循環。循環結束后72℃延伸10 min。采用NEBcutter 2分析已知L.plantarum和L.pentosus的hsp60序列的酶切位點,選用合適的限制性內切酶HhaI對L.plantarum群的乳桿菌hsp60基因擴增產物進行酶切即:酶切反應總體積為20μL,含2μL PCR反應產物,2 U內切酶和2μL10×反應緩沖液。37℃消化3 h,消化完畢在酶切產物中加0.5倍體積的10×Loading Buffer終止酶切,取6 μL酶切產物用5 g/d L聚丙烯酰胺膠(PAGE)凝膠電泳,銀染并將凝膠用掃描儀成像。
3 結果與分析
3.1 DNA提取結果
通過微量紫外分光光度計測定9株待測乳酸菌DNA的濃度和純度。A260/A280比值在1.8~2.0范圍內,1.0 g/d L瓊脂糖凝膠電泳結果見圖1。基因組DNA在23 kb處,DNA條帶清晰。從OD比值和DNA濃度可看出提取DNA質量較好,滿足后續試驗要求。
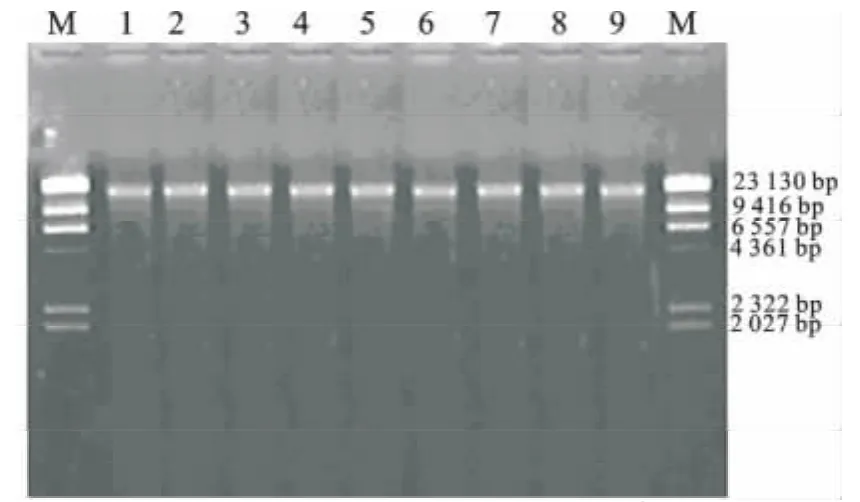
圖1 DNA瓊脂糖凝膠電泳圖Fig.1 Agarose gel electrophoresis of DNA of Lactic acid bacteria
3.2 乳酸菌16S r DNA擴增產物電泳結果
16S r RNA基因的擴增產物于1 g/d L瓊脂糖凝膠電泳后經溴化乙錠染色拍照,見圖2。從圖2中可以看出,PCR擴增比較成功,達到預期效果,即在1 500 bp附近均有清晰可見的明亮條帶,并且沒有非特異性擴增條帶,故9株待測菌株的16S r DNA擴增產物可以測序。

圖2 乳酸菌16S r DNA基因的PCR擴增產物電泳圖Fig.2 Agarose gel electrophoresis of PCR amplification of 16S rDNA in Lactic acid bacteria
3.3 測序及構建系統發育樹
通過對序列測定結果分析可知,9株待測乳酸菌的16S r DNA序列長度為1 500 bp左右。由待測9株乳酸菌的16S r DNA序列與8株參考菌株的16S r DNA序列用Mega4.0軟件繪制系統發育樹見圖3。從總體上可將這9株待測乳酸菌分為3大群:即L.plantarumATCC 14917、L.pentosusATCC 8041、IMAU 80296、IMAU 80297、IMAU 80323和IMAU 80325為第一群;IMAU 80252和L.helveticusATCC 15009為第二群;Leuconostoc mesenteroidesATCC 8293、IMAU 80248、IMAU 80249、IMAU 80250和IMAU 80251為第三群。通過BLAST序列比對分析和系統進化樹分析可知,第一類群菌株中IMAU 80296、IMAU 80297、IMAU 80323 和 IMAU 80325 與L.plantarumATCC 14917和L.pentosusATCC 8041相似性均達到99%以上,暫歸為植物乳桿菌群,這4株菌還需要采用其他方法進一步鑒定;第二類群菌株中IMAU 80252與Lactobacillus helveticusATCC 15009的同源性在99%以上,從而將其歸屬為瑞士乳桿菌;第三類群菌株IMAU 80248、IMAU 80249、IMAU 80250和IMAU 80251與菌株Leuconostoc mesenteroidesATCC 8293的同源性達99%以上,故判定此4株菌為腸膜明串珠菌。
3.4 rec A基因鑒定
利用recA基因進一步鑒定植物乳桿菌群中的4株菌,經PCR反應可得到不同相對分子質量的PCR產物,L.pentosus的 PCR 產物 為218 bp,L.plantarum的PCR產物為318 bp[12,14]。由圖4可得:菌株 IMAU 80325、IMAU 80297、IMAU 80296、IMAU 80323 與 標 準 菌 株L.plantarumATCC 14917條帶一致,因此可將待測4菌株鑒定為植物乳桿菌。
3.5 hsp60酶切產物-RFLP電泳圖譜及結果
為了更準確鑒定植物乳桿菌群中的4株菌,進一步采用hsp60/HhaI-RFLP法鑒定。由圖5可知:待測菌株IMAU 80296、IMAU 80297、IMAU 80323和IMAU 80325與標準菌株L.plantarumATCC 14917條帶一致,但與標準菌株L.pentosusATCC 8041存在差異。因此可將菌株IMAU 80296、IMAU 80297、IMAU 80323和IMAU 80325鑒定為植物乳桿菌,同時能夠看出經限制性核酸內切酶HhaI可以完成對植物乳桿菌和戊糖乳桿菌 的區分。

圖3 乳酸菌16S r DNA的系統發育樹Fig.3 Phylogenetic tree based on 16S rDNA sequence analyses
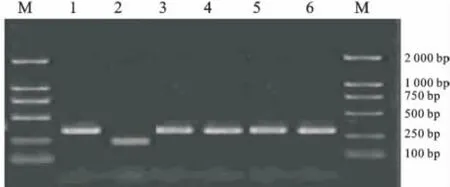
圖4 rec A基因的PCR擴增產物電泳圖Fig.4 Agarose gel electrophoresis of PCR amplification of rec A in Lactic acid bacteria
4 結語
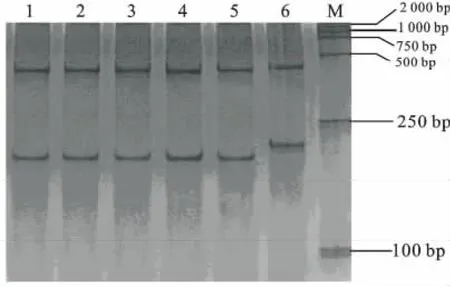
圖5 hsp60/Hha I-RFLP的聚丙烯酰胺凝膠電泳圖Fig.5 Polyacrylamide gel electrophoresis of Hsp60/Hha IRFLP
以乳酸菌16S r DNA序列測定為基礎,并構建系統發育樹,初步揭示9株乳酸菌的親緣關系。由于植物乳桿菌群(植物乳桿菌和戊糖乳桿菌)的16S r DNA序列同源性為99%,難以從系統發育樹上區分到種的水平。為了進一步鑒定植物乳桿菌群中的4株菌,采用recA-PCR和hsp60-RFLP相結合的技術,結果表明兩種方法鑒定結果相同,即菌株IMAU 80296、IMAU 80297、IMAU 80323 和IMAU 80325全部為植物乳桿菌。綜上所述,作者通過16S r DNA序列測定,并結合recA-PCR和hsp60-RFLP技術,成功將9株乳酸菌準確鑒定到種的水平,為其他部分乳酸菌的分子鑒定提供了簡便快速的方法。
(References):
[1]凌代文,東秀珠.乳酸菌的分類鑒定及實驗基礎 [M].北京:中國輕工業出版社,1999:1-5.
[2]金世琳.乳酸菌的科學與技術[J].中國乳品工業,1998,26(2):14-17.
JIN Shi-lin.Science and technology of lactic acid bacteria[J].China Dairy Industry,1998,26(2):14-17.(in Chinese)
[3]ZHANG H P,Xu J,WANG J G,et al.A survey on chemical and microbiological composition of kurut,naturally fermented yak milk from Qinghai in China[J].Food Control,2008,19(6):578-586.
[4]王煒宏.蒙古國傳統發酵乳制品中乳酸菌的分離鑒定 [D].呼和浩特:內蒙古農業大學,2010.
[5]DUAN Y H,Tan Z F,Wang Y P,et al.Identification and characterization of lactic acid bacteria isolated from Tibetan Qula cheese[J].Journal of General and Applied Microbiology,2008,54:51-60.
[6]段宇珩,談重芳,王雁萍,等.乳酸菌鑒定方法在食品工業中的應用及研究 [J].食品工業科技,2007,28(2):242-244.
DUAN Yu-xian,TAN Chong-fang,WANG Yan-ping,et al.Research on the identification of lactic acid bacteria in the food industry[J].Science and Technology of Food Industry,2007,28(2):242-244.(in Chinese)
[7]王煒宏,杜曉華,張家超,等.內蒙古鄂爾多斯地區酸粥發酵液中乳酸菌的分離鑒定 [J].食品與生物技術學報,2010,29(2):265-270.
WANG Wei-hong,DU Xiao-hua,ZHANG Jia-chao,et al.Isolation and identification of LAB from naturally fermented congee in Ordos area of Inner Mongolia[J].Journal of Food Science and Biotechnology,2010,29(2):265-270.
[8]WANG JG,CHEN X,LIU WJ,et al.Identification of Lactobacillus from koumiss by conventional and molecular methods[J].European Food Research and Technology,2008,227(5):1555-1561.
[9]于潔,孫志宏,張家超,等.16S r DNA-RFLP技術鑒定西藏地區乳制品中的乳桿菌[J].食品與生物技術學報,2009,28(6):804-810.
YU Jie,SUN Zhi-hong,ZHANG Jia-chao,et al.Identification of lactobacillus isolated from home-made fermented milk in Tibet by 16S rDNA-RFLP[J].Journal of Food Science and Biotechnology,2009,28(6):804-810.(in Chinese)
[10]劇檸,張家超,孫志宏,等.制作云南乳扇用酸乳清中乳桿菌的多樣性分析 [J].食品與生物技術學報,2010,29(5):735-741.
JU Ning,ZHANG Jia-chao,SUN Zhi-hong,et al.The Polymorphism ofLactobacillifrom acid whey for dairy fan in Yunnan Area[J].Journal of Food Science and Biotechnology,2010,29(5):735-741.
[11]小崎道雄,內村泰,岡田早苗,等.乳酸菌実験マニュアル[M].東京:朝倉書店,1992:29-72.
[12]Torriani S,Felis G E,Dellaglio F.Differentiation ofLactobacillus plantarum,L.pentosusandL.paraplantarumbyrecA gene sequence analysis and multiplex PCR assay withrecA gene-derived primers[J].American Society for Microbiology,2001,67(8):3450-3454.
[13]Goh S H,Facklam R R,Chang M,et al.Identification ofEnterococcusspecies and phenotypically similarLactococcusandVagococcusspecies by reverse checkerboard hybridization to chaperonin 60 gene sequences[J].Journal of Clinical Microbiology,2000,38:3953-3959.
[14]劉長建,權春善,范圣第,等.16S rDNA和rec A-gene對乳酸II32的鑒定 [J].大連民族學院學報,2007,36:50-52.
LIU Chang-jian,QUAN Chun-shan,FAN Sheng-di,et al.Identification of lactic acid bacteria II32 by sequence analysis of 16S r DNA and rec A-gene[J].Journal of Dalian Nationalities University,2007,36:50-52.(in Chinese)
Molecular Identification of Nine Strains Lactic Acid Bacteria Isolated from Yak Milk and Triton in Sichuan
BAO Qiu-hua,LU Qiang,YU Jie,WANG Hong-mei,ZHANG He-ping,SUN Tian-song*
(Key Laboratory of Dairy Biotechnology and Engineering Ministry of Education,Inner Mongolia Agricultural University,Hohhot 010018,China)
In this study,To accurately identify lactic acid bacteria(LAB)that were isolated from fresh yak milk and Qula in Sichuan region of China,nine LAB strains were classified and identified to species level by using 16S r DNA sequencing analysis,multiplex PCR assay of rec A gene,hsp60-PCR-RFLP analysis etc.The results showed that nine LAB strains were initially characterized as Lactobacillus plantarum group (4 strains),Leuconostoc mesenteroides(4 strains)andLactobacillus helveticus(1 strain)by 16S r DNA sequencing analysis.However,16S r DNA sequencing analysis could not distinguish betweenL.plantarumandL.pentosus.For further identification of strains inL.plantarumgroup,multiplex PCR assay ofrecA gene andhsp60-RFLP analysis were conducted.The results revealed that multiplex PCR assay ofrecA gene andhsp60-RFLP could identifyL.plantarumgroup to species level and 4 LAB strains inL.plantarumgroup were identified asL.plantarum.
lactic acid bacteria,identification,16S r DNA,RecA gene,Hsp60,RFLP
*通信作者:孫天松(1967-),女,內蒙古呼和浩特人,工學博士,教授,博士研究生導師,主要從事乳品微生物方面的研究。Email:sts9940@sina.com
Q 789
A
1673-1689(2012)04-0396-06
2011-03-23
國家自然科學基金地區重點項目(30860219);內蒙古自治區自然科學基金重點項目(2010Zd15)。
包秋華(1973-),女,內蒙古赤峰人,乳品生物技術與工程博士研究生,助理研究員,E-mail:nmgbqh@126.com

