RNA干擾載體構建方法的研究進展
粟 挺 ,劉愛玲 ,陳信波
(1.湖南農業大學生物科學技術學院,湖南 長沙 210128;2.湖南農業大學作物基因工程重點實驗室,湖南 長沙 410128)
隨著模式植物擬南芥和水稻全基因組測序工作的完成,植物基因組學的工作重心已由結構基因組學轉向功能基因組學,因此迫切需要一種高效、高通量分析方法從生物個體的整體水平闡明基因功能。RNA干擾是通過外源或內源性的雙鏈RNA在細胞內誘導同源序列的基因表達沉默的現象,其表現的性狀類似于基因功能缺失產生的表型[1-2]。RNA干擾現象自20世紀90年代中期被發現以來,就被廣泛應用于基因功能的分析驗證,成為后功能基因組學研究的熱點;在農作物選育方面也被廣泛應用于農作物的品種改良和農作物的抗病毒[3]研究。進行RNA干擾研究需構建可轉錄為雙鏈RNA的表達載體,然后獲得基因沉默的轉基因植株,進而分析目的基因的功能,這種方法的優點是可以獲得遺傳上穩定的突變體。強啟動子下游插入反向重復片段的表達載體是誘導RNA干擾的最基本結構,最有效的雙鏈RNA構件是能轉錄的發夾RNA(hairpin RNA,hpRNA)[4]。研究者一般從方法是否簡便、快捷、成功率高來評價植物RNA干擾載體構建方法的優劣。筆者對目前廣泛使用的構建RNA干擾表達載體的4種方法進行了歸納和總結。
1 傳統的“酶切—連接”方法構建RNA干擾表達載體
“酶切—連接”方法是構建RNA干擾載體最常用的方法,被廣泛應用于各種生物之中[5-8]。其構建過程是:通過PCR方法得到目的基因的片段(添加了合適的酶切位點),使之正反相連形成順式片段和反式片段,然后在順反式片段之間插入一段中間間隔序列以增強基因沉默效果[9-10],最后將表達載體和RNA干擾片段相連接,形成可用于遺傳轉化的RNA干擾表達載體。
傳統的“酶切—連接”方法對于在載體上添加1到2個基因比較容易,對于需添加多個基因的表達載體是比較困難的。除此之外,這種方法的試驗周期比較長,至少需要3個“酶切—連接”的循環才能完成載體構建,耗時耗力;構建成功率不太高,在添加最后一個基因片段時,無論是順式片段還是反式片段連接到表達載體上都比較困難;對原始載體和目的片段的限制條件比較多,原始載體至少有4個可用酶切位點,這就增加了原始載體選擇的難度。傳統的“酶切—連接”方法構建RNA干擾載體是最原始的構建方法,雖然成功地構建了多個植物RNA干擾表達載體,但其過程相對比較繁瑣,受到限制因素也比較多,不適合做高通量的研究。
2 “零背景篩選”技術構建RNA干擾表達載體
“零背景篩選”技術是Chen等[11]開發出來的一種高通量和高效率的構建RNA干擾表達載體的方法。該方法只需簡單的兩步PCR反應(圖1)就能形成干擾載體所需的“發夾”結構,免去了傳統方法中一系列的“酶切—連接”過程;并且此方法篩選陽性克隆的方法簡單,且出現“假陽性”的概率非常小,幾乎為零,所以叫做“零背景篩選”。通過中間載體中的致死基因ccdB來篩選陽性克隆,與目的基因連接成功的載體能存活,反之不能存活。此方法只需兩步簡單的PCR反應和一次“酶切—連接”過程就能完成RNA干擾表達載體的構建。其具體步驟如下。
2.1 兩步PCR反應
第一步,PCR過程中使用的引物P2和P4是在正常引物前加上一個含有特殊序列的接頭,而引物P2前面的接頭和P4前面接頭的序列是反向互補的。第二步,PCR過程所用的DNA聚合酶是一種特殊的酶,它可以在PCR產物后面加上A尾巴,如圖1。
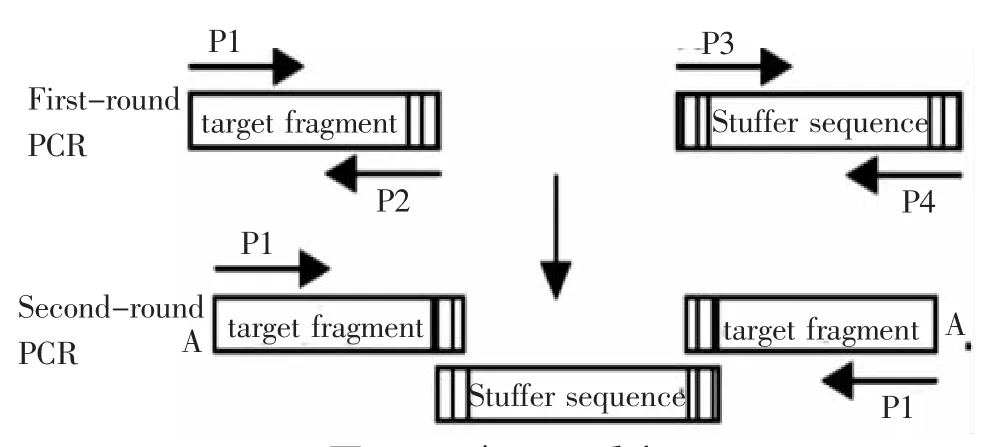
圖1 兩步PCR反應
2.2 一次“酶切—連接”過程
表達載體PCXUN和PCR產物的“酶切—連接”(圖2)。表達載體PCXUN經過XcmI限制性內切酶酶切后,會形成一個T尾巴,正好與PCR產物的A尾巴形成TA克隆。由于原始表達載體PCXUN中的致死基因ccdB被酶切掉,重組干擾載體不含致死基因ccdB,因此陽性克隆能存活。
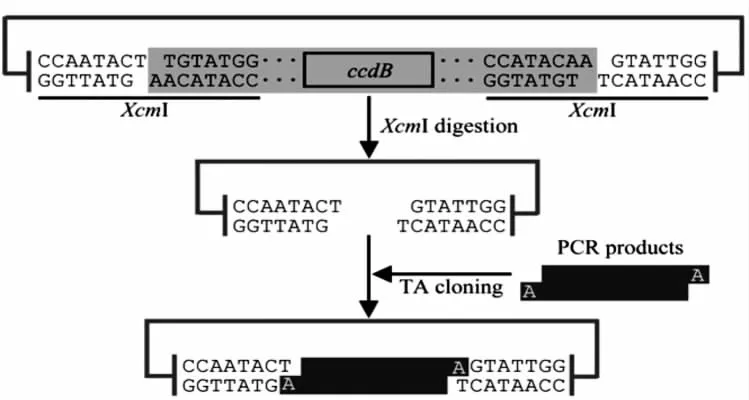
圖2 “酶切-連接”過程
“零背景篩選”技術通過經典的重組PCR方法[12]將兩個或多個基因片段通過一個基因片段的3′端與另一個基因片段的5′端的互補,利用重疊延伸的方法進行高效和快速的體外基因片段拼接。應用此方法構建目的基因的RNA干擾構件的關鍵是引物設計,在兩條引物前需分別加上一對序列反向互補的接頭,此外這個方法需要進行兩輪PCR反應,應使用保真性高的DNA聚合酶以保證擴增的真實性。在陽性重組子的篩選過程中由于原始載體致死基因ccdB的存在,導致陽性重組子的篩選效率極高。此方法只需兩步PCR反應就能得到RNA干擾構件[13],只需一步酶切—連接過程即可以將干擾構件連接于載體上,構建成RNA干擾表達載體,所以大大降低了對原載體上可用酶切位點的要求。使用這種技術,Chen等構建了一套由12個臨時載體和12個穩定載體組成的遺傳轉化載體用來研究植物的基因表達,并在水稻和擬南芥中驗證了這些載體的表達。這種方法較傳統構建方法更簡便、適用性更廣泛。
3 Gateway技術構建RNA干擾表達載體
Gateway技術是Invitrogen公司開發的一項基因克隆和表達的新技術,只需BP和LR兩個反應就可以完成RNA干擾表達載體的構建,不需要使用限制性內切酶和連接酶。
Gateway技術過程:BP反應(圖3)主要是將目的基因或PCR產物重組入供體載體(donor vector)。在獲得目的基因PCR片段時,需在PCR產物的5′端加入attB1位點,3′端加入attB2位點,供體載體兩端具有attP1和attP2位點,PCR產物和供體載體在BP反應酶催化下,通過同源重組形成新的位點attL1和attL2,生成帶有目的基因的入門載體。LR反應(圖4)主要是將目的基因從入門載體重組入目的載體,反應由LR反應酶混合物催化。入門載體基因兩端具有attL1和attL2位點,目的載體上含有attR1和attR2位點,在LR反應酶作用下發生定向重組,形成新的位點attB1和attB2,從而將目的基因轉移到目的表達載體中。篩選陽性克隆的機制是:入門載體質粒上帶有卡那霉素抗性基因,而目的載體質粒上帶有壯觀霉素抗性基因,重組目的載體質粒的選擇標記為壯觀霉素[14]。具體工作原理見圖4。
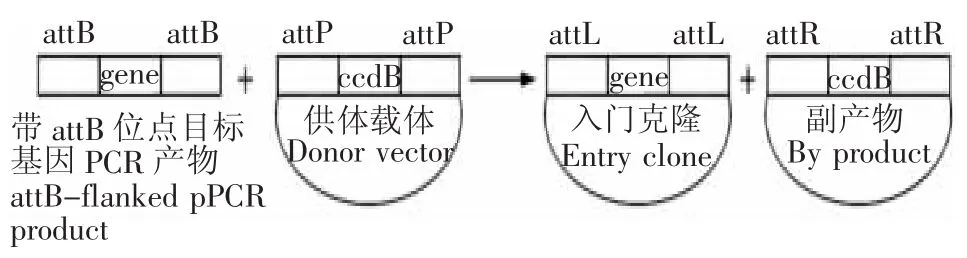
圖3 BP反應
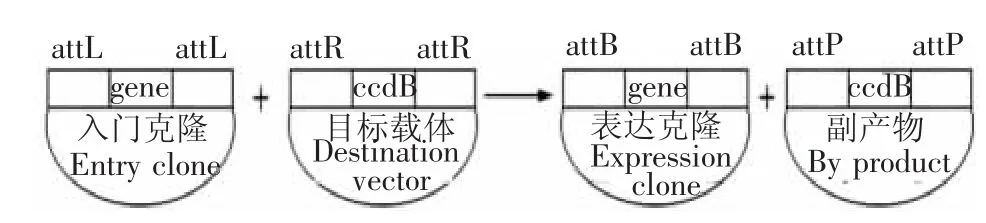
圖4 LR反應
Gateway技術是一項基因克隆和表達的新技術,與傳統的“酶切—連接”方法構建表達載體比較,該技術具有耗時短、操作簡單等特點,即只需通過高效的BP和LR反應就可實現把目的基因定向轉入表達載體中,克隆效率可達到95%[15]。Gateway技術應用于構建RNA干擾表達載體具有簡便、快捷、成功率高、對原始載體和目的片段的限制少等優勢,可避免構建RNA干擾載體時存在的困難和繁瑣的“酶切—連接”步驟。很多科研工作者已利用Gateway技術成功地構建了多種RNA干擾表達載體用于研究基因的功能,姜玲等[16]成功構建了番木瓜環斑病毒CP基因的RNA干擾表達載體;徐化學等[17]利用Gateway技術快速成功地構建了水稻OsDAD1基因的RNA干擾表達載體并用以研究基因的功能;晏立英等[18]構建了花生條紋病毒基因的RNA干擾表達載體。
4 以人工miRNA為基礎的方法構建RNA干擾表達載體
miRNA是一類內源單鏈非編碼小分子RNA,約為22 nt[19-20],廣泛存在真核生物中。其小分子RNA和靶mRNA通過特異的堿基配對結合,形成RISC沉默復合體,阻礙基因的正常表達。人工miRNA[21-22]技術指利用miRNA的表達特性,使用生物體內源的miRNA前體[23-24]作為人工miRNA的表達框架,產生出小分子RNA介導的靶基因沉默。因此,在二級結構不變的前提下,可以通過改變生物體內源miRNA前體的一些核苷酸,生成針對特異目的基因沉默的人工miRNA,目前被廣泛使用的miRNA前體骨架有 ath-miR159a,ath-miR164b,ath-miR169d,ath-miR172a,ath-miR319a和 osa-miR528。
Weigel等[25]建立了一個人工miRNA設計平臺,在 WMD3(Web MicroRNA Designer,http://wmd3.weigelworld.org/cgi-bin/webapp.cgi)系統中,可以對100多種植物設計人工miRNA。使用WMD3的“Designer”工具,選擇所要干涉的植物的基因組數據庫,輸入目的基因序列,在線提交,該系統通過和植物基因組數據庫比對,防止“脫靶”,并根據人工miRNA的相關參數(如Tm值等)列出候選的人工miRNA。Weigel等提供了基于擬南芥miR319a、水稻miR528、萊氏衣藻的pChlamiRNA2/3載體作為構建人工miRNA的模板,用PCR方法替換載體上miRNA片段,構建成人工miRNA表達載體。
人工miRNA技術是一項新興的生物技術,具有高效、精確、可控等優點,是替代RNA干擾的有效工具。幾乎所有可以利用RNA干擾的地方都可以用人工miRNA替代。與RNA干擾技術相比,人工miRNA具有獨特的優勢。人工miRNA可以同時干涉多個序列具有同源性的基因,因此可以用來研究多拷貝的基因或功能存在互補效應的基因;利用人工miRNA可以建立基因沉默的突變體庫,進行基因組功能研究。
5 結語
RNA干擾技術作為一種基因工程策略,被廣泛應用于農作物的遺傳改良、抗病毒和抗寄生線蟲[26]等方面的研究。構建RNA干擾表達載體是最常用和最基本的試驗步驟。隨著分子生物學技術的不斷發展,植物RNA干擾表達載體的構建方法也會不斷推陳出新,變得越來越簡便、有效。“零背景篩選”技術突破了傳統的限制性酶切法進行基因連接的概念,將重組PCR技術應用于植物RNA干擾表達載體的構建,是近兩年發展起來的一項新技術,被證明是可行的;Gateway技術利用同源重組的原理來構建RNA干擾表達載體,也是一種簡便、快捷的方法,適合高通量的研究;人工miRNA的方法具有干擾成功率高、并且允許堿基錯配、允許同時表達幾個人工miRNA分子,組織特異性的調節表達等優點[27],在植物發育調控、逆境應答及激素調節等方面應用價值更高[28]。隨著基因工程技術的發展,富有創新性的RNA干擾表達載體的構建方法必將為植物基因功能研究提供更有力的技術支撐。
[1]Napoli C,Lemieux C,Jorgensen R.Introduction of a chimeric chalcone synthase gene into petunia results in reversible co-suppression of homologous gene in trans[J].The Plant Cell,1990,2:279-289.
[2]Fire A,Xu S,Montgomery M K,et al.Potent and specific genetic interference by double-stranded RNA in Caenorhabditis elegans[J].Nature,1998,391(6669):806-811.
[3]Pinto Y M,Kok R A,Band com be D C.Resistance to rice yellow mottle virus(RYMV)in cultivated African rice varieties containing RYMV transgenes[J].Nat Biotechnology,1999,17(7):702-707.
[4]Smith N A,Singh S P,Wang M B.Total silencing by intron spliced hairpin RNAs[J].Nature,2000,407(6802):319-320.
[5]柴曉杰,王丕武,關淑艷,等.玉米淀粉分支酶基因反義表達載體的構建和功能分析[J].作物學報,2005,31(12):1654-1656.
[6]邢珍娟,王振營,何康來.轉Bt基因玉米幼苗殘體中CrylAb殺蟲蛋白田間降解動態[J].中國農業科學,2008,41(2):412-416.
[7]王 鐳,才 華,柏 錫,等.轉OsCDPK7基因水稻的培育與耐鹽性分析[J].遺傳,2008,30(8):1051-1052.
[8]呂 品,柴曉杰,王丕武,等.大豆胰蛋白酶抑制劑KSTI3基因的克隆及其植物表達載體的構建[J].吉林農業大學學報,2007,29(3):275-278.
[9]Kong Z S,Li M N.A novel nuclear-localized CCCH-type zinc finger protein,OsDOS is involved in delaying leaf sense cence in rice[J].Plant Physiology,2006,4:1376-1388.
[10]Daisuke M,Rikaiton,Koshimamoto.RNA silencing of single and multiple members in a gene family of rice[J].Plant Physiology,2005,138:1903-1913.
[11]Chen S B,Pattavipha S,Liu J L,et al.A Versatile Zero Background T-Vector System for Gene Cloning and Functional Genomics[J].Plant Physiology,2009,150:1111-1121.
[12]Horton R M,Pullen J K.Gene splicing by over extension[J].Methods Enzymol,1992,217:270-279.
[13]Xiao Y H,Yin M H,Hou L,et a1.Direct amplification of intron-containing hairpin RNA construct from genomic DNA[J].Biology Techniques,2006,41:548-552.
[14]Hartley J L,Temple G F,and Brasch M A.DNA cloning using in vitro site-specific recombination[J].Genome Research,2000,10(11):1788-1795.
[15]Wesley S V,Helliwell C A,Smith N A,et a1.Construct design for efficient effective and high throughput gene silencing in plants[J].Plant Journal,2001,27:581-590.
[16]姜玲,秦長平,伏 卉.Gateway系統快速構建番木瓜環斑病毒CP基因反向重復序列表達載體 [J].農業生物技術學報,2008,16(3):526-529.
[17]徐化學,熊建華,傅彬英.應用Gateway技術構建水稻OsDAD1基因的 RNA 干涉載體[J].分子植物育種,2007,5(1):133-136.
[18]晏立英,許澤永,陳坤榮,等.Gateway重組系統快速構建花生條紋病毒CP基因反向重復序列載體[J].農業生物技術學報,2007,15(2):356-357.
[19]王 磊,范云六.植物中微小RNA(microRNAs)研究進展[J].中國農業科技導報,2007,3(9):18-23.
[20]彭建斐,戴良英,何玉科,等.水稻微小RNA研究進展[J].湖南農業科學,2010,(8):4-6,10.
[21]Schwab R,Ossowski S,Riester M,et al.Highly specific gene silencing by artificial microRNAs in Arabidopsis[J].Plant Cell,2006,18:1121-1133.
[22]Alvarez J P,Pekker I,Goldshmidt A,et al.Endogenous and synthetic microRNAs stimulate simultaneous,efficient,and localized regulation of multiple targets in diverse species[J].Plant Cell,2006,18:1134-1151.
[23]Niu Q W,Lin S,Reyes J L,et al.Expression of artificial microRNAs in transgenic Arabidopsis thaliana confers virus resistance[J].Nature Biotechnology,2006,24:1420-1428.
[24]Parizotto E.A,Dunoyer P,Rahm N,Himber C,Voinnet O.In vivo investigation of the transcription,processing,endonucleolytic activity,and functional relevance of the spatial distribution of a plant miRNA[J].Genes and Development,2004,18:2237-2242.
[25]Weigel D,Warthmann N,Chen H,et al.Highly specific gene silencing by artificial miRNAs in rice[J].PLoS ONE,2008,3,e1829.
[26]Boutla A,Kalantidis K,Tavernarakis N.Induction of RNA interference in Caenorhabditis elegans by RNAs derived from plants exhibiting post-transcriptional gene silencing[J].Nucleic Acids Research,2002,30(7):1688-1694.
[27]Liu Q,Chen Y Q.Insights into the mechanism of plant development:Interactions of miRNAs pathway with phytohormone response[J].Biochemical and Biophysical,2009,384(1):1-5.
[28]Moxon S,Jing R,Szittya G,et a1.Deep sequencing of tomato short RNAs identifies microRNAs targeting genes involved in fruit ripening[J].Genome Research,2008,18:1602-1609.

