基于礦石納米材料的DNA轉化的機理初探及方法改進
譚海東,王磊,林金濤,趙宗保
中國科學院大連化學物理研究所生物技術部,大連 116023
微生物的 DNA轉化方法是基因工程中的重要內容,目前對微生物轉化主要有化學法和電轉化法[1]。但這些轉化方法,仍然存在許多缺點,比如感受態細胞準備時間長,處理過程容易導致細胞活性降低,處理后要有溫育等過程。2001年,Yoshida等發表了一系列關于使用溫石棉 (Chrysotile asbestos) 實現質粒對原核生物轉化的有趣方法[2-5],這種方法被命名為Yoshida效應。但是這種方法仍然有很多缺點:操作繁瑣、利用的介質對人體致癌等。對該法的可靠性有很多質疑,限制了它的應用[6],所以這種新穎的DNA轉化法很少被其他科學家引用。
最近,這種方法又進一步被Wilharm等改進[6]。他們使用的一種價格便宜、資源豐富、對人類健康友好的礦石納米材料海泡石 (Sepiolite) 作為介質,實現了對多種微生物的質粒轉化。作者在本方法中使用的參數與 Yoshida等報道的一致,并根據后者的報道,提出了借助海泡石進行DNA轉化的相同機制。問題是海泡石和溫石棉屬于不同的介質:前者沒有生物活性,后者有一定的生物活性;前者對人體友好,而后者可以致癌等。因此,我們認為這種新的轉化方法,可能會存在不同的DNA轉化機制。Wilharm 等雖然對方法進行了改進,但該工作是以非常短的通訊形式報道,很多參數未優化。我們認為對這種轉化機制的充分理解和對該試驗參數的優化,會促進這種方法的推廣和實現對特殊物種的DNA轉化。為此,我們進行了本研究。
1 材料與方法
1.1 材料
1.1.1 質粒和菌種
pET15b質粒有本組保存。E. coli DH5α菌株購自北京鼎國生物技術有限公司。短乳酸桿菌Lactobacillus brevis AS 1.579購自中國科學院微生物研究所菌種保藏中心。
1.1.2 試劑
蛋白酶K、250 bp DNA marker購自TaKaRa公司大連分公司。200~300 bp的RNA從短乳酸桿菌AS 1.579提取,在本組保存。海泡石購自德國Kremer Pigmente有限公司 (Kremer Pigmente GmbH & Co. KG,Hauptstr. 41?47DE 88317 Aichstetten,Germany),產品編號 58945。溶菌酶、鹽酸胍、十六烷基三甲基溴化胺 (CTAB)、苯酚和瓊脂糖購自北京鼎國生物技術有限公司。其他培養基及生化試劑牛肉膏、胰蛋白胨、酵母粉、氯仿和異戊醇等均為分析醇,購自大連博諾生物化學試劑廠。
1.2 方法
1.2.1 海泡石懸浮液和培養基的準備
海泡石懸浮液根據文獻準備4%儲存液[6]:海泡石121℃滅菌20 min;用去離子水配含200 mmol/L KCl,5 mmol/L HEPES緩沖液,用NaOH調pH到7.4,過膜除菌。最后配成5% (W/V) 海泡石儲存液。另外配10×海泡石緩沖液 (2 mol/L KCl,50 mmol/L HEPES,7.4) 備用。
LB培養基:在950 mL去離子水中加入胰蛋白胨10 g,酵母提取物5 g,NaCl 10 g,搖動容器直至溶解,用NaOH調pH至7.0,用去離子水定容至1 L。取200 mL,加入3 g瓊脂粉。在120℃滅菌20 min。待溫度降到50℃左右,加入終濃度為100 μg/mL的氨芐青霉素,準備40個抗性平板。
MRS培養基的配制:1 L蒸餾水中溶解下列組分:胰蛋白胨10 g,牛肉膏10 g,酵母提取物5 g,葡萄糖20 g,磷酸氫二鉀2 g,檸檬酸三銨2 g,乙酸鈉5 g,吐溫80 1 mL,硫酸鎂 200 mg,硫酸錳50 mg,用高壓鍋在121℃滅菌15 min,調pH到6.8。其中葡萄糖單獨滅菌。
1.2.2 海泡石吸附pET15b試驗及轉化E. coli DH5α的試驗
從于 37℃培養 20 h的新鮮平板中挑取一個E. coli DH5α單菌落,接種于10 mL LB液體培養基中,37℃下振蕩培養過夜。將該菌懸液以1∶100的比例接種于100 mL LB液體培養基中,37℃振蕩培養3 h至OD600=0.5左右。將培養液轉入離心管中,冰上放置10 min, 然后于4℃下4 000 r/min離心10 min。棄去上清,用預冷的海泡石緩沖液10 mL輕輕懸浮細胞,4℃下4 000 r/min離心10 min。最后用2 mL的海泡石緩沖液懸浮,置于冰上備用。
取40 μL pET15b溶液 (100 ng/μL),加入10 μL的5% (W/V) 海泡石儲存液,充分混合,取5 μL備用。剩余的溶液12 000 r/min離心30 s,用1 mL的海泡石緩沖液連續洗滌 2次。吸附結果用 1%的瓊脂糖凝膠電泳檢測。
取備用的細胞50 μL,加入5 μL質粒飽和的海泡石 (海泡石終濃度為0.1%),充分混合。取備用的細胞50 μL,加入1 μL洗滌后的海泡石 (海泡石終濃度為0.1%),充分混合。將2種懸浮液加到2個LB氨芐青霉素抗性平板 (100 μg/mL)。立即用玻璃板均勻涂抹30 s,最后放在37℃培養過夜。
1.2.3 利用海泡石對pET15b轉化E. coli DH5α的試驗參數優化
海泡石緩沖液對DNA轉化率的影響:取備用的細胞50 μL 2份,1份用LB洗滌細胞,另外1份仍然保留海泡石緩沖液。分別加入10 ng pET15b和1 μL 5% (W/V) 海泡石儲存液,充分混合。將2種懸浮液加到2個LB氨芐青霉素抗性平板 (100 μg/mL),立即用玻璃板均勻涂平板30 s,最后放在37℃培養過夜。
細胞濃度對DNA轉化率的影響:分別取備用的細胞500 μL、50 μL和5 μL,前者通過離心后用50 μL海泡石緩沖液懸浮,后者加入45 μL海泡石緩沖液懸浮,這樣 3個樣品的 OD600分別是 200、20和2。分別加入10 ng pET15b和1 μL 5% (W/V) 海泡石儲存液,充分混合。將3種懸浮液加到2個LB氨芐青霉素抗性平板 (100 μg/mL),立即用玻璃板均勻涂平板30 s,最后放在37℃培養過夜。
海泡石的濃度對DNA轉化率的影響:分別取備用的細胞50 μL 3份,分別配成海泡石終濃度為1%,0.1%和0.01%的混合液。分別加入10 ng pET15b充分混合。將3種懸浮液加到3個LB氨芐青霉素抗性平板 (100 μg/mL),立即用玻璃板均勻涂平板30 s,最后放在37℃培養過夜。
涂板次數和預干處理對DNA轉化率的影響:取備用的細胞50 μL 7份,分別加入10 ng pET15b和1 μL 5% (W/V) 海泡石儲存液,充分混合。將4份懸浮液加到4個LB氨芐青霉素抗性平板 (100 μg/mL)。立即用玻璃板分別均勻涂抹10次、20次、40次和100次,最后放在37℃培養過夜。另外3份細胞,1份立即用玻璃板均勻涂平板30 s,第2份10 min后用玻璃板均勻涂平板30 s,第3份20 min后用玻璃板均勻涂平板30 s,最后放在37℃培養過夜。
1.2.4 利用海泡石對pET15b直接轉化E. coli DH5α單菌落
取4℃存放1個月的E. coli DH5α單菌落,加入20 μL海泡石緩沖液,加入100 ng pET15b和1 μL 5% (W/V) 海泡石儲存液,充分混合。將細胞懸浮液加到LB氨芐青霉素抗性平板 (100 μg/mL)。立即用玻璃板均勻涂平板30 s,最后放在37℃培養過夜。
1.2.5 短乳酸桿菌AS1.579小片段RNA的提取
為了獲得短乳酸桿菌AS1.579小片段RNA,該加入100 μL 5 mol/L NaCl混勻,再加入80 μL的CTAB/NaCl混勻,65 ℃ 10 min。加入固體的鹽酸胍至終濃度6 mol/L[10],待細胞徹底裂解后,加入等體積酚/氯仿/異戊醇混合液 (25∶24∶1),混勻,12 000 r/min離心5 min,取上清,加0.6倍體積的異丙醇,混勻,室溫放置10 min。12 000 r/min離心10 min。沉淀用 75%的乙醇洗滌,晾干,最后用20 μL TE溶解,取3 μL電泳檢測。
1.2.6 小片段RNA對pET15b轉化E. coli DH5α的影響
取1 μL 5% (W/V) 海泡石儲存液,加入5 μL小片段的RNA提取液 (200 ng/μL),充分混合,讓海泡石飽和吸附RNA。加入10 ng pET15b和備用的細胞50 μL,充分混合。將細胞懸浮液加到LB氨芐青霉素抗性平板 (100 μg/mL)。立即用玻璃板均勻涂平板30 s,最后放在37℃培養過夜。并與不加小片段RNA的作對照。提取使用不加 RNAase的基因組提取方法,參考分子克隆實驗指南[7],并對此方法進行優化。簡述如下:取單克隆短乳酸桿菌到10 mL MRS培養基中,200 r/min、37℃培養過夜。將過夜培養物按 1∶50接入 100 mL MRS中,200 r/min、37℃培養至OD600=0.5。10 000 r/min離心30 s,收集沉淀,使用500 mmol/L的EDTA洗滌3次,最后用2 mL的500 mmol/L EDTA懸浮,200 W微波處理1 min[8-9]。10 000 r/min離心30 s收集沉淀,取50 mg菌體用400 μL TE重懸于1.5 mL離心管中,加入終濃度為10 mg/mL溶菌酶,10 mmol/L的DTT,37℃保溫1 h。加入50 μL蛋白酶K (10 mg/mL),混勻,65℃,1 h。
2 結果
2.1 海泡石吸附pET15b試驗
從圖1可以看出,用pET15b飽和后的海泡石,經過海泡石緩沖液充分洗滌后的海泡石幾乎檢測不到DNA。此結果說明海泡石對質粒的pET15b吸附能力很弱,幾乎看不到DNA吸附。用pET15b飽和的海泡石轉化細胞,長出的克隆數遠遠高于洗滌后的海泡石轉化數 (圖2,16-17)。這些結果說明,利用海泡石實現質粒轉化,有可能不是海泡石將DNA帶入宿主內的,而是由海泡石在宿主表面產生的瞬間小孔,導致DNA的隨機進入。
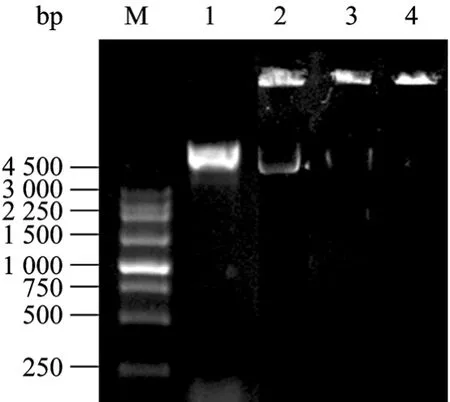
圖1 1%瓊脂糖凝膠電泳檢測海泡石吸附pET15b能力Fig. 1 Analysis of 1% agarose electrophoresis for the ability of sepiolite absorbing pET15b. 1: pET15b; 2: the sepiolite absorbing pET15b saturatedly; 3: the sepiolite saturated with DNA was washed once; 4: the sepiolite saturated with DNA was washed two times.
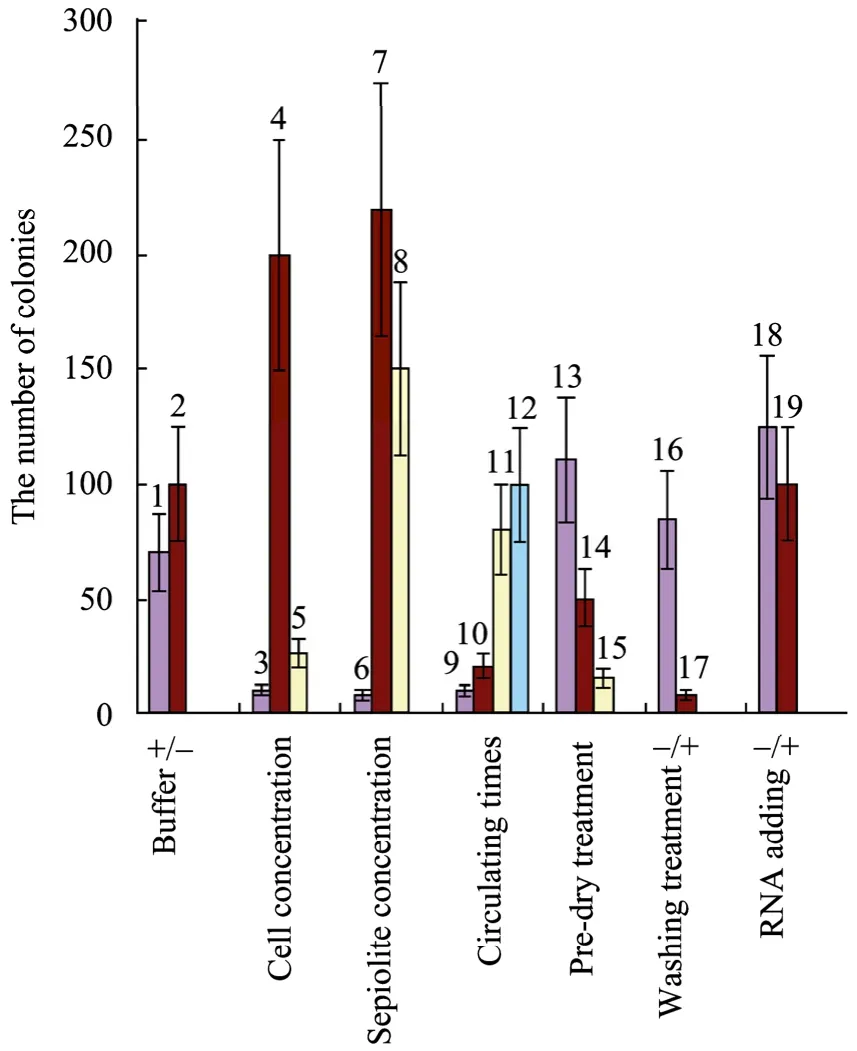
圖2 海泡石用于E. coli DH5αDNA轉化的參數優化Fig. 2 Optimization of DNA transformation for E. coli DH5α based on sepiolite. 1?2: the effect of sepiolite buffer for transformation. 3?5: the cell concentration is OD600=200, 20 and 2 in sample 3, 4 and 5 respectively. 6?8: the percent content of sepiolite is 1%, 0.1% and 0.01% in sample 6, 7 and 8 respectively; 9?12: the circulating times are 10, 20, 40 and 100 in sample 9, 10, 11 and 12 respectively. 13?15: the pre-dry treatment is 0 min, 10 min and 20 min in sample 13, 14 and 15 respectively; 16?17: washing effect for transformation; 18?19: the effect of RNA treatment for transformation.
2.2 利用海泡石對pET15b轉化E. coli DH5α的試驗參數優化
2.2.1 緩沖液
試驗結果表明,使用培養基轉化率略高于使用海泡石緩沖液 (圖2,1~2)。另外海泡石緩沖液配制和過膜除菌的操作繁瑣,利用培養基懸浮細胞,將使轉化變得更加直接。
2.2.2 細胞濃度
細胞濃度是決定轉化率高低的重要參數,我們研究表明當細胞濃度在OD600=20時轉化率最高 (圖2,3~5)。不同于電轉,細胞濃度越高轉化率越高。這可能與海泡石物理運動空間需要有關,細胞濃度太高,海泡石纖維不能充分運動,限制了質粒對細胞的有效轉化。
2.2.3 海泡石濃度
試驗結果表明使用海泡石的濃度比報道的高了近10倍,而轉化率有5倍以上增加 (圖2,6~8)。我們認為使用0.1%的海泡石濃度,而不是0.01%,轉化率會更高些。具體原因不很清楚,是否由于不同批次產品之間存在納米纖維材料數量的差異。
2.2.4 涂平板次數和狀態
菌液不經預干處理,直接涂抹平板,隨著涂抹次數的增加,轉化率逐漸增加 (圖2,9~12)。另外,隨著預干處理時間的延長,轉化率會逐漸降低 (圖2,13~15)。
2.3 利用海泡石對pET15b直接轉化E. coli DH5α單菌落
在 4℃儲存 1個月的單菌落,利用海泡石仍然可以實現質粒的轉化。取 4℃儲存 1個月的單菌落,利用海泡石直接轉化,轉化率低于 100/μg pET15b。但是,該操作大大節省了時間,不需要轉化前的再培養,沒有感受態制備或超低溫凍存和轉化后的溫育等過程。同時單菌落的直接轉化,由于菌量過少,通過鈣轉和電轉難以實現。當然,本操作不適用于對轉化率要求非常高的實驗,但可以在條件非常落后的實驗室中普及。該操作也可用于某些特殊的實驗,比如某些致病菌對抗性質粒的易感染性等。
2.4 短乳酸桿菌AS1.579小片段RNA的提取
為了得到理想化的RNA小片段,采取基因組提取方法得到降解的RNA片段。經過該試驗,可以從短乳酸桿菌得到理想的小片段RNA:濃度較高,大于200 ng/μL;分子量在300 bp左右 (圖3),根據文獻報道,非常適合用于海泡石上 DNA競爭結合試驗。同時,我們也嘗試了其他微生物的RNA提取,但是得到的RNA總是一條模糊的帶,分子量集中在500 bp和2 kb之間,不符合本試驗的需要。對這種結果的差異,無法從試驗過程中得知。
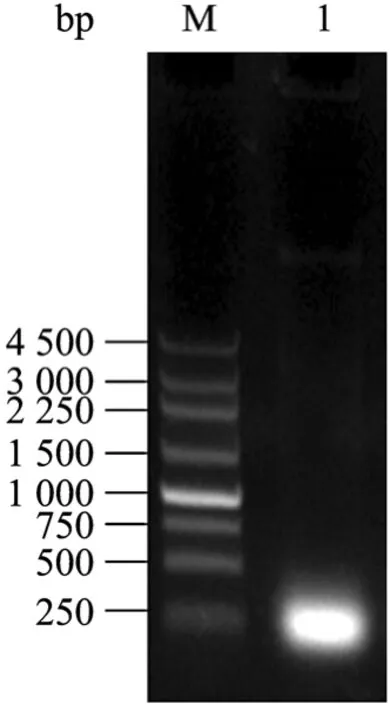
圖3 1%瓊脂糖凝膠電泳檢測短乳酸桿菌AS1.579小片段RNA的提取Fig. 3 Analysis of 1% agarose for small RNA extraction from Lactobacillus brevis AS1.579. M: marker; 1: small RNA.
2.5 小片段RNA對pET15b轉化E. coli DH5α的影響
經小片段RNA飽和的海泡石轉化E. coli DH5α,與對照組相比,幾乎沒有什么變化 (圖2,18~19)。同樣說明,利用海泡石實現質粒轉化,有可能不是海泡石帶 DNA進入宿主內,而是由海泡石在宿主表面產生的瞬間小孔,導致外源DNA的隨機進入。
3 討論
利用海泡石轉化有以下優點:可以實現質粒對細胞的轉化;這種介質對人體健康友好,無致癌物質;本身無生物活性,不會對宿主生長產生影響;操作過程中不需特殊的設備;不需要感受態的制備就可以得到較高的轉化率;另外,資源豐富、價格便宜,使這種轉化方法容易推廣。
但是,關于基于礦石海泡石納米材料的 DNA轉化機制仍然不很清楚。Yoshida等利用溫石棉進行DNA試驗,提出競爭機制,即海泡石溫石棉大量吸附DNA,借助涂平板的機械摩擦力,纖維絲插入宿主內。宿主內的RNA與海泡石溫石棉上的DNA競爭,從而實現外源基因對原核生物的轉化[6,11]。Wilharm等對此方法進行了改進,采用無生物活性,對人環境友好的海泡石進行了DNA轉化。但他們提出的轉化機制仍然與 Yoshida等報道的相同。而我們試驗表明海泡石吸附DNA的能力不是很強,洗滌后,轉化率大大降低。而用RNA飽和后的海泡石,轉化率幾乎沒有降低,這些結果說明,海泡石的作用是借助一種瞬間機械力,在細胞膜上擊出孔,借此外源 DNA隨即進入細胞。這種開孔現象是可逆的,移開海泡石,細胞膜會自動修復,將開孔重新封閉。細胞的這一特性,可使一些細胞外源DNA轉入細胞內,從而達到復制或表達的目的。另外,由于對DNA轉化機制理解的不同,導致操作方法有差異。Wilharm等根據他們提出的機制認為,在涂平板前要經過預干處理[6]。根據我們對該機制的理解,預干處理對提高DNA轉化率不利。不經預干過程,用玻璃棒立即涂平板,轉化率較高。而對照組,隨著預干處理時間的延長,轉化率會逐漸降低 (圖2,樣品13~15)。
在重新了解這種基于礦石納米材料微生物轉化機制基礎上,我們對該轉化進行了優化:使用對數生長初期的細胞為宿主,細胞濃度 OD600在 10~20之間,海泡石的濃度為0.1%,不經預干過程,不需要特定的緩沖液,持續涂平板時間在1 min以上,可以實現較高的轉化率,有時高于鈣轉。這些不同于最近的報道[6],但我們的轉化方法變得更直接,轉化時間變得更短。由于我們使用轉化率較低的pET15b作為實驗對象,說明這種方法可以用于多種質粒的轉化。使用其他質粒如pUC18等可以得到大于1×106/μg質粒的轉化率。這種轉化方法適用于雙鏈環狀DNA (質粒),線型DNA和其他微生物的轉化有待摸索。
當然這種轉化方法與電轉的轉化率還有很大差距,不同的人操作,會有較大的差異。但該方法無需感受態的制備、不需要貴重的儀器、甚至單菌落冷藏 1個月都可以直接實現轉化,所以這種方法的簡便性會得到很多研究者的青睞。海泡石用于微生物的DNA轉化屬于一種非常新的轉化方法,而且提升的空間很高。在我們的試驗中發現將DNA與E. coli DH5α在微型離心管內混合,槍頭的隨機抽動就可以實現DNA的轉化,該現象預示可以通過海泡石、DNA和微生物的共培養就可以實現DNA的轉化,這為研究特種微生物的DNA轉化提供很新的方法。另外,海泡石可以用于革蘭氏陽性菌的轉化[1]。但該方法用于真核生物的DNA轉化還沒有報道,根據現有的轉化機制推理,這種方法應該很快被用于真核生物的轉化。
REFERENCES
[1] Yoshida N, Sato M. Plasmid uptake by bacteria: a comparison of methods and efficiencies. Appl Microbiol Biotechnol, 2009, 83(5): 791?798.
[2] Yoshida N. Discovery and application of the Yoshida effect: nano-sized acicular materials enable penetration of bacterial cells by sliding friction force. Recent Pat Biotechnol, 2007, 1(3): 194?201.
[3] Yoshida N, Saeki Y. Chestnut bur-shaped aggregates of chrysotile particles enable inoculation of Escherichia coli cells with plasmid DNA. Appl Microbiol Biotechnol, 2004, 65(5): 566?575.
[4] Yoshida N, Kodama K, Nakata K, et al. Escherichia coli cells penetrated by chrysotile fibers are transformed to antibiotic resistance by incorporation of exogenous plasmid DNA. Appl Microbiol Biotechnol, 2002, 60(4): 461?468.
[5] Yoshida N, Nakajima-Kambe T, Matsuki K, et al. Novel plasmid transformation method mediated by chrysotile, sliding friction, and elastic body exposure. Anal Chem Insights, 2007, 2: 9?15.
[6] Wilharm G, Lepka D, Faber F, et al. A simple and rapid method of bacterial transformation. J Microbiol Methods, 2010, 80(2): 215?216.
[7] Sambrook J, Russel DW. Molecular Cloning. 3rd ed. Beijing: Science Press, 2002.薩姆布魯克丁, 拉塞爾 DW. 分子克隆實驗指南. 3版.北京: 科學出版社, 2002.
[8] Helander IM, Nurmiaho-Lassila EL, Ahvenainen R, et al. Chitosan disrupts the barrier properties of the outer membrane of gram-negative bacteria. Int J Food Microbiol, 2001, 71(2): 235?244.
[9] Goodwin DC, Lee SB. Microwave miniprep of total genomic DNA from fungi, plants, protists and animals for PCR. Biotechniques, 1993, 15(3): 438?444.
[10] Kamdar SJ, Evans R. Modifications of the guanidine hydrochloride procedure for the extraction of RNA: isolation from a variety of tissues and adherent/nonadherent cell types. Biotechniques, 1992, 12(5): 632?638.
[11] Yoshida N, Ide K. Plasmid DNA is released from nanosized acicular material surface by low molecular weight oligonucleotides: exogenous plasmid acquisition mechanism for penetration intermediates based on the Yoshida effect. Appl Microbiol Biotechnol, 2008, 80(5): 813?821.

